Un equipo de investigación ha logrado una mejora en la secuencia del genoma resuelto por haplotipos del cultivar de arroz japonica Nipponbare. Esta mejora revela la identificación y anotación de más de 3000 genes nuevos, lo que potencialmente ofrece avances significativos en la mejora de cultivos y estrategias de mejoramiento.
El cultivar de arroz japonica Nipponbare ha sido fundamental como referencia en la genómica del arroz desde su primera secuenciación hace más de dos décadas, lo que supone un avance significativo en la genómica de las plantas. A pesar de las continuas mejoras en la tecnología de secuenciación, el ensamblaje del genoma de Nipponbare aún contiene lagunas sin resolver, principalmente debido a secuencias repetitivas de ADN.
Los esfuerzos en curso y los avances tecnológicos han mejorado el ensamblaje del genoma en otras especies de arroz y se han extendido a la secuenciación de los telómeros. Sin embargo, lograr un ensamblaje completamente resuelto de haplotipos sigue siendo un problema no abordado en la investigación genómica del arroz, lo que presenta un área crítica para estudios futuros.
Un estudio publicado en Tropical Plants el 3 de abril de 2024, genera un genoma de arroz resuelto con haplotipos mejorado para una mejora integral de telómero a telómero (T2T).
En este estudio, se utilizaron lecturas PacBio HiFi y Hi-C para generar un ensamblaje contig con Hifiasm, lo que resultó en un ensamblaje en fases de haplotipo. Este proceso de ensamblaje produjo cóntigos distintos para nueve de los cromosomas. Por el contrario, el ensamblaje de los tres cromosomas restantes involucró dos cóntigos separados para cada uno.
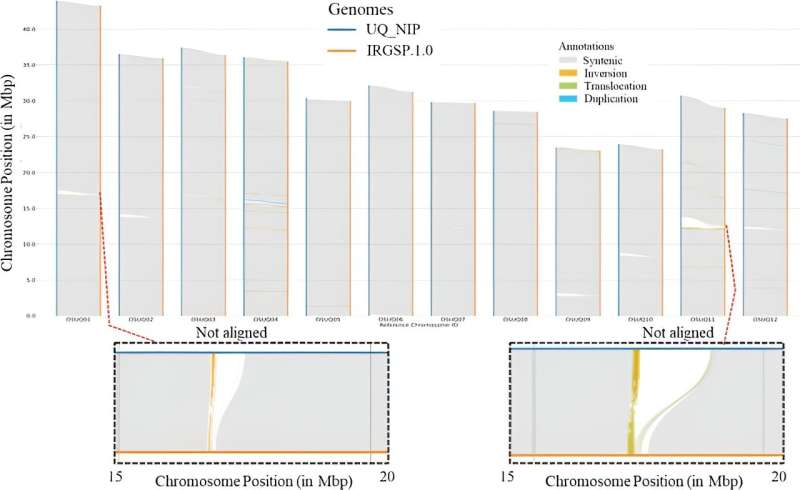
Luego, los contigs ensamblados se organizaron jerárquicamente en 12 pseudocromosomas utilizando la herramienta de andamiaje YaHS, culminando en un ensamblaje T2T que era más grande y más completo que la referencia anterior IRGSP-1.0. Este ensamblaje refinado reveló la presencia de 3050 genes nuevos, de los cuales más del 95 % están respaldados por evidencia de transcripción, lo que indica una mejora significativa en la anotación y la comprensión estructural del genoma.
Los hallazgos resaltan el inmenso potencial de las nuevas tecnologías de secuenciación para ampliar y refinar los datos genómicos, mejorando sustancialmente la información genética de los genomas establecidos. El genoma ampliado y más detallado, que cubre el 99,3% de los genes universales de copia única con un N50 de 30,7 Mb, proporciona un marco sólido para futuros estudios genéticos del arroz y programas de mejoramiento.
El análisis comparativo también iluminó variantes estructurales y regiones adicionales no alineadas, enriqueciendo la comprensión de la arquitectura y funcionalidad genómica.
Según el investigador principal del estudio, Robert J. Henry, "este genoma en fases será un recurso útil para la investigación del arroz". De cara al futuro, este equipo se centra en aplicar sus técnicas avanzadas de secuenciación y ensamblaje a otras variedades de arroz y especies estrechamente relacionadas.
En resumen, este trabajo no sólo subraya la rápida evolución de la tecnología genómica del arroz, sino que también enfatiza la necesidad crítica de avances continuos para mapear con precisión genomas complejos, facilitando así avances significativos en la genómica agrícola.
Más información: Muhammad Abdullah et al, Un genoma resuelto con haplotipos mejorado revela más genes de arroz, Plantas tropicales (2024). DOI:10.48130/tp-0024-0007
Proporcionado por la Academia China de Ciencias