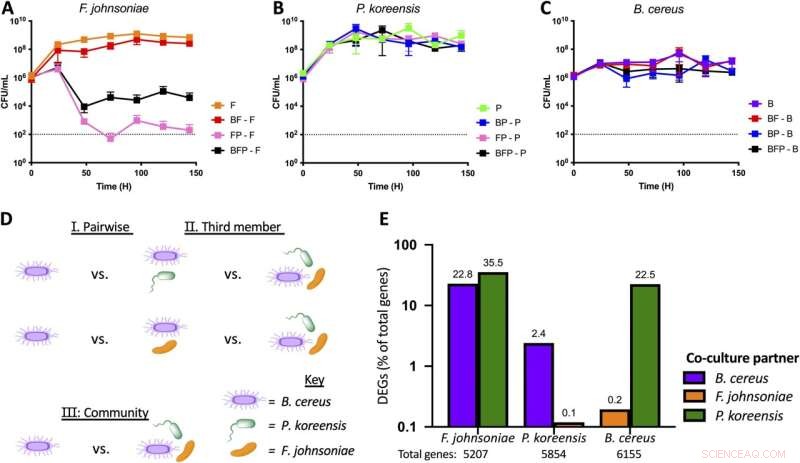
El miembro de THOR con defecto de aptitud de cocultivo exhibe la mayor respuesta transcripcional. Cada miembro de THOR se inoculó a 1 × 106 CFU/ml solo, en cocultivo por parejas y comunidad completa. Cada 24 h, los cultivos se cuantificaron por dilución en placas con antibióticos específicos de especie para determinar los niveles de UFC/mL de F. johnsoniae (A), P. koreensis (B) y B. cereus (C) en las diferentes condiciones de cocultivo durante 6 días. Los datos se muestran como duplicados biológicos y técnicos. Las líneas horizontales punteadas indican el límite de detección. (D) Esquema de comparaciones para descubrir cambios de expresión diferencial en respuesta a la cocultura por pares (categoría I), la adición del tercer miembro de THOR (categoría II) y la comunidad completa (categoría III). Se muestra como ejemplo una comparación de las condiciones de B. cereus. (E) Los cambios de expresión por pares globales (> 2 veces) se muestran como un porcentaje del número total de genes dentro de cada especie. Crédito:mBio (2022). DOI:10.1128/mbio.02486-21
Las bacterias, los organismos vivos más pequeños del mundo, forman comunidades donde cuerpos unificados de individuos viven juntos, contribuyen con una parte de la propiedad y comparten intereses comunes.
El suelo alrededor de las raíces de una planta contiene millones de organismos que interactúan constantemente:demasiados jugadores ocupados para estudiarlos a la vez, a pesar de la importancia de comprender cómo se mezclan los microbios.
En un estudio publicado en la revista mBio , investigadores de la Universidad de Wisconsin-Madison descubrieron que un modelo drásticamente reducido de una comunidad microbiana hace posible observar algunas de las interacciones complejas. Al hacerlo, descubrieron un actor clave en la comunicación microbiana:la presencia o ausencia de un compuesto antibiótico producido por uno de los miembros de la comunidad afectaba el comportamiento de los otros dos miembros.
Se sabe poco acerca de cómo los microbios individuales interactúan entre sí en las comunidades, pero ese conocimiento es increíblemente prometedor.
Por ejemplo, la bacteria Bacillus cereus puede proteger las plantas mediante la producción de un antibiótico que disuade al patógeno que causa la "depresión", una enfermedad que mata las plántulas y es costosa para los agricultores. Pero los agentes de biocontrol como B. cereus no siempre son efectivos. A veces, las plantas tratadas con B. cereus florecen, a veces no, y los investigadores están tratando de entender por qué.
"Las bacterias no viven aisladas", dice Amanda Hurley, autora principal del nuevo estudio; becario de política científica y tecnológica de la AAAS; y ex postdoctorado en el laboratorio del profesor de UW-Madison Jo Handelsman, director del Instituto de Descubrimiento de Wisconsin.
"Si pudiéramos descubrir cómo cambian las interacciones entre especies en presencia de múltiples especies, podemos comenzar a comprender las tendencias de comunicación de comunidades microbianas completas. Usando química o genética, podríamos interrumpir ciertas conversaciones y amplificar otras, lo que lleva a microbiomas que interactúan con sus ambientes de manera más positiva y predecible, ya sean humanos, cultivos o el suelo mismo".
Descifrar las interacciones entre microorganismos podría ayudar a diseñar un entorno más favorable para Bacillus cereus. Hurley y los coautores Marc Chevrette, ex postdoctorado en el laboratorio de Handelsman y actualmente profesor asistente en la Universidad de Florida, y Natalia Rosario-Melendez, estudiante de posgrado en el laboratorio de Handelsman, se dispusieron a decodificar y traducir las conversaciones químicas. El grupo creó un sistema modelo compuesto por tres especies:se aislaron Flavobacterium johnsoniae y Pseudomonas koreensis con B. cereus de raíces de soja cultivadas en el campo, a las que llamaron "Los autostopistas de la rizosfera" o THOR.
Las bacterias a menudo se comunican a través del lenguaje de la química. Manipular esa química usando genes y sustancias químicas podría cambiar la conversación y hacer que Bacillus cereus se sienta bienvenido en las raíces de las plantas.
Los investigadores construyeron perfiles de los organismos THOR utilizando su ARNm, moléculas producidas cuando se expresa un gen. En cada combinación de bacterias THOR, los investigadores buscaron diferencias en la expresión génica. Los organismos THOR respondieron entre sí de manera diferente en cada combinación, y cuando las tres especies estaban juntas, empezaron a suceder cosas nuevas que no ocurrían en ninguno de los pares o condiciones individuales.
En la comunidad THOR, la expresión génica estuvo dominada por interacciones con un miembro, P. koreensis. Los resultados estuvieron mediados por la presencia del antibiótico koreenceine, el martillo metafórico de THOR. Esta sola molécula parece afectar la expresión e interacción de miles de genes a través de redes comunitarias. Determinar cómo la koreenceína regula los genes de la comunidad será una vía fructífera para futuras investigaciones, según los investigadores.
El estudio valida la idea inicial de Handelsman de que vale la pena investigar las comunidades, porque la actividad dentro de la comunidad no es solo la suma de los miembros sino que refleja las propiedades de la comunidad.
"Tradicionalmente, las personas solo observan un solo organismo. Lo que hace que nuestro estudio sea diferente es que observamos a la comunidad", dice Chevrette. "Las comunidades son diferentes. Hay algo intrínsecamente único en una comunidad que la hace diferente a la suma de sus partes. Utilizar la simplicidad de los microbiomas modelo puede ayudarnos con el desafío de comprender los microbios en comunidades complejas y cómo pueden modificarse para mejorar la salud humana, ambiental y agrícola". THOR convierte microbiomas complejos en un modelo para mejorarlos