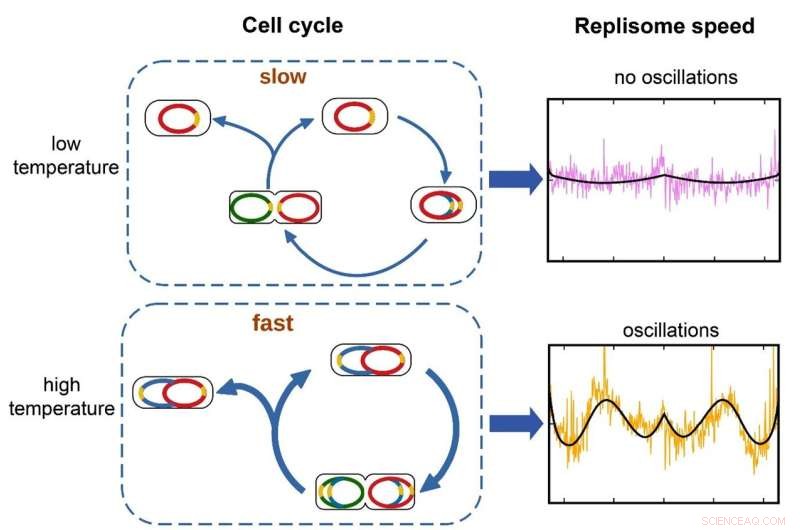
Cuando la tasa de crecimiento es rápida, como cuando las bacterias se cultivan a alta temperatura, aparecen oscilaciones en la velocidad del replisoma. Esto puede deberse a que múltiples copias del genoma son replicaciones al mismo tiempo. A tasas de crecimiento lentas, cuando solo se copia un genoma a la vez, las oscilaciones desaparecen. Crédito:Instituto de Ciencia y Tecnología de Okinawa
La división celular es fundamental para la vida, ya que permite que los organismos crezcan, reparen tejidos y se reproduzcan. Para que una célula se divida, primero se debe copiar todo el ADN dentro de la célula (el genoma), en un proceso llamado replicación del ADN. Pero la dinámica precisa de los replisomas, la maquinaria proteica que copia el ADN, ha sido difícil de determinar para los científicos.
Ahora, investigadores del Instituto de Ciencia y Tecnología de Okinawa (OIST) en Japón han desarrollado un nuevo modelo que puede determinar las variaciones en la velocidad a la que los replisomas copian los genomas bacterianos. El modelo, combinado con experimentos, muestra que ciertas secciones de ADN se copian más rápido que otras y revela un vínculo intrigante entre la velocidad de replicación y la tasa de error. La investigación fue publicada en eLife el 25 de julio de 2022.
"Las máquinas que copian el ADN son asombrosas, son muy rápidas y muy precisas", dijo Simone Pigolotti, profesora asociada de OIST que dirige la Unidad de Complejidad Biológica. "Comprender estas máquinas puede decirnos qué es importante para las células:qué errores son tolerables, qué errores no lo son, qué tan rápida debe ser la replicación".
El modelo se basa en medir qué tan abundantes son las diferentes ubicaciones de ADN dentro de una población de células bacterianas que se dividen constantemente. En las bacterias, para iniciar la replicación del ADN, dos replisomas se unen al ADN en un punto de origen establecido y se dirigen en direcciones opuestas a lo largo del bucle de ADN, copiando el ADN hasta que se encuentran en el otro lado. Esto significa que el ADN más cercano al punto de origen se copia primero, mientras que el ADN más cercano al punto de terminación se copia en último lugar.
"Si dejas que una población de bacterias crezca libremente, entonces, en un momento dado, la mayoría de las células estarán en el proceso de división celular. Debido a que la replicación del ADN siempre comienza desde el mismo lugar, esto significa que si luego secuencias todo el ADN , habrá una mayor abundancia de ADN que esté más cerca del punto de origen y una cantidad mucho menor de ADN que esté más cerca del punto final", explicó el profesor Pigolotti.
En el estudio, los investigadores de la Unidad de Ingeniería y Química de Ácidos Nucleicos del OIST cultivaron bacterias Escherichia coli (E. coli) a diferentes temperaturas. La Sección de Secuenciación luego secuenció el ADN de la bacteria.
Al analizar las características de la curva de distribución, los investigadores pudieron determinar la velocidad exacta de la maquinaria de proteínas. Descubrieron que a medida que aumentaba la temperatura, aumentaba la velocidad de replicación. Aún más interesante, los investigadores descubrieron que los replisomas variaban su velocidad en diferentes puntos a lo largo del genoma.
Una razón potencial de su velocidad fluctuante, especula el profesor Pigolotti, es que puede haber límites en los recursos necesarios para la replicación, como los nucleótidos, los componentes básicos del ADN.
En E. coli, cuando las condiciones son buenas, una sola célula bacteriana puede dividirse cada 25 minutos. Pero el proceso de replicación del ADN lleva más tiempo, alrededor de 40 minutos. Por lo tanto, para mantener altas tasas de crecimiento, se replican varias copias del genoma al mismo tiempo, lo que aumenta el número de replisomas en funcionamiento. La competencia por los nucleótidos podría hacer que los replisomas se ralentizaran.
Evidencia adicional respalda esta hipótesis. A bajas temperaturas y en cultivos pobres en nutrientes, cuando la tasa de crecimiento de la bacteria es baja y solo se copiaría un genoma a la vez, estas fluctuaciones en la velocidad de replicación desaparecen.
Curiosamente, los investigadores también encontraron que las oscilaciones observadas en la velocidad de replicación también coincidían con las oscilaciones en la tasa de mutación documentadas en otros estudios. Cuando superpusieron los dos patrones, encontraron que las áreas del genoma que se copiaron más rápido también tenían una tasa de mutación más alta.
"Esto parece intuitivo:si pensamos en una acción, como escribir en un teclado, cuanto más rápido escribamos, es más probable que cometamos un error", dijo el profesor Pigolotti. "Así que pensamos que cuando los replisomas van más rápido, su tasa de error es mayor".
Para el Prof. Pigolotti, el siguiente paso es determinar cómo cambia la velocidad de replicación en las cepas mutantes de E. coli, como aquellas a las que les faltan proteínas que ayudan en la replicación. También tiene curiosidad por ver si el patrón se mantiene en otras cepas de bacterias.
"Es una dirección de investigación realmente emocionante", dijo el profesor Pigolotti. "Y todo el trabajo se realizó en colaboración con otras unidades aquí. Es el tipo de colaboración interdisciplinaria que solo puede ocurrir en OIST". Dos proteínas ralentizan el tren de replicación del ADN en Drosophila