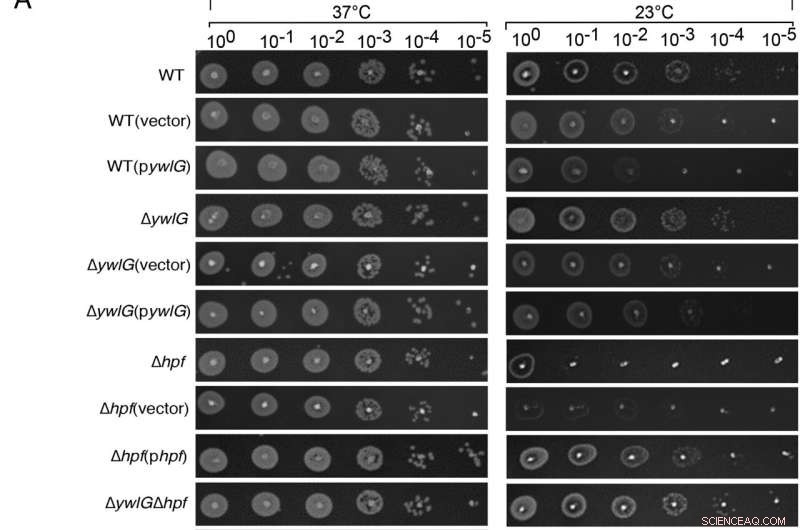
La inactivación de ywlG suprime la sensibilidad al frío de un mutante nulo Δhpf de una manera dependiente del medio. La complementación con ywlG y hpf codificados por plásmidos confirmó la supresión mediada por YwlG y la respuesta al frío dependiente de HPF. Se realizaron ensayos puntuales de dilución en serie en (A) placas de agar basadas en caldo de lisogenia (LB) o (B) basadas en caldo de soja tríptico (TSB). Células en crecimiento exponencial (OD600 =0,6) en TSB se ajustaron a OD600 =0,2 después de dos lavados en 1xPBS. Después de las diluciones en serie, se colocaron 2 µL de cada dilución en las placas de agar. Los resultados se registraron después de 24 h y 48 h de incubación a 37 °C y 23 °C, respectivamente. Las imágenes son representativas de tres experimentos independientes. Crédito:Actas de la Academia Nacional de Ciencias (2022). DOI:10.1073/pnas.2207257119
Los investigadores de Northwestern Medicine han descubierto nuevos mecanismos reguladores que promueven la supervivencia bacteriana, según los hallazgos publicados en Proceedings of the National Academy of Sciences. .
El estudio, dirigido por M.-N. Frances Yap, Ph.D., profesora asociada de Microbiología-Inmunología, también sienta las bases para identificar nuevos objetivos terapéuticos para tratar infecciones bacterianas.
La hibernación de los ribosomas es un mecanismo llevado a cabo por bacterias y otros organismos, incluidos los eucariotas, para garantizar la supervivencia a largo plazo. Los ribosomas 100S en hibernación en las bacterias consisten en dos complejos 70S unidos por una proteína del factor promotor de la hibernación (HPF).
Este complejo 100S tiene dos propósitos principales:prevenir la degradación de la maquinaria de biosíntesis de proteínas (ribosomas) y conservar energía en la célula al detener la traducción del ARNm.
En el estudio actual, el equipo de Yap utilizó espectrometría de masas para estudiar las interacciones proteicas en Staphylococcus aureus resistente a la meticilina, una bacteria comúnmente conocida por causar infecciones por estafilococos. A partir de estos análisis, descubrieron que HPF interactúa con un compañero de unión previamente no identificado ubicado fuera del ribosoma, una proteína llamada YwlG.
Además, al resolver la estructura atómica de YwlG, descubrieron que esta proteína participa en la actividad de la glutamato hidrogenasa, que es una vía metabólica esencial necesaria para la supervivencia de las bacterias.
"En condiciones normales, una fracción de la proteína YwlG se une al HPF. Al unirse, YwlG no puede estimular la actividad de la glutamato hidrogenasa y el HPF no puede unirse al ribosoma, por lo que es un 'secuestro mutuo' de las dos proteínas para garantizar la cantidad correcta de Se producen complejos 100S y glutamato deshidrogenasa celular", dijo Yap.
Los hallazgos apuntan tanto a HPF como a YwlG como factores que influyen en la colonización bacteriana y la gravedad de la infección. El objetivo final, según Yap, es identificar un compuesto que pueda interrumpir la formación de estos complejos y, a su vez, inhibir la supervivencia a largo plazo de las bacterias.
"Una ventaja clave es que la proteína similar a HPF o YwlG no existe en humanos, por lo que podría desarrollar un antibacteriano dirigido a HPF o YwlG que inhiba el crecimiento bacteriano pero no dañe a los huéspedes", dijo Yap. Los ribosomas en hibernación ayudan a las bacterias a sobrevivir