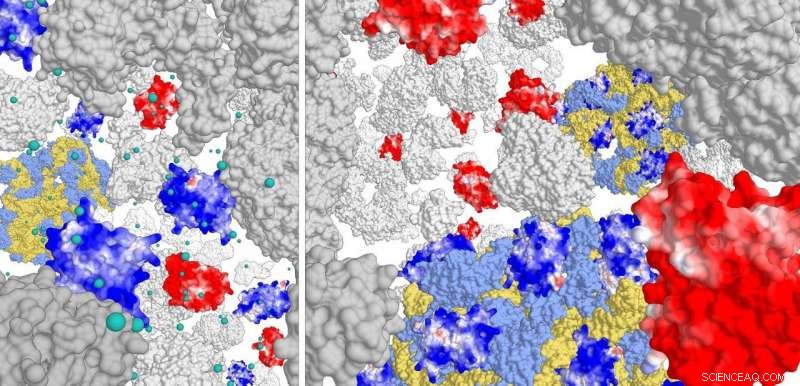
Panel derecho:interacción de proteínas cargadas positivamente (azul oscuro) con el complejo ribosómico (azul claro / amarillo). Las proteínas cargadas negativamente no interactúan. A alta fuerza iónica (panel izquierdo), las proteínas positivas apenas interactúan con el ribosoma. Crédito:Poolman lab, Universidad de Groningen
Las células están llenas de macromoléculas, que limita la difusión de proteínas, especialmente en células procariotas sin transporte activo en el citoplasma. Mientras investigaba la relación entre el hacinamiento, fuerza iónica y difusión de proteínas, Los bioquímicos de la Universidad de Groningen hicieron un descubrimiento fascinante:las proteínas cargadas positivamente se adhieren a la superficie de los complejos de ribosomas. Esto explica por qué la mayoría de las proteínas solubles en agua tienen una carga negativa general. El descubrimiento aparecerá pronto en la revista. eLife .
La velocidad del movimiento de las proteínas dentro de las células es importante:muchos procesos en las células biológicas dependen de las interacciones entre macromoléculas (proteínas y ácidos nucleicos) y, por tanto, de su capacidad para encontrarse entre sí. 'Pero el citoplasma celular es un lugar bullicioso y esto afectará la difusión de proteínas y ARN', comentarios Bert Poolman, profesor de bioquímica de la Universidad de Groningen.
Cargos
Su grupo estudió los efectos del hacinamiento en la difusión, y encontró una correlación entre el tamaño de la proteína y la velocidad de difusión. Pero para algunas proteínas no encontramos esta correlación, así que nos dispusimos a investigar por qué ”. El equipo utilizó tres procariotas diferentes con una fuerza iónica creciente:la bacteria Gram-negativa Escherichia coli, el Gram-positivo Lactococcus lactis y el extremófilo Haloferax volcanii, que vive en concentraciones muy elevadas de sal.
Para este estudio, los investigadores construyeron diferentes variantes de proteína fluorescente verde (GFP), con cargas superficiales que oscilan entre -30 y +25. Luego estudiaron el movimiento de estas variantes de GFP en los tres tipos de células. «Vimos que las proteínas cargadas positivamente se difundían muy lentamente. Se quedaron atrapados en la celda ', explica Poolman. Un análisis más detallado mostró que las proteínas positivas no se unían al ADN ni a la membrana celular, sino al complejo ribosómico.
Interesante
Un análisis bioinformático de los proteomas de microorganismos y células eucariotas mostró que, en la mayoría de los casos, aproximadamente el 70 por ciento de las proteínas están cargadas negativamente. 'Curiosamente, el 30 por ciento restante son proteínas de membrana o proteínas involucradas en el funcionamiento o plegamiento del ribosoma o ARNm ”.
Las proteínas de la membrana están protegidas por chaperonas durante la biogénesis, para que no se peguen a los ribosomas. Por lo tanto, no existen proteínas citoplasmáticas "libres" con una carga positiva lo suficientemente alta como para que se asienten en los ribosomas. La carga negativa del complejo ribosómico y la fuerza iónica ambiental del citoplasma parecen haber dado forma a la evolución de las cargas en el proteoma celular.
Inesperado
La idea nueva e inesperada de que la movilidad de las proteínas es una función de la carga de las proteínas puede explicar por qué es difícil expresar algunas proteínas en sistemas bacterianos con baja fuerza iónica. «Observamos que una fuerza iónica más alta reduce la adherencia de las proteínas cargadas positivamente. Eso podría ser una valiosa información para la construcción de plataformas de expresión de proteínas ”.
Una observación final en el artículo de eLife es que los genomas de varios endosimbiontes muestran una abundancia de proteínas cargadas positivamente. 'Este hallazgo realmente nos desconcierta', admite Poolman. «Es de esperar que todas estas proteínas se sientan atraídas por los ribosomas de los endosimbiontes. Hasta aquí, no tenemos una explicación de cómo estos organismos son capaces de lidiar con la difusión lenta y los ribosomas envueltos en proteínas positivas ”.