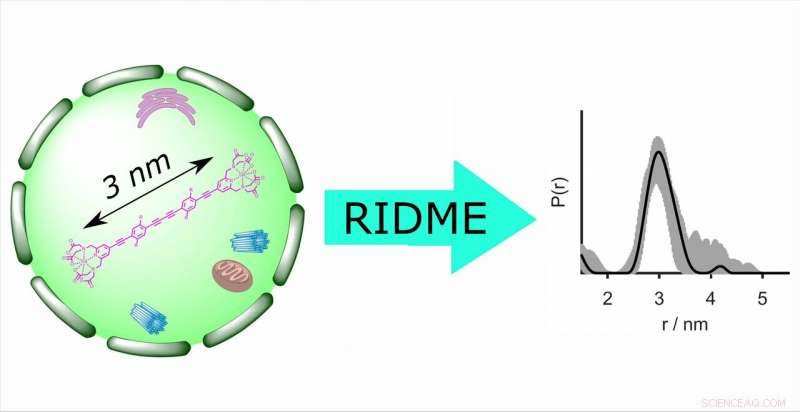
La determinación de la distancia dentro de la célula por EPR revela información estructural esencial sobre biomacromoléculas en condiciones nativas. Por primera vez, Se utilizó la técnica de EPR pulsado RIDME (mejora de la modulación dipolar inducida por relajación) para las mediciones de distancia dentro de las células. Proporciona una sensibilidad cinco veces mejorada en comparación con el método de resonancia de doble electrón-electrón utilizado anteriormente. Crédito:Grupo de investigación Profesor Malte Drescher, Universidad de Konstanz
En un documento conjunto, investigadores de la Universidad de Konstanz, La Universidad de Bielefeld y ETH Zurich demuestran por primera vez que la técnica de resonancia paramagnética electrónica (EPR) RIDME (mejora de la modulación dipolar inducida por relajación) se puede aplicar para determinar distancias entre marcadores de espín basados en gadolinio (III) en las células. La determinación de la distancia dentro de la celda por resonancia paramagnética electrónica (EPR) revela información estructural esencial sobre biomacromoléculas, incluyendo su conformación así como los procesos de plegado y desplegado.
Los métodos convencionales para la determinación de distancias dentro de la celda, como la resonancia de doble electrón-electrón (DEER o PELDOR), son principalmente mucho menos sensibles que RIDME. proporcionan profundidades de modulación hasta cinco veces más pequeñas, tienen ciertas limitaciones con respecto al ancho de banda de excitación y son técnicamente más exigentes. Como técnica de frecuencia única que utiliza giros de giro inducidos por relajación para determinar la distancia entre dos etiquetas de giro, es decir, entre dos electrones no apareados, RIDME supera todas estas desventajas.
Esta técnica permite a los investigadores trabajar con moléculas en condiciones nativas, como el profesor Malte Drescher y el autor principal, el Dr. Mykhailo Azarkh, ambos de la Universidad de Konstanz, enfatizar:"Comenzamos analizando la conformación de una proteína dentro de la célula. Con técnicas menos sensibles, nos vemos obligados a insertar y etiquetar mucha proteína para poder observarla, que no es en absoluto lo que sucede en la naturaleza. Idealmente, queremos trabajar con concentraciones que sean fisiológicamente relevantes. Dado que RIDME es mucho más sensible que DEER, nos permite hacer precisamente eso. Ahora estamos en condiciones de abordar cuestiones que de otro modo no podríamos abordar ".
El rendimiento de RIDME en la célula se evaluó en la banda Q utilizando reglas moleculares rígidas marcadas con Gd (III) -PyMTA y microarticuladas en ovocitos de Xenopus laevis (rana africana con garras). En otras palabras, los investigadores utilizaron un sistema modelo en el que ya se conocía la distancia precisa entre las etiquetas de giro, permitiéndoles verificar las medidas RIDME. El artículo resultante titulado "Mejora de la modulación dipolar inducida por relajación de Gd (III) -Gd (III) para la determinación de la distancia de resonancia paramagnética electrónica en la celda" se publicó en línea en el Revista de letras de química física el 13.03.2019.
La determinación de la distancia dentro de la celda RIDME se desarrolló y probó como parte del proyecto en curso financiado por ERC "SPICE - Espectroscopía en celdas", por lo que Malte Drescher, Profesor Heisenberg de espectroscopia de sistemas complejos en la Universidad de Konstanz, y su equipo de investigación fueron reconocidos con una ERC Consolidator Grant por valor de aproximadamente dos millones de euros en 2017. Su objetivo es desarrollar nuevos enfoques de espectroscopia que les permitan explorar estructuras biológicas más grandes y complejas a nivel molecular de la célula.
El siguiente paso en esta línea de investigación será identificar otras etiquetas de espín adecuadas y desarrollar RIDME para su aplicación en moléculas donde se desconoce la distancia entre las etiquetas de espín. Se prestará especial atención a las moléculas asociadas con enfermedades neurodegenerativas como el Alzheimer y el Parkinson.