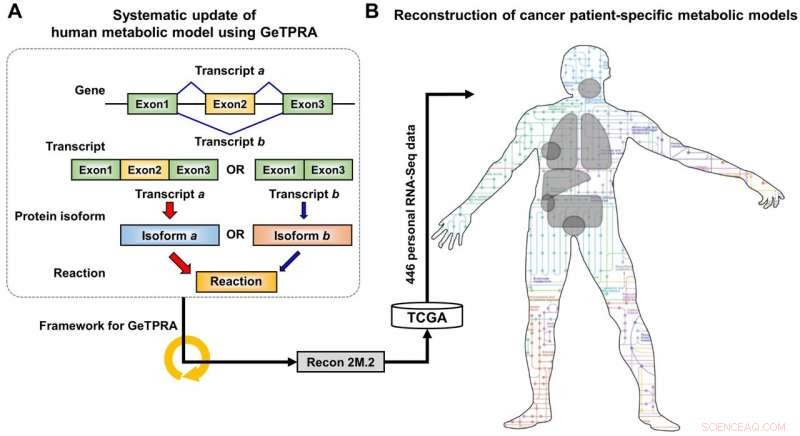
Esquema de desarrollo de Recon 2M.2 y su uso en la reconstrucción de modelos metabólicos personales a escala del genoma (GEM). (A) Al desarrollar Recon 2M.2, se consideró un concepto de empalme alternativo de genes humanos, e incorporado en el modelado a través del marco computacional para Asociaciones de Reacción de Proteína-Transcripción Genética (GeTPRA). (B) Tras el desarrollo de Recon 2M.2, Los GEM específicos de pacientes con cáncer podrían reconstruirse utilizando datos biológicos personales (datos de RNA-Seq). En este estudio, Los datos de RNA-Seq específicos del paciente se obtuvieron de The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Crédito:KAIST
Un equipo de investigación coreano de KAIST desarrolló un marco computacional que permite la reconstrucción de un modelo computacional integral del metabolismo humano, que permite una predicción precisa de las características metabólicas personales (o fenotipos).
Comprender los fenotipos metabólicos personales nos permite diseñar estrategias terapéuticas efectivas para diversas enfermedades crónicas e infecciosas. Un modelo computacional humano llamado modelo metabólico a escala del genoma (GEM) contiene información sobre miles de genes metabólicos y sus correspondientes reacciones y metabolitos. y ha jugado un papel importante en la predicción de fenotipos metabólicos. Aunque se han lanzado varias versiones de GEM humanos, tenían espacio para un mayor desarrollo, especialmente en cuanto a la incorporación de información biológica procedente de un mecanismo genético humano llamado "empalme alternativo". El empalme alternativo es un mecanismo genético que permite que un gen dé lugar a reacciones múltiples, y está fuertemente asociado con la patología.
Para abordar este problema, Jae Yong Ryu (estudiante de doctorado), Dr. Hyun Uk Kim (becario de investigación), y el distinguido profesor Sang Yup Lee, todos del Departamento de Ingeniería Química y Biomolecular de KAIST, desarrolló un marco computacional que genera sistemáticamente reacciones metabólicas, y los agrega al GEM humano. Se demostró que la GEM humana resultante predice con precisión los fenotipos metabólicos en diversas condiciones ambientales. Los resultados de la investigación se publicaron en línea en procedimientos de la Academia Nacional de Ciencias ( PNAS ) el 24 de octubre, 2017, bajo el título "Marco y recurso para más de 11, 000 asociaciones gen-transcripción-proteína-reacción en el metabolismo humano ".
El equipo de investigación primero actualizó el contenido biológico de una versión anterior del GEM humano. Los contenidos biológicos actualizados incluyen genes metabólicos y sus correspondientes metabolitos y reacciones. En particular, Las reacciones metabólicas catalizadas por isoformas de proteínas ya conocidas se incorporaron adicionalmente a la GEM humana; Las isoformas de proteínas son múltiples variantes de proteínas generadas a partir de genes individuales mediante el proceso de corte y empalme alternativo. Cada isoforma de proteína es a menudo responsable del funcionamiento de una reacción metabólica. Aunque múltiples isoformas de proteínas generadas a partir de un gen pueden desempeñar diferentes funciones al tener diferentes conjuntos de dominios de proteínas y / o localizaciones subcelulares, dicha información no se consideró adecuadamente en versiones anteriores de GEM humanos.
Tras la actualización inicial del GEM humano, llamado Recon 2M.1, Posteriormente, el equipo de investigación implementó un marco computacional que genera sistemáticamente información sobre las asociaciones de reacciones de proteínas, transcripciones y genes (GeTPRA) para identificar isoformas de proteínas que antes no se identificaban. Este marco fue desarrollado en este estudio. Como resultado de la implementación del marco para GeTPRA, más de 11, 000 GeTPRA se predijeron automáticamente, y validado a fondo. A continuación, se añadieron reacciones metabólicas adicionales a Recon 2M.1 basadas en el GeTPRA predicho para las isoformas de proteínas no caracterizadas previamente; Recon 2M.1 pasó a llamarse Recon 2M.2 a partir de esta actualización. Finalmente, Recon 2M.2 se integró con 446 conjuntos de datos biológicos personales (datos de RNA-Seq) para construir modelos de cáncer específicos para el paciente. Estos modelos de cáncer específicos del paciente se utilizaron para predecir las actividades del metabolismo del cáncer y los objetivos anticancerígenos.
Se espera que el desarrollo de una nueva versión de GEM humanos junto con el marco computacional para GeTPRA impulse los estudios en genética y medicina humanas fundamentales. Archivos de modelo de los GEM humanos Recon 2M.1 y 2M.2, una lista completa de la GeTPRA y el código fuente del marco computacional para predecir la GeTPRA están disponibles como parte de la publicación de este estudio.
El distinguido profesor Lee dijo:"Se espera que la GeTPRA predicha del marco computacional sirva como guía para futuros experimentos sobre genética y bioquímica humana, mientras que el Recon 2M.2 resultante se puede utilizar para predecir objetivos de fármacos para diversas enfermedades humanas ".