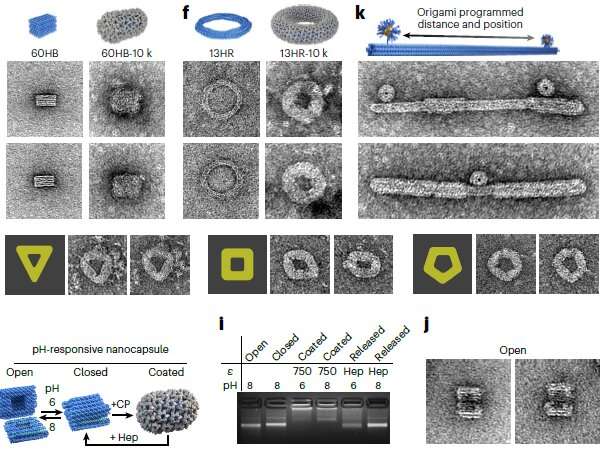
Los investigadores de la Universidad Griffith han desempeñado un papel clave en el uso de plantillas de "origami" de ADN para controlar la forma en que se ensamblan los virus.
El equipo global detrás de la investigación, titulada "Polimorfismo de la cápside del virus dirigido por origami de ADN", publicado en Nature Nanotechnology , desarrolló una forma de dirigir el ensamblaje de las cápsides virales (la cubierta proteica de los virus) en condiciones fisiológicas de una manera precisa y programable.
El Dr. Frank Sainsbury y la Dra. Donna McNeale del Instituto Griffith para el Descubrimiento de Fármacos formaron parte del equipo de investigación y dijeron que forzar a los virus a ensamblarse en ADN doblado en diferentes formas "como origami" era una pregunta que este proyecto respondía.
"Logramos controlar la forma, el tamaño y la topología de la proteína del virus mediante el uso de nanoestructuras de origami de ADN definidas por el usuario como plataformas de unión y ensamblaje, que quedaron incrustadas dentro de la cápside", dijo el Dr. Sainsbury.
"Los recubrimientos de proteínas del virus podrían proteger el origami de ADN encapsulado de la degradación.
"Esta actividad es más como envolver un regalo:las proteínas del virus se depositan encima de la forma diferente definida por la forma del origami del ADN.
"Y las diferentes proteínas virales son como diferentes papeles de regalo, lo que sería relevante para diferentes usos del origami de ADN recubierto".
Un control preciso sobre el tamaño y la forma de las proteínas del virus tendría ventajas en el desarrollo de nuevas vacunas y sistemas de administración.
"Pero las herramientas actuales para controlar el proceso de ensamblaje de manera programable eran difíciles de alcanzar", afirmó el Dr. McNeale.
"Nuestro enfoque tampoco se limita a un solo tipo de unidad de proteína de la cápside del virus y también se puede aplicar a estructuras de origami de ARN y ADN para allanar el camino para estrategias de focalización y protección de carga de próxima generación".
Actualmente, el Dr. Sainsbury y su equipo están trabajando para lograr una comprensión más profunda de cómo se autoensamblan los diferentes virus y cómo se pueden utilizar para encapsular diferentes cargas.
Esto les permitirá diseñar y modificar otras partículas similares a virus para una variedad de usos. Por ejemplo, descubrieron que un virus encontrado en ratones es capaz de transportar cargas de proteínas a través de entornos inhóspitos y hasta un compartimento subcelular específico en las células humanas.
"Con el enorme espacio de diseño existente entre los virus que podrían usarse como portadores, todavía hay mucho que aprender al estudiarlos. Continuaremos ampliando los límites de cómo se pueden ensamblar partículas similares a virus y qué se puede aprender al usarlos. como transportadores de medicamentos, vacunas y recipientes de reacción bioquímica", afirmó el Dr. Sainsbury.
La siguiente etapa de la investigación del equipo GRIDD utilizará este enfoque para observar por qué los virus no se ensamblan en diferentes formas por sí mismos.
Más información: Polimorfismo de la cápside del virus dirigido al origami de ADN, Nanotecnología natural (2023). DOI:10.1038/s41565-023-01443-x, https://www.nature.com/articles/s41565-023-01443-x
Información de la revista: Nanotecnología de la naturaleza
Proporcionado por la Universidad Griffith