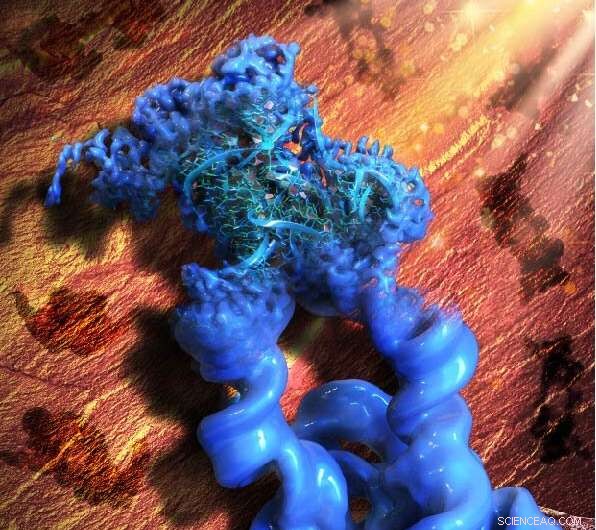
Esta ilustración está inspirada en la pintura rupestre paleolítica de la cueva de Lascaux, que significa el acrónimo de nuestro método, ROCK. En sentido figurado, los patrones del arte rupestre en el fondo (marrón) son las proyecciones 2D de la construcción dimérica diseñada del intrón del grupo I de Tetrahymena, mientras que el objeto principal en el frente (azul) es el mapa crio-EM 3D reconstruido del dímero, con un monómero en foco y refinado a la alta resolución que permitió a los colaboradores construir un modelo atómico del ARN. Crédito:Instituto Wyss de la Universidad de Harvard
Vivimos en un mundo hecho y dirigido por ARN, el hermano igualmente importante de la molécula genética ADN. De hecho, los biólogos evolutivos plantean la hipótesis de que el ARN existía y se autorreplicaba incluso antes de la aparición del ADN y las proteínas codificadas por él. Avance rápido a los humanos de hoy en día:la ciencia ha revelado que menos del 3% del genoma humano se transcribe en moléculas de ARN mensajero (ARNm) que a su vez se traducen en proteínas. Por el contrario, el 82% se transcribe en moléculas de ARN con otras funciones, muchas de las cuales siguen siendo enigmáticas.
Para comprender lo que hace una molécula de ARN individual, es necesario descifrar su estructura 3D al nivel de sus átomos constituyentes y enlaces moleculares. Los investigadores han estudiado rutinariamente moléculas de ADN y proteínas convirtiéndolas en cristales empaquetados regularmente que pueden examinarse con un haz de rayos X (cristalografía de rayos X) u ondas de radio (resonancia magnética nuclear). Sin embargo, estas técnicas no se pueden aplicar a moléculas de ARN con casi la misma eficacia porque su composición molecular y flexibilidad estructural les impiden formar cristales con facilidad.
Ahora, una colaboración de investigación dirigida por el miembro de la facultad de Wyss Core, Peng Yin, Ph.D. en el Instituto Wyss de Ingeniería Biológicamente Inspirada en la Universidad de Harvard, y Maofu Liao, Ph.D. en la Escuela de Medicina de Harvard (HMS), ha informado de un enfoque fundamentalmente nuevo para la investigación estructural de las moléculas de ARN. ROCK, como se le llama, utiliza una técnica nanotecnológica de ARN que le permite ensamblar múltiples moléculas de ARN idénticas en una estructura altamente organizada, lo que reduce significativamente la flexibilidad de las moléculas de ARN individuales y multiplica su peso molecular. Aplicado a ARN modelo bien conocidos con diferentes tamaños y funciones como puntos de referencia, el equipo demostró que su método permite el análisis estructural de las subunidades de ARN contenidas con una técnica conocida como microscopía crioelectrónica (crio-EM). Su avance se informa en Nature Methods .
"ROCK está rompiendo los límites actuales de las investigaciones estructurales del ARN y permite desbloquear estructuras 3D de moléculas de ARN a las que es difícil o imposible acceder con los métodos existentes y con una resolución casi atómica", dijo Yin, quien junto con Liao dirigió el estudio. . "Esperamos que este avance vigorice muchas áreas de investigación fundamental y desarrollo de fármacos, incluido el floreciente campo de las terapias de ARN". Yin también es líder de la Iniciativa de Robótica Molecular del Instituto Wyss y profesor en el Departamento de Biología de Sistemas en HMS.
Obteniendo control sobre el ARN
El equipo de Yin en el Instituto Wyss ha sido pionero en varios enfoques que permiten que las moléculas de ADN y ARN se autoensamblen en grandes estructuras basadas en diferentes principios y requisitos, incluidos los bloques de ADN y el origami de ADN. Ellos plantearon la hipótesis de que tales estrategias también podrían usarse para ensamblar moléculas de ARN naturales en complejos circulares altamente ordenados en los que su libertad para flexionarse y moverse está altamente restringida al unirlos específicamente. Muchos ARN se pliegan de formas complejas pero predecibles, con pequeños segmentos que se emparejan entre sí. El resultado a menudo es un "núcleo" estabilizado y "bucles de tallo" que sobresalen hacia la periferia.
"En nuestro enfoque, instalamos 'bucles de besos' que vinculan diferentes bucles de tallo periféricos que pertenecen a dos copias de un ARN idéntico de una manera que permite que se forme un anillo estabilizado general, que contiene múltiples copias del ARN de interés", dijo Di. Liu, Ph.D., uno de los dos primeros autores y becario postdoctoral en el grupo de Yin. "Especulamos que estos anillos de orden superior podrían analizarse con alta resolución mediante crio-EM, que se había aplicado a moléculas de ARN con un primer éxito".
Representación de ARN estabilizado
En crio-EM, muchas partículas individuales se congelan rápidamente a temperaturas criogénicas para evitar más movimientos y luego se visualizan con un microscopio electrónico y la ayuda de algoritmos computacionales que comparan los diversos aspectos de las proyecciones de superficie 2D de una partícula y reconstruyen su arquitectura 3D. . Peng y Liu se asociaron con Liao y su exestudiante de posgrado François Thélot, Ph.D., el otro coautor del estudio. Liao y su grupo han hecho contribuciones importantes al campo de la crio-EM que avanza rápidamente y al análisis experimental y computacional de partículas individuales formadas por proteínas específicas.
"Cryo-EM tiene grandes ventajas sobre los métodos tradicionales para ver detalles de alta resolución de moléculas biológicas, incluidas proteínas, ADN y ARN, pero el tamaño pequeño y la tendencia a moverse de la mayoría de los ARN impiden la determinación exitosa de las estructuras de ARN. Nuestro método novedoso para ensamblar multímeros de ARN resuelve estos dos problemas al mismo tiempo, al aumentar el tamaño del ARN y reducir su movimiento", dijo Liao, quien también es profesor asociado de biología celular en HMS. "Nuestro enfoque ha abierto la puerta a la determinación rápida de la estructura de muchos ARN mediante crio-EM". La integración de la nanotecnología de ARN y los enfoques crio-EM llevaron al equipo a nombrar su método "Crio-EM habilitado para la oligomerización de ARN mediante la instalación de bucles de besos" (ROCK).
Para proporcionar una prueba de principio para ROCK, el equipo se centró en un ARN de intrón grande de Tetrahymena, un organismo unicelular, y un ARN de intrón pequeño de Azoarcus, una bacteria fijadora de nitrógeno, así como el llamado ribointerruptor FMN. . Los ARN de intrones son secuencias de ARN no codificantes dispersas a lo largo de las secuencias de ARN recién transcritos y tienen que ser "empalmados" para que se genere el ARN maduro. El ribointerruptor FMN se encuentra en los ARN bacterianos involucrados en la biosíntesis de metabolitos de flavina derivados de la vitamina B2. Al unirse a uno de ellos, el mononucleótido de flavina (FMN), cambia su conformación 3D y suprime la síntesis de su ARN madre.
"El ensamblaje del intrón del grupo I de Tetrahymena en una estructura similar a un anillo hizo que las muestras fueran más homogéneas y permitió el uso de herramientas computacionales que aprovechan la simetría de la estructura ensamblada. Si bien nuestro conjunto de datos es relativamente modesto en tamaño, las ventajas innatas de ROCK nos permitieron para resolver la estructura a una resolución sin precedentes", dijo Thélot. "El núcleo del ARN se resuelve en 2,85 Å [un Ångström es diez mil millones (EE. UU.) de un metro y la métrica preferida utilizada por los biólogos estructurales], lo que revela características detalladas de las bases de nucleótidos y la columna vertebral de azúcar. No creo que podamos podría haber llegado allí sin ROCK, o al menos no sin muchos más recursos".
Cryo-EM también puede capturar moléculas en diferentes estados si, por ejemplo, cambian su conformación 3D como parte de su función. Al aplicar ROCK al ARN del intrón Azoarcus y al riboconmutador FMN, el equipo logró identificar las diferentes conformaciones por las que pasa el intrón Azoarcus durante su proceso de autoempalme y revelar la rigidez conformacional relativa del sitio de unión del ligando del riboconmutador FMN. .
"Este estudio realizado por Peng Yin y sus colaboradores muestra elegantemente cómo la nanotecnología de ARN puede funcionar como un acelerador para avanzar en otras disciplinas. Ser capaz de visualizar y comprender las estructuras de muchas moléculas de ARN naturales podría tener un impacto tremendo en nuestra comprensión de muchos aspectos biológicos y patológicos. procesos a través de diferentes tipos de células, tejidos y organismos, e incluso permiten nuevos enfoques de desarrollo de fármacos", dijo el Director Fundador de Wyss, Donald Ingber, M.D., Ph.D. Nuevos conocimientos sobre las estructuras y los mecanismos de proteínas clave involucradas en la fotosíntesis microbiana