Diseño del chip RAGE. Crédito:por Xu Teng
Una nueva técnica de secuenciación basada en funciones que utiliza pinzas ópticas y aprovecha las propiedades de la gravedad permite a los investigadores analizar las células bacterianas una por una. El estudio, realizado por investigadores del Instituto de Bioenergía y Tecnología de Bioprocesos de Qingdao (QIBEBT) de la Academia de Ciencias de China, fue publicado en Pequeña el 9 de junio.
Las células bacterianas son tan pequeñas que ha sido muy difícil analizar una sola célula bacteriana, o bacteria, a la vez. Como resultado, muchos de ellos, a veces millones a la vez, tienen que ser analizados simultáneamente. Esto nos dice mucho sobre el grupo en su conjunto, pero evita que los investigadores puedan investigar el vínculo entre el genotipo de una sola bacteria, o conjunto completo de genes; y su fenotipo, o el conjunto de características que resultan de la interacción de sus genes y el medio ambiente.
Una forma sencilla de pensar en la distinción entre genotipo y fenotipo es observar que, si bien el genotipo de una sola planta de maíz podría permitirle crecer tres pies de altura, si no se aplica mucho fertilizante, entonces, el fenotipo de la planta de maíz podría ser que solo creciera hasta dos pies de altura.
El análisis del vínculo entre genotipo y fenotipo es sencillo para un organismo tan grande, y muy útil también.
Conocimientos similares sobre la relación genotipo-fenotipo de una sola bacteria, sobre todo con respecto a las enfermedades infecciosas, han sido buscados durante mucho tiempo pero obstaculizados por el tamaño de una bacteria, que suelen tener unas pocas millonésimas de metro de longitud.
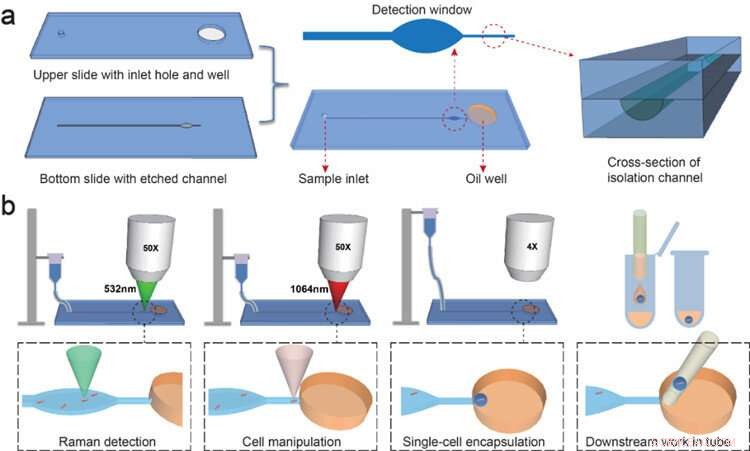
Investigadores del Single-Cell Center en QIBEBT han desarrollado una técnica de creación de perfiles de bacterias llamada Encapsulación y secuenciación unicelular impulsada por gravedad activada por Raman. o secuenciación de RAGE. En la técnica, los fenotipos de las células individuales se analizan uno por uno, luego se empaqueta cuidadosamente en una 'microgotita de picolitro' (una billonésima parte de un litro) que se exporta e indexa en una célula por tubo de ensayo, lista para la secuenciación de genes más adelante.
El proceso implica un 'chip' RAGE de dos capas de cuarzo unidas entre sí y que tienen un orificio de entrada, pozo de petróleo, y micro-canales grabados en ellos. 'Pinzas ópticas', o un rayo láser altamente enfocado que produce una fuerza atractiva o repulsiva, manipula la bacteria en líquido a través del canal, asistido por la gravedad.
La forma, La estructura y las características metabólicas de la bacteria, esencialmente su fenotipo, se investigan luego a través de una ventana de detección utilizando 'espectroscopía Raman', una técnica analítica que aprovecha la interacción de la luz con los enlaces químicos dentro de un material.
"Finalmente, la bacteria se encapsula en la microgotita, que luego se transfiere a un tubo para la secuenciación de genes o el cultivo de la célula, "dijo el profesor Ma Bo, autor correspondiente del estudio.
El empaque de microgotas es extremadamente importante, ya que permite que la cantidad muy pequeña de ADN en una sola célula bacteriana se amplifique de manera muy uniforme, un desafío clave para la decodificación completa de su genoma, según Xu Teng, un estudiante de posgrado en el equipo que desarrolló el método.
"Somos capaces de, directamente de una muestra de orina, obtener características de resistencia a los antibióticos y una secuencia del genoma esencialmente completa de forma simultánea a partir de una sola célula. Esto ofrece la resolución más alta posible para el diagnóstico bacteriano y el tratamiento farmacológico. "Dijo el profesor Xu Jian, otro autor correspondiente del estudio.
Basado en esta tecnología, los investigadores han desarrollado un instrumento llamado CAST-R para respaldar la selección rápida de antibióticos y la secuenciación del genoma de patógenos, todo al nivel de una celda. Este instrumento significa un tratamiento antibiótico mucho más rápido y preciso, y una sensibilidad mucho mayor para rastrear y combatir la resistencia bacteriana a los antibióticos, que es una gran amenaza para el futuro de la sociedad humana.