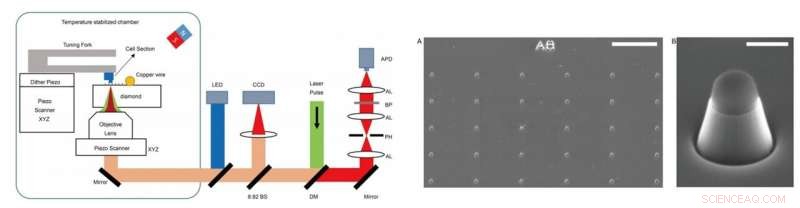
IZQUIERDA:configuración experimental. El experimento se llevó a cabo en una instalación construida en casa, que combinó microscopía de resonancia magnética detectada ópticamente (ODMR) con microscopía de fuerza atómica (AFM). DM:espejo dicroico. BP:filtro de paso de banda que trabaja a 650–775 nm. APD:fotodiodo de avalancha. CCD:dispositivo de carga acoplada. LED:diodo emisor de luz de 470 nm. AL:lente acromática. PH:agujero de alfiler con un tamaño de 30 μm. BS:divisor de haz. DERECHA - Imágenes de los nanopilares sobre diamantes. (A) Imágenes SEM de los nanopilares de diamante fabricados justo después del grabado con iones reactivos (RIE). La parte superior del nanopilar está cubierta por silsesquioxano de hidrógeno (HSQ) para proteger el centro NV. (B) Un solo nanopilar con forma de cilindro trapezoidal para detectar secciones de células adheridas en la punta de AFM. Barras de escala, 10 µm (A); 400 nm (B). Crédito:Science Advances, doi:10.1126 / sciadv.aau8038.
En ciencias de la vida, la capacidad de medir la distribución de biomoléculas dentro de una célula in situ es un objetivo de investigación importante. Entre una variedad de técnicas, Los científicos han utilizado imágenes magnéticas (MI) basadas en el centro de vacantes de nitrógeno (NV) en diamantes como una herramienta poderosa en la investigación biomolecular. Sin embargo, La obtención de imágenes a nanoescala de proteínas intracelulares sigue siendo un desafío hasta ahora. En un estudio reciente ahora publicado en Avances de la ciencia , Pengfei Wang y sus colegas de los departamentos interdisciplinarios de física, biomacromoléculas, la información cuántica y las ciencias de la vida en China, usó proteínas de ferritina para demostrar la realización de MI de proteínas endógenas en una sola célula, utilizando el centro de nitrógeno vacante (NV) como sensor. Obtuvieron imágenes de ferritinas intracelulares y orgánulos que contienen ferritina utilizando MI y microscopía electrónica correlativa para allanar el camino para la obtención de imágenes magnéticas (MI) a nanoescala de proteínas intracelulares.
Se requiere aumentar la resolución espacial existente de imágenes biomédicas para lograr las demandas continuas en imágenes médicas, y por lo tanto, entre una variedad de técnicas, Las imágenes magnéticas son de gran interés en la actualidad. La resonancia magnética (MRI) se usa ampliamente para cuantificar la distribución de espines nucleares, pero la MRI convencional solo puede alcanzar una resolución de 1 µm en imágenes de espín nuclear donde la resolución está limitada por la sensibilidad de detección eléctrica. Los científicos han desarrollado una serie de técnicas para romper esta barrera de resolución, incluyendo un dispositivo superconductor de interferencia cuántica y microscopía de fuerza de resonancia magnética. Sin embargo, estos informes requieren un entorno criogénico y un alto vacío para la obtención de imágenes, limitando la implementación experimental y su traducción a la práctica clínica.
Un método de detección cuántica desarrollado recientemente basado en el centro de vacantes de nitrógeno en el diamante ha empujado radicalmente el límite de las técnicas de MI a nanoescala para detectar moléculas orgánicas y proteínas en el laboratorio. Los científicos han combinado la detección cuántica con los centros de NV y la microscopía de sonda de barrido para demostrar la resonancia magnética a nanoescala para el espín de un solo electrón y el conjunto de espín nuclear pequeño mientras utilizan el centro de NV como un magnetómetro biocompatible para obtener imágenes no invasivas de partículas ferromagnéticas dentro de las células en la escala subcelular (0,4 µm). Por ejemplo, La despolarización del centro NV se puede utilizar como un magnetómetro de banda ancha para detectar y medir el ruido fluctuante de los iones metálicos y los espines nucleares. Sin embargo, Hasta el momento, no se ha informado de tales imágenes de proteínas individuales a través de MI a nanoescala en una sola célula.
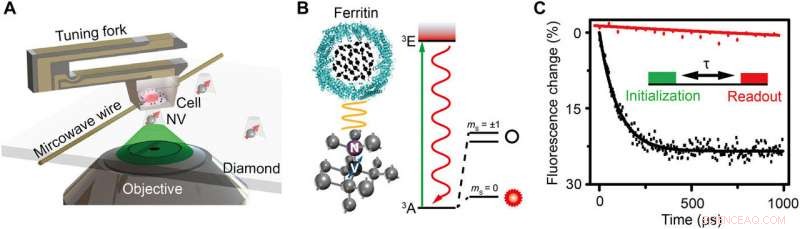
Esquema de la configuración y principio experimental. (A) Vista esquemática de la configuración experimental. La celda incrustada en resina se une a un diapasón y se escanea por encima del nanopilar de diamante que contiene un centro NV poco profundo. Se utiliza un cable de cobre para enviar el pulso de microondas al centro NV. Se utiliza un láser verde (532 nm) del microscopio confocal (CFM) para abordar, inicializar, y leer el centro NV. (B) Izquierda:celosía cristalina y nivel de energía del centro NV. El centro NV es un defecto puntual que consta de un átomo de nitrógeno sustitutivo y una vacante adyacente en el diamante. Derecha:vista esquemática de una ferritina. Las flechas negras indican los giros de electrones de Fe3 +. (C) Demostración experimental de la detección de ruido de espín con y sin ferritina en forma de decaimiento de polarización para el mismo centro NV. El recuadro es la secuencia de pulsos para la detección y formación de imágenes de la ferritina. Se utiliza un láser verde de 5 μs para inicializar el estado de giro a ms =0, seguido de un tiempo de evolución libre τ para acumular el ruido magnético, y finalmente se lee el estado de giro detectando la intensidad de la fluorescencia. La secuencia de pulsos se repite unas 105 veces para adquirir una buena relación señal / ruido (SNR). El tiempo de relajación se ajusta a 0,1 y 3,3 ms por caída exponencial para el caso con y sin ferritina, respectivamente, lo que indica un ruido de giro de 0,01 mT2. Crédito:Science Advances, doi:10.1126 / sciadv.aau8038.
En el presente trabajo, Wang y col. informó de dos avances técnicos para permitir MI a nanoescala de proteínas intracelulares dentro de una sola célula. Para esto, congelaron la celda a un estado sólido y la segmentaron intrincadamente en forma de cubo, luego lo colocó en una sonda de escaneo de diapasón de un microscopio de fuerza atómica (AFM) para obtener imágenes, donde la sección transversal plana de la celda estaba expuesta al aire. Los científicos utilizaron la configuración de colocación de la muestra para permitir que el sensor NV se colocara dentro de los 10 nm de las proteínas objetivo y utilizaron el AFM para suprimir la deriva térmica durante el posicionamiento de la muestra. Luego diseñaron nanopilares trapezoidales en forma de cilindro en una superficie de diamante a granel para la adquisición de imágenes. acortando técnicamente el tiempo de adquisición de imágenes en un orden en comparación con los métodos anteriores. En el presente estudio, Los científicos utilizaron esta técnica para realizar MI in situ del ruido fluctuante magnético de las proteínas de ferritina intracelular (un biomarcador de las reservas de hierro y la saturación de transferrina en el cuerpo) dentro de la configuración experimental.
La ferritina es un complejo proteico globular con un diámetro exterior de 12 nm, que contiene una cavidad de 8 nm de diámetro que permite almacenar hasta 4500 átomos de hierro dentro de la proteína. El ruido magnético de los iones férricos se puede detectar debido a sus efectos sobre la T 1 tiempo de relajación de un centro NV. En este trabajo, Wang y col. confirmó la observación utilizando medidas de fluorescencia de la desintegración dependiente del tiempo de la población de centros NV (espín magnético, metro S =0 estado), en una superficie de diamante recubierta de ferritinas. Adicionalmente, los científicos detectaron el ruido magnético con métodos sin etiquetas utilizando el centro NV a través de microscopía electrónica de transmisión (TEM). The work allowed the development of a correlated MI and TEM scheme to obtain and verify the first nanoscale MI of a protein in situ.
The scientists used the hepatic carcinoma cell line (HepG2) for the experiments and studied iron metabolism by treating the cells with ferric ammonium citrate (FAC), which significantly increased the amount of intracellular ferritin. They verified this using confocal microscopy (CFM), western blotting and TEM techniques at first. The results showed the primary localization of ferritins in the intracellular puncta around the nucleus, among the cytoplasm. The scientists used bulk electron paramagnetic resonance (EPR) spectroscopy to confirm the paramagnetic properties of ferritin in the FAC-treated HepG2 cells and mass spectroscopy to measure the interference due to other paramagnetic metal ions.
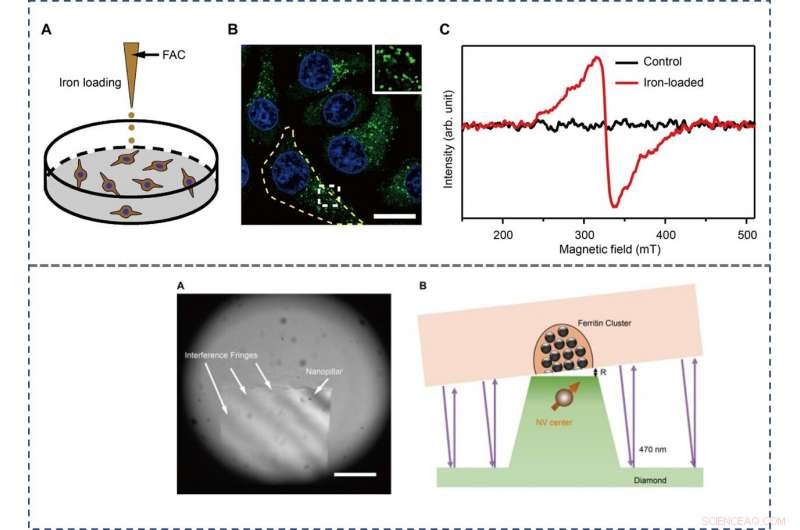
TOP - The preparation and characterization of ferritin-rich HepG2 cell samples. (A) Schematic view of the treatment to cultured cells. Following iron loading or no treatment, the HepG2 cells were examined for fluorescence images and EPR spectra, respectivamente. For the MI and TEM imaging, cell samples were treated through high-pressure freezing, freeze substitution, and sectioning. (B) Representative confocal microscopy (CFM) image of ferritin structures (green) in iron-loaded HepG2 cells. The ferritin proteins were immunostained by anti-ferritin light chain antibody. The nuclei are indicated by 4′, 6-diamidino-2-phenylindole (DAPI) in the blue channel. Inset displays magnified ferritin structures. The yellow dashed line outlines the contour of a cell. Barra de escala, 20 μm. (C) EPR spectra of control and iron-loaded HepG2 cells at T =300 K. BOTTOM - Adjusting the distance between the NV center and the cell section. (A) Interference fringes between the cell cube and the diamond surface. Barra de escala, 20 μm. (B) The geometric relation and the gap R between cell samples and diamond-pillars for MI. The top surface diameter of the nanopillar is 400 nm. Credit:Science Advances, doi:10.1126/sciadv.aau8038.
Wang y col. then used ultrafast, high-pressure freezing to immobilize all intracellular components of the Fe-loaded cells. The process stabilized the intracellular structures and molecules by minimizing Brownian motion in cells, which typically contributes to random motion of proteins up to 100 nm in vivo. To image the samples, they embedded and polymerized the frozen cells in LR White medium, followed by gluing the embedded cell sample to the AFM tuning fork with a few cells at the tip. Using a diamond knife, the scientists then sectioned the tip surface to nanometer flatness to examine the cuboid cell section under AFM. They acquired MI images of ferritins by scanning the cell cube along the diamond nanopillars and simultaneously measured NV spin repolarization rate using the "leapfrog" scanning mode of the microscope as detailed previously.
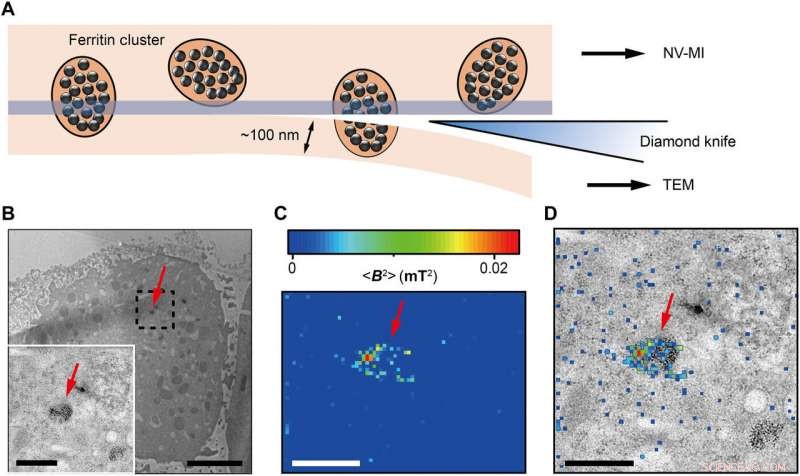
Correlative MI and TEM images. (A) Schematic view of sectioning for correlative MI and TEM imaging. The last section and the remaining cube were transferred for TEM imaging and MI scanning, respectivamente. The sectioning resulted in some split ferritin clusters that could be imaged under both microscopes. A transparent blue strip of ~10 nm indicates the imaging depth of the MI, while in the TEM, the imaging depth is ~100 nm. (B) Distribution of ferritins from the last ultrathin section under TEM. Inset:Magnified figure of the part in black dashed box. (C) MI result of the remaining cell cube. The pixel size is 43 nm. (D) The merged MI and TEM micrograph shows ferritins in a membrane-bound organelle. The red arrows in (B) to (D) indicate the same ferritin cluster. Scale bars, 5 μm (B) and 1 μm [B (inset), C, and D]. Credit:Science Advances, doi:10.1126/sciadv.aau8038.
The scientists measured fluorescence decay at a fixed free evolution time of 50 microseconds (τ =50 μs) to reveal the degree of NV sensor spin polarization, which correlated with the amount of ferritin in the sensing volume. They observed the appearance of some clusters via both TEM and MI images, although some details were not observed in MI, the results confirmed that spin noise from intracellular ferritin contributed to depolarize the NV center. In order to obtain details of the ferritin clusters at higher resolution, the scientists minimized the pixel size to 8.3 nm and acquired MI of high resolution of the proteins as expected.
De este modo, Wang y col. explored the sensitivity of NV centers as an appropriate sensor for biological imaging applications at the level of the single molecule. They used the technique as a sensor in the experimental setup to obtain the first MI of a protein at a resolution of 10 nm in situ. The scientists aim to improve the stability and sensitivity of the technique to speed up the scanning process and image a larger area of interest in the cell and locate ferritin beyond the nucleus in association with additional organelles.
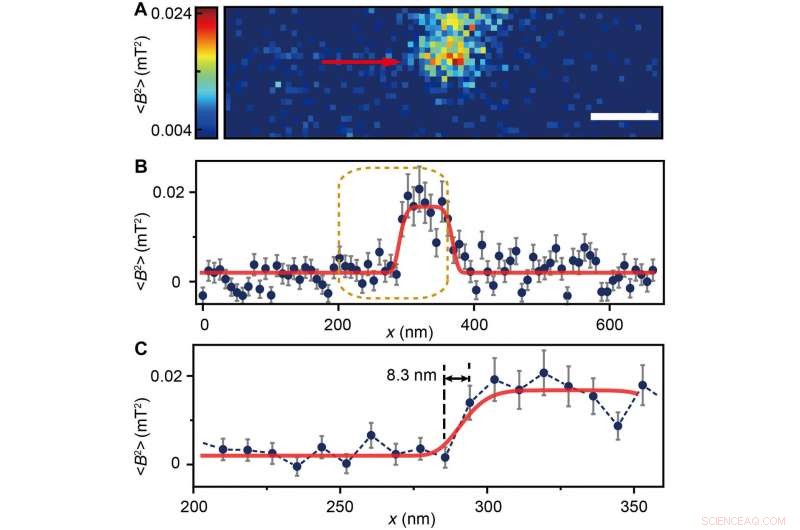
(A) Ferritin cluster imaged by the NV sensor with 80 × 24 pixels and a pixel size of 8.3 nm. Barra de escala, 100 nm. (B) Trace data of the scanning line in (A) directed by the red arrow. The platform indicates the ferritin cluster. The red curve fitted by a plateau function serves as a guide to the eye. (C) Magnified figure of the gold dashed box in (B). The sharp transition indicated by the red arrow around x =283 nm shows the scanning from the blank area to the area with ferritins. Credit:Science Advances, doi:10.1126/sciadv.aau8038.
The work will contribute to clinical diagnostics to determine biomarker-based iron storage and release in cells. This will include studies on the regulatory mechanisms of iron metabolism during the progression of hemochromatosis, anemia, liver cirrhosis and Alzheimer's disease. Wang y col. propose to extend the approach in situ to other cellular components with paramagnetic signals, including magnetic molecules, metalloproteins and special spin-labelled proteins. The scientists envision that further studies will explore additional targets suitable for high-resolution MI and correlated TEM imaging techniques, with optical microscopy detection incorporated to the experimental setup to extend the work and determine protein nuclear spin MRI as well as perform three-dimensional cell tomography.
© 2019 Science X Network