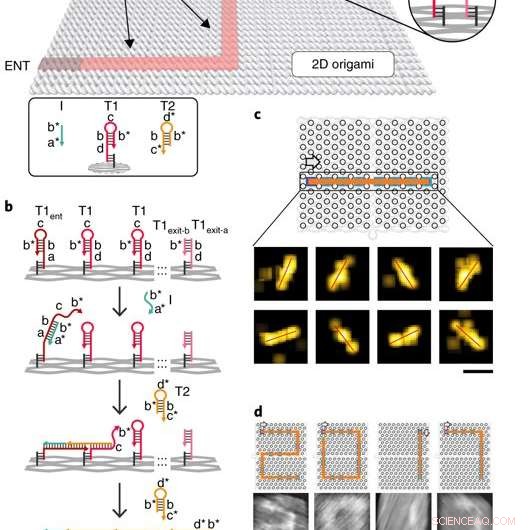
Implementación del navegador de ADN de molécula única. a) Ilustración esquemática del sistema PSEC. Se construye un gráfico conectado acíclico (es decir, un árbol) sobre un sustrato de origami de ADN rectangular de 100 x 70 nm ^ 2. T1 y T2 son dos tipos de combustibles que impulsan el PSEC en el árbol. El iniciador I se utilizó para desencadenar el inicio del PSEC desde el vértice de entrada ENT. b) mecanismo de propagación del sistema PSEC. El iniciador I reconoce y abre la horquilla T1ent en el vértice ENT. La horquilla abierta luego captura y abre una horquilla T2 del entorno para iniciar la cascada. c) Una línea recta pavimentada por PSEC y visualizada con DNA-PAINT. d) Cuatro dígitos pavimentados de “2017” fotografiados usando microscopía de fuerza atómica (AFM). Las horquillas T1 en los puntos de las esquinas se modificaron para evitar tramos no deseados. Las flechas indican la dirección de propagación. Crédito: Materiales de la naturaleza , doi:10.1038 / s41563-018-0205-3.
El campo de la nanorobótica inteligente se basa en la gran promesa de los dispositivos moleculares con capacidades de procesamiento de información. En un nuevo estudio que respalda la tendencia de los portadores de información basados en ADN, Los científicos han diseñado un sistema de navegación de ADN que puede funcionar con una sola molécula, paralelo, operaciones de búsqueda en profundidad en una plataforma de origami bidimensional.
Las operaciones de búsqueda de caminos con navegadores de ADN utilizan un proceso de cascada de intercambio de hebras localizado iniciado en un sitio de activación único en la plataforma de origami. La progresión automática a lo largo de las rutas está habilitada por horquillas de ADN que contienen una secuencia transversal universal. Por diseño, cada navegador de una sola molécula puede explorar de forma autónoma cualquiera de los posibles caminos a través de un árbol con raíces de 10 vértices construido en el estudio. Los laberintos equivalían a un árbol con una entrada en la raíz y una salida por una de las hojas. El estudio realizado por Jie Chao y sus colaboradores resultó en la exploración de todos los caminos tomados por los navegadores de ADN para extraer una ruta de solución específica que conectaba un par dado de vértices de inicio y final en el laberinto. Como resultado, la ruta de la solución se colocó claramente en la plataforma de origami y se ilustró utilizando imágenes de una sola molécula. El enfoque ahora se publica en Materiales de la naturaleza , detallando la realización de materiales moleculares con funciones computacionales biomoleculares integradas para operar al nivel de una sola molécula con potencial para diseñar nanorobots inteligentes para futuras aplicaciones en la industria y la medicina.
En el pasado se utilizaron sofisticadas herramientas moleculares para crear máquinas moleculares que convierten productos químicos, energía fotónica o eléctrica en movimientos rotativos o lineales a nanoescala. Por ejemplo, El movimiento browniano a nanoescala se puede convertir de forma controlable en movimientos dirigidos dentro de nanomáquinas basadas en ADN mediante reacciones de hibridación de ADN. Estas máquinas basadas en ADN funcionan de forma autónoma siguiendo un "programa molecular" integrado, prediseñado como una reacción en cascada que se activa manualmente mediante un estímulo externo para cada paso de la operación.
El enfoque del campo ha cambiado progresivamente para actualizar los circuitos lógicos basados en ADN utilizando aptámeros y ADNzimas para diseñar puertas lógicas moleculares. Por ejemplo, en 2006, Stojanovic y sus colaboradores integraron más de 100 puertas lógicas de ADN para diseñar una automatización llamada MAYA-II para jugar un juego de Tic-Tac-Toe. Estudios anteriores demostraron un sistema informático libre de enzimas basado en reacciones en cadena de hibridación (HCR) para crear puertas lógicas y circuitos lógicos para un rendimiento más robusto y eficiente que los sistemas originales.
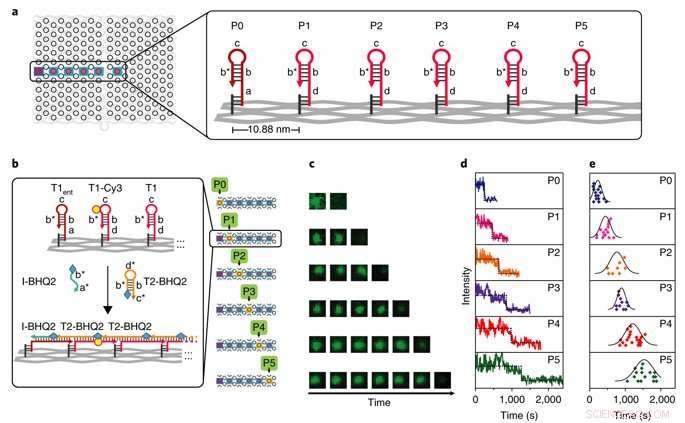
Caracterización de una sola molécula de la cinética de PSEC. a) Se utilizó una línea recta en el medio del origami de ADN como banco de pruebas. El vértice P0 es el punto de partida y los vértices P1-P5 son puntos intermedios. b) Detalles del diseño de seis pruebas paralelas para medir la cinética con TIRF resuelto en el tiempo. Se usó T2 marcado con BHQ2 para apagar el T1 marcado con Cy3, ensamblando un perfil cinético en cada paso. El ejemplo ilustrado muestra el mecanismo de enfriamiento. c) Las imágenes TIRF de ejemplo muestran el cambio de fluorescencia con el tiempo en las seis pruebas paralelas de P0 a P5. d) Trazos típicos de fluorescencia de una sola molécula utilizados para monitorear los eventos de extinción que ocurren en los vértices P0 a P5. e) Gráficos de dispersión que muestran el análisis estadístico de la distribución de longitudes por trayecto. Crédito: Materiales de la naturaleza , doi:10.1038 / s41563-018-0205-3.
En el presente estudio de Chao et al, Se utilizó el mismo principio básico del esquema de reacción de HCR en un contexto computacional diferente para desarrollar un sistema de navegador de ADN de una sola molécula. La plataforma exploró todos los caminos posibles a través de un gráfico de árbol diseñado en una estructura de origami como un laberinto simplemente conectado sin caminos cíclicos. Estas estructuras de origami de ADN son nanoestructuras portadoras de información por naturaleza con geometría a nanoescala bien definida. El laberinto podría explorarse mediante la cascada de intercambio de hebra proximal (PSEC) basada en reacciones en cadena de hibridación. Los investigadores demostraron que un sistema con una gran cantidad de navegadores de ADN de una sola molécula podría realizar colectivamente búsquedas paralelas en profundidad (PDFS) en el árbol para realizar de manera eficiente la resolución de laberintos con origami 2-D. Inicialmente, los investigadores realizaron estudios para probar el diseño de PSEC.
El sistema de cascada de intercambio de hebra proximal (PSEC) (principio de funcionamiento del navegador de ADN) se facilitó en un sustrato de origami rectangular hecho de tres componentes, que incluía la implementación física de un gráfico de árbol, hebras completas y una hebra iniciadora. Las áreas vacías sin extensiones de grapas correspondían a paredes en el laberinto, impidiendo la propagación de la cascada de intercambio de cadenas. La entrada y la salida se definieron y se indicaron como ENT y EXIT respectivamente. En el segundo componente, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
Por diseño, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
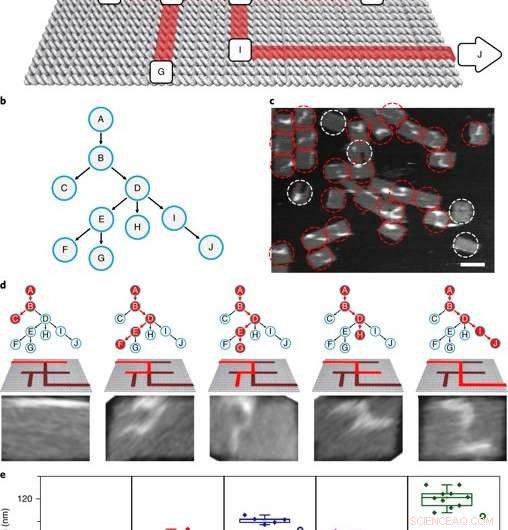
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Solo P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Crédito: Materiales de la naturaleza , doi:10.1038/s41563-018-0205-3.
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, D, E) an intermediate vertex (I) and four dead ends (C, F, GRAMO, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
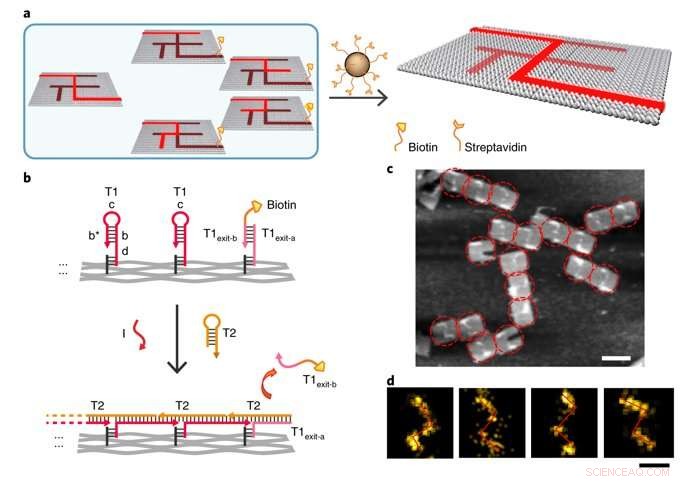
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Crédito: Materiales de la naturaleza , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Como resultado, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Science X Network