El ácido ribonucleico (ARN) se utiliza para crear circuitos lógicos capaces de realizar varios cálculos. En nuevos experimentos, Green y sus colegas han incorporado puertas lógicas de ARN en células bacterianas vivas, que actúan como pequeñas computadoras. Crédito:Jason Drees para el Biodesign Institute
El nexo interdisciplinario de biología e ingeniería, conocida como biología sintética, está creciendo a un ritmo rápido, abriendo nuevas perspectivas que apenas podían imaginarse hace poco tiempo.
En una nueva investigación, Alex Green, profesor del Instituto de Biodiseño de ASU, demuestra cómo se puede inducir a las células vivas a realizar cálculos a la manera de pequeños robots o computadoras.
Los resultados del nuevo estudio tienen implicaciones significativas para el diseño inteligente de fármacos y la administración inteligente de fármacos. producción de energía verde, tecnologías de diagnóstico de bajo costo e incluso el desarrollo de nanomáquinas futuristas capaces de detectar células cancerosas o desactivar genes aberrantes.
"Estamos utilizando interacciones ARN-ARN muy predecibles y programables para definir lo que pueden hacer estos circuitos, "dice Green." Eso significa que podemos usar software de computadora para diseñar secuencias de ARN que se comporten de la manera que queremos que lo hagan en una célula. Hace que el proceso de diseño sea mucho más rápido ".
El estudio aparece en la edición avanzada en línea de la revista. Naturaleza .
Diseñador de ARN
El enfoque descrito utiliza circuitos compuestos de ácido ribonucleico o ARN. Estos diseños de circuitos, que se asemejan a los circuitos electrónicos convencionales, autoensamblado en células bacterianas, permitiéndoles detectar los mensajes entrantes y responder a ellos produciendo una salida computacional particular, (en este caso, una proteína).
En el nuevo estudio, En el laboratorio se diseñaron circuitos especializados conocidos como puertas lógicas, luego se incorpora a las células vivas. Los diminutos interruptores del circuito se activan cuando los mensajes (en forma de fragmentos de ARN) se adhieren a sus secuencias de ARN complementarias en el circuito celular, activando la puerta lógica y produciendo una salida deseada.
Los interruptores de ARN se pueden combinar de varias maneras para producir puertas lógicas más complejas capaces de evaluar y responder a múltiples entradas, al igual que una computadora simple puede tomar varias variables y realizar operaciones secuenciales como sumas y restas para llegar a un resultado final.
El nuevo estudio mejora drásticamente la facilidad con la que se puede realizar la computación celular. El enfoque de solo ARN para producir nanodispositivos celulares es un avance significativo, ya que los esfuerzos anteriores requirieron el uso de intermediarios complejos, como proteínas. Ahora, las partes necesarias de ribocomputación se pueden diseñar fácilmente en la computadora. Las propiedades simples de emparejamiento de bases de las cuatro letras de nucleótidos del ARN (A, C, G y U) aseguran el autoensamblaje y el funcionamiento predecibles de estas partes dentro de una célula viva.
El trabajo de Green en esta área comenzó en el Instituto Wyss de Harvard, donde ayudó a desarrollar el componente central utilizado en los circuitos celulares, conocido como un interruptor de pie de ARN. El trabajo se llevó a cabo mientras Green era un postdoctorado trabajando con el experto en nanotecnología Peng Yin, junto con los biólogos sintéticos James Collins y Pamela Silver, que son todos coautores del nuevo artículo. "Los primeros experimentos fueron en 2012, "Green dice". Básicamente, los interruptores de pie funcionaban tan bien que queríamos encontrar la manera de aprovecharlos mejor para aplicaciones móviles ".
Después de llegar a ASU, El primer estudiante de posgrado de Green, Duo Ma, trabajó en experimentos en el Biodesign Institute, mientras que otro postdoctorado, Jongmin Kim continuó con un trabajo similar en el Instituto Wyss. Ambos también son coautores del nuevo estudio.
Chip Pentium de la naturaleza
La posibilidad de utilizar ADN y ARN, las moléculas de la vida, para realizar cálculos similares a los de una computadora fue demostrado por primera vez en 1994 por Leonard Adleman de la Universidad del Sur de California. Desde entonces, el rápido progreso ha avanzado considerablemente en el campo, y recientemente, tal computación molecular se ha logrado dentro de las células vivas. (Las células bacterianas generalmente se emplean para este propósito, ya que son más simples y fáciles de manipular).
La técnica descrita en el nuevo artículo aprovecha el hecho de que el ARN, a diferencia del ADN, es monocatenario cuando se produce en células. Esto permite a los investigadores diseñar circuitos de ARN que se pueden activar cuando una cadena de ARN complementaria se une a una secuencia de ARN expuesta en el circuito diseñado. Esta unión de hebras complementarias es regular y predecible, con nucleótidos A siempre emparejándose con U y C siempre emparejándose con G.
Con todos los elementos de procesamiento del circuito hechos con ARN, que puede asumir un número astronómico de secuencias potenciales, el verdadero poder del método recién descrito radica en su capacidad para realizar muchas operaciones al mismo tiempo. Esta capacidad de procesamiento paralelo permite un cálculo más rápido y sofisticado al tiempo que hace un uso eficiente de los recursos limitados de la celda.
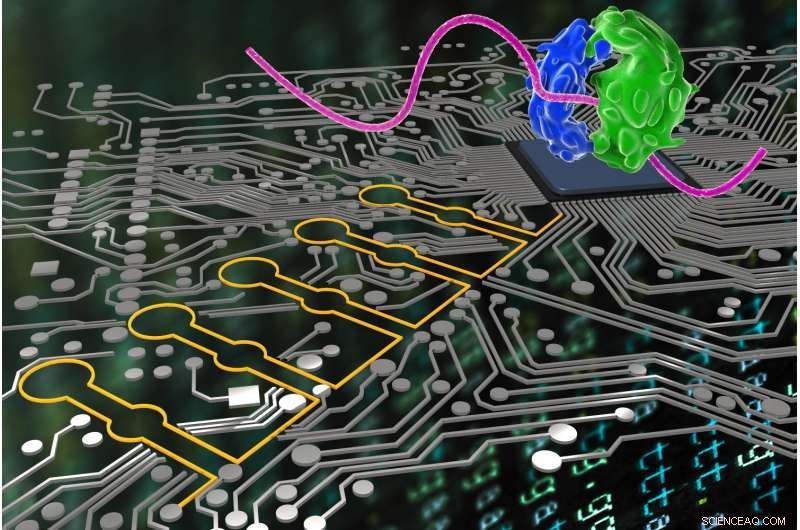
Similar a cómo los científicos de la computación usan el lenguaje lógico para que sus programas sean precisos Y, O y NO decisiones hacia un objetivo final, Los biólogos sintéticos pueden utilizar ahora los "dispositivos de ribocomputación" (estilizados aquí en amarillo) desarrollados por un equipo del Instituto Wyss para detectar e interpretar múltiples señales en las células e instruir lógicamente a sus ribosomas (estilizados en azul y verde) para que produzcan diferentes proteínas. Crédito:Instituto Wyss de la Universidad de Harvard
Resultados lógicos
En el nuevo estudio, puertas lógicas conocidas como AND, O y NO fueron diseñados. Una puerta AND produce una salida en la celda solo cuando están presentes dos mensajes de ARN A Y B. Una puerta OR responde a A O B, mientras que una puerta NOT bloqueará la salida si está presente una entrada de ARN determinada. La combinación de estas puertas puede producir una lógica compleja capaz de responder a múltiples entradas.
Usando interruptores de pie de ARN, los investigadores produjeron los primeros dispositivos de ribocomputación capaces de Y de cuatro entradas, OR de seis entradas y un dispositivo de 12 entradas capaz de realizar una combinación compleja de Y, OR y NOT lógica conocida como expresión de forma normal disyuntiva. Cuando la puerta lógica encuentra las secuencias de unión de ARN correctas que conducen a la activación, se abre un interruptor de pie y tiene lugar el proceso de traducción a proteína. Todas estas funciones de detección de circuitos y de salida se pueden integrar en la misma molécula, haciendo que los sistemas sean compactos y más fáciles de implementar en una celda.
La investigación representa la siguiente fase del trabajo en curso utilizando los interruptores de pie de ARN altamente versátiles. En un trabajo anterior, Green y sus colegas demostraron que un Un conjunto de interruptores de pie de ARN en papel podría actuar como una plataforma de alta precisión para diagnosticar el virus del Zika. La detección de ARN viral por la matriz activó los interruptores del pie, desencadenando la producción de una proteína, que se registró como un cambio de color en la matriz.
El principio básico de utilizar dispositivos basados en ARN para regular la producción de proteínas se puede aplicar a prácticamente cualquier entrada de ARN, marcando el comienzo de una nueva generación de diagnósticos de bajo costo para una amplia gama de enfermedades. El enfoque sin células es especialmente adecuado para las amenazas emergentes y durante los brotes de enfermedades en el mundo en desarrollo. donde los recursos médicos y el personal pueden ser limitados.
La computadora dentro
Según Green, la siguiente etapa de la investigación se centrará en el uso de la tecnología de apoyo de ARN para producir las llamadas redes neuronales dentro de las células vivas, circuitos capaces de analizar una variedad de entradas excitatorias e inhibitorias, promediarlos y producir un resultado una vez que se alcanza un umbral particular de actividad, de la misma manera que una neurona promedia las señales entrantes de otras neuronas. Por último, los investigadores esperan inducir a las células a comunicarse entre sí a través de señales moleculares programables, formando un verdadero interactivo, red similar a un cerebro.
"Debido a que estamos usando ARN, una molécula universal de vida, sabemos que estas interacciones también pueden funcionar en otras células, por lo que nuestro método proporciona una estrategia general que podría trasladarse a otros organismos, "Green dice, aludiendo a un futuro en el que las células humanas se conviertan en entidades totalmente programables con amplias capacidades biológicas.