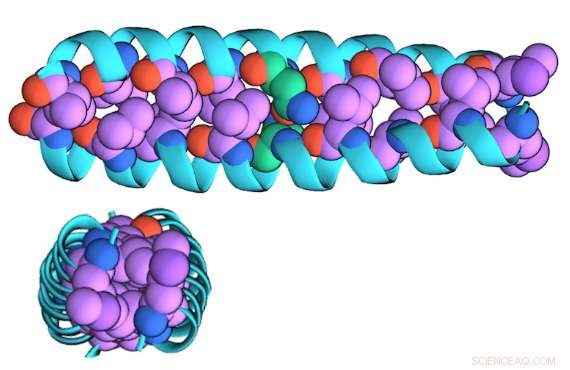
Una ilustración de la forma de dímero "comprimido" de GCN4-p1 vista desde el lado (arriba) y arriba (abajo)
Investigadores de la Universidad de Pensilvania han avanzado hacia un nuevo método de secuenciación de genes. Se leen las bases de una hebra de ADN a medida que se pasan a través de un orificio nanoscópico.
En un nuevo estudio, han demostrado que esta técnica también se puede aplicar a las proteínas como forma de conocer más sobre su estructura.
Los métodos existentes para este tipo de análisis son laboriosos, típicamente implica la recolección de grandes cantidades de proteína. También suelen requerir modificar la proteína, limitando la utilidad de estos métodos para comprender el comportamiento de la proteína en su estado natural.
La técnica de translocación de los investigadores de Penn permite el estudio de proteínas individuales sin modificarlas. Las muestras tomadas de un solo individuo podrían analizarse de esta manera, abrir aplicaciones para el diagnóstico y la investigación de enfermedades.
El estudio fue dirigido por Marija Drndić, profesor en el Departamento de Física y Astronomía de la Escuela de Artes y Ciencias; David Niedzwiecki, investigadora postdoctoral en su laboratorio; y Jeffery G. Saven, profesor en el Departamento de Química de Penn Arts &Sciences.
Fue publicado en la revista ACS Nano .
La técnica del equipo de Penn se deriva del trabajo de Drndić sobre la secuenciación de genes de nanoporos, cuyo objetivo es distinguir las bases en una hebra de ADN por el porcentaje diferente de la apertura que cada uno bloquea al pasar a través de un poro nanoscópico. Diferentes siluetas permiten que pasen diferentes cantidades de un líquido iónico. El cambio en el flujo de iones se mide mediante componentes electrónicos que rodean el poro; los picos y valles de esa señal pueden correlacionarse con cada base.
Si bien los investigadores trabajan para aumentar la precisión de estas lecturas a niveles útiles, Drndić y sus colegas han experimentado con la aplicación de la técnica a otras moléculas biológicas y estructuras a nanoescala.
Colaborando con el grupo de Saven, se propusieron probar sus poros en moléculas biológicas aún más complicadas.
"Hay muchas proteínas que son mucho más pequeñas y más difíciles de manipular que una hebra de ADN que nos gustaría estudiar, ", Dijo Saven." Estamos interesados en aprender sobre la estructura de una proteína determinada, como si existe como monómero, o combinado con otra copia en un dímero, o un agregado de múltiples copias conocido como oligómero ".
La detección también suele ser una limitación.
"No hay formas de amplificar péptidos y proteínas como las que existen para el ADN, ", Dijo Drndić." Si quieres estudiar proteínas de una fuente en particular, estás atrapado con muestras muy pequeñas. Con este método, sin embargo, simplemente puede recopilar la cantidad de datos que necesita y la cantidad de proteínas que desea que pasen a través del poro y luego estudiarlas una a la vez, ya que existen naturalmente en el cuerpo ".
Usando nanoporos de nitruro de silicio del grupo Drndić, que se puede perforar a diámetros personalizados, el equipo de investigación se propuso probar su técnica en GCN4-p1, una proteína seleccionada porque contiene un motivo estructural común que se encuentra en factores de transcripción y receptores intracelulares.
"La versión de dímero está 'comprimida' junto, "Niedzwiecki dijo:"Es una 'bobina enrollada' de hélices entrelazadas que es aproximadamente cilíndrica. La versión de monómero está descomprimida y probablemente no sea helicoidal; probablemente sea más como una cuerda".
Los investigadores pusieron diferentes proporciones de versiones con y sin cremallera de la proteína en un fluido iónico y las pasaron a través de los poros. Si bien no puede distinguir la diferencia entre proteínas individuales, los investigadores pudieron realizar este análisis en poblaciones de la molécula.
"La forma de dímero y monómero de la proteína bloquea un número diferente de iones, por lo que vemos una caída diferente en la corriente cuando atraviesan el poro, ", Dijo Niedzwiecki." Pero obtenemos un rango de valores para ambos, ya que no todos los eventos de translocación molecular son iguales ".
Determinar si una muestra específica de estos tipos de proteínas se está agregando o no podría usarse para comprender mejor la progresión de la enfermedad.
"Muchos investigadores, "Saven dijo, "han observado estos largos enredos de péptidos y proteínas agregados en enfermedades como el Alzheimer y el Parkinson, pero existe un creciente cuerpo de evidencia que sugiere que estos enredos ocurren después del hecho, que lo que realmente está causando el problema son los ensamblajes de proteínas más pequeños. Averiguar qué son esos ensamblajes y qué tan grandes son actualmente es realmente difícil de hacer, por lo que esta puede ser una forma de resolver ese problema ".