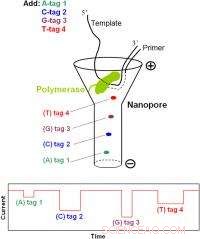
Esquema de secuenciación de ADN de una sola molécula mediante un nanoporo con nucleótidos marcados con fosfato. Cada uno de los cuatro nucleótidos llevará una etiqueta diferente. Durante SBS, estas etiquetas, unido a través del fosfato terminal del nucleótido, se liberarán en el nanoporo de uno en uno, donde producirán firmas de bloqueo de corriente únicas para la determinación de la secuencia. Una gran variedad de estos nanoporos conducirá a una secuenciación de ADN de alto rendimiento.
(Phys.org) —La secuenciación del ADN es la fuerza impulsora detrás de los descubrimientos clave en medicina y biología. Por ejemplo, la secuencia completa del genoma de un individuo proporciona marcadores y pautas importantes para el diagnóstico médico y el cuidado de la salud. Hasta ahora, el principal obstáculo ha sido el costo y la velocidad de obtener secuencias de ADN de alta precisión. Si bien se han logrado numerosos avances en los últimos 10 años, La mayoría de los instrumentos de secuenciación de alto rendimiento actuales dependen de técnicas ópticas para la detección de los cuatro componentes básicos del ADN:A, C, G y T. Para avanzar aún más en la capacidad de medición, También se ha desarrollado la secuenciación electrónica de ADN de un conjunto de plantillas de ADN.
Recientemente, Se ha demostrado que el ADN se puede enhebrar a través de los poros de las proteínas a nanoescala bajo una corriente eléctrica aplicada para producir señales electrónicas a nivel de una sola molécula. Sin embargo, porque los cuatro nucleótidos son muy similares en sus estructuras químicas, no se pueden distinguir fácilmente con esta técnica. Por lo tanto, La investigación y el desarrollo de una plataforma electrónica de secuenciación de ADN de una sola molécula es el área de investigación más activa y tiene el potencial de producir un secuenciador de ADN portátil capaz de descifrar el genoma para la medicina personalizada y la investigación biomédica básica.
Un equipo de investigadores de la Universidad de Columbia, encabezada por el Dr. Jingyue Ju (el profesor de ingeniería Samuel Ruben-Peter G. Viele, Catedrático de Ingeniería Química y Farmacología, Director del Centro de Tecnología Genómica e Ingeniería Biomolecular), con colegas del Instituto Nacional de Estándares y Tecnología (NIST) dirigido por el Dr. John Kasianowicz (Miembro de la Sociedad Estadounidense de Física), han desarrollado un enfoque novedoso para secuenciar potencialmente el ADN en nanoporos electrónicamente a nivel de molécula única con resolución de base única. Este trabajo, titulado "Nucleótidos etiquetados con PEG y detección de nanoporos para secuenciación de ADN de una sola molécula por síntesis" ya está disponible en la revista en línea de acceso abierto, Informes científicos , del grupo Nature Publication.
La estrategia informada de secuenciación por síntesis basada en nanoporos (Nano-SBS) puede distinguir con precisión cuatro bases de ADN mediante la detección de 4 etiquetas de diferentes tamaños liberadas de nucleótidos modificados con 5'-fosfato a nivel de molécula única para la determinación de la secuencia. El principio básico de la estrategia Nano-SBS se describe a continuación. A medida que cada análogo de nucleótido se incorpora a la cadena de ADN en crecimiento durante la reacción de la polimerasa, su etiqueta se libera mediante la formación de enlaces fosfodiéster. Las etiquetas entrarán en un nanoporo en el orden de su liberación, Produciendo firmas de bloqueo de corriente iónica únicas debido a sus distintas estructuras químicas, determinando así la secuencia de ADN electrónicamente a nivel de molécula única con resolución de base única. Como prueba de principio, el equipo de investigación adjuntó cuatro etiquetas poliméricas de diferentes longitudes al fosfato terminal de 2'-desoxiguanosina-5'-tetrafosfato (un bloque de construcción de ADN modificado) y demostró una incorporación eficiente de los análogos de nucleótidos durante la reacción de la polimerasa, así como mejor discriminación que la línea de base entre las cuatro etiquetas a nivel de molécula única en función de sus firmas de bloqueo de corriente iónica nanoporo. Este enfoque, junto con la polimerasa unida a los nanoporos en un formato de matriz, debería producir una plataforma electrónica Nano-SBS de una sola molécula.
En trabajos anteriores, el Centro de Tecnología Genómica e Ingeniería Biomolecular de la Universidad de Columbia, dirigido por el profesor Ju y el Dr. Nicholas J. Turro (profesor de química William P. Schweitzer), desarrolló una plataforma de secuenciación de ADN por síntesis (SBS) de cuatro colores utilizando terminadores reversibles de nucleótidos fluorescentes escindibles (NRT), que tiene licencia para Intelligent Bio-Systems, C ª., una empresa QIAGEN. SBS con NRT fluorescentes escindibles es el enfoque dominante utilizado en los sistemas de secuenciación de ADN de próxima generación. El Dr. Kasianowicz y su grupo en NIST fueron pioneros en la investigación de nanoporos para el análisis de moléculas individuales. Anteriormente informaron que los polímeros de diferentes longitudes, polietilenglicoles (PEG), podrían distinguirse por sus efectos únicos sobre las lecturas actuales en nanoporos de una proteína α-hemolisina a nivel de molécula única y posteriormente desarrollaron una teoría para el método. Sus resultados proporcionan la prueba de concepto para la espectrometría de masas de una sola molécula. La combinación del concepto SBS con las distintas etiquetas electrónicas detectables por nanoporos para etiquetar los bloques de construcción del ADN llevó al desarrollo del enfoque Nano-SBS electrónico de una sola molécula que se describe en la actualidad. Informes científicos artículo.
Como señala el autor principal, el Dr. Shiv Kumar, "La novedad de nuestro enfoque radica en el diseño y uso de cuatro nucleótidos etiquetados de manera diferente, que tras la incorporación por la ADN polimerasa, liberan cuatro etiquetas de diferentes tamaños que se distinguen entre sí en el nivel de una sola molécula cuando pasan a través del nanoporo. Este enfoque supera las limitaciones impuestas por las pequeñas diferencias entre los cuatro nucleótidos, un desafío al que se han enfrentado la mayoría de los métodos de secuenciación de nanoporos durante décadas ". la técnica es bastante flexible; con etiquetas PEG como prototipos, Se pueden elegir otras etiquetas químicas para proporcionar una separación óptima en diferentes sistemas de nanoporos.
Con un mayor desarrollo de este enfoque Nano-SBS, como el uso de grandes conjuntos de proteínas o nanoporos sólidos, este sistema tiene el potencial de secuenciar con precisión un genoma humano completo rápidamente y a bajo costo, lo que permite su uso en diagnósticos médicos de rutina.