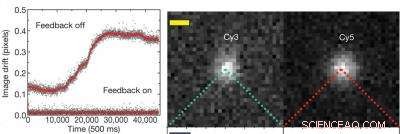
El gráfico de la izquierda muestra que con el sistema de retroalimentación activo desactivado hay una desviación de resolución de aproximadamente 0,3 píxeles o 19 nanómetros, pero con el sistema de retroalimentación sobre la resolución se mantiene mejor que 0.01 píxeles, o aproximadamente 0,64 nanómetros. La imagen de la derecha muestra moléculas de colorante fluorescente de cianina (Cy) individuales, Cy3 y Cy5, que se utilizan para marcar 20 pares de bases de ADN de doble hebra. Crédito:Cortesía del grupo de investigación Steven Chu
La sabiduría convencional sostiene que la microscopía óptica no se puede utilizar para "ver" algo tan pequeño como una molécula individual. Pero como es costumbre, la ciencia inteligente ha vuelto a anular la sabiduría convencional. Secretario de Energía, El premio Nobel y ex director del Laboratorio Nacional Lawrence Berkeley (Berkeley Lab) Steven Chu dirigió el desarrollo de una técnica que permite el uso de microscopía óptica para obtener imágenes de objetos o la distancia entre ellos con resoluciones tan pequeñas como 0,5 nanómetros, la mitad de una milmillonésima parte de un metro, o un orden de magnitud menor que el mejor anterior.
"La capacidad de obtener una resolución subnanométrica en entornos acuosos biológicamente relevantes tiene el potencial de revolucionar la biología, particularmente biología estructural, "dice el secretario Chu." Una de las motivaciones de este trabajo, por ejemplo, era medir distancias entre proteínas que forman dominios múltiples, estructuras muy complejas, como el ensamblaje de proteínas que forma el sistema de ARN polimerasa II humana, que inicia la transcripción del ADN ".
El secretario Chu es coautor de un artículo que ahora aparece en la revista. Naturaleza que describe esta investigación. El artículo se titula "Localización subnanométrica de una sola molécula, registro y mediciones de distancia ". Los otros autores son Alexandros Pertsinidis, investigador postdoctoral y miembro del grupo de investigación de Chu en la Universidad de California (UC) Berkeley, que ahora es profesor asistente en el Sloan-Kettering Institute, y Yunxiang Zhang, miembro del grupo de investigación de Chu en la Universidad de Stanford.
Según una ley de la física conocida como "límite de difracción, "la imagen más pequeña que puede resolver un sistema óptico es aproximadamente la mitad de la longitud de onda de la luz utilizada para producir esa imagen. Para la óptica convencional, esto corresponde a unos 200 nanómetros. En comparación, una molécula de ADN mide aproximadamente 2,5 nanómetros de ancho.
Mientras que los sistemas de imágenes no ópticas, como microscopios electrónicos, puede resolver objetos en la escala subnanométrica, estos sistemas operan en condiciones no ideales para el estudio de muestras biológicas. Detectar etiquetas fluorescentes individuales adheridas a moléculas biológicas de interés mediante dispositivos de carga acoplada (CCD):matrices de chips de silicio que convierten la luz entrante en una carga eléctrica. ha producido resoluciones de hasta cinco nanómetros. Sin embargo, hasta ahora, esta tecnología no ha sido capaz de obtener imágenes de moléculas individuales o distancias entre un par de moléculas mucho menos de 20 nanómetros.
Chu y sus coautores pudieron utilizar la misma tecnología de fluorescencia CCD para resolver distancias con precisión subnanométrica y exactitud al corregir un truco de la luz. Las cargas eléctricas en una matriz CCD se crean cuando los fotones golpean el silicio y desprenden electrones, siendo la fuerza de la carga proporcional a la intensidad de los fotones incidentes. Sin embargo, dependiendo precisamente de dónde un fotón golpea la superficie de un chip de silicio, puede haber una ligera diferencia en cómo se absorbe el fotón y si genera una carga medible. Esta falta de uniformidad en la respuesta de la matriz de silicio CCD a los fotones entrantes, que probablemente sea un artefacto del proceso de fabricación de chips, da como resultado una imagen borrosa de los píxeles que dificulta la resolución de dos puntos que se encuentran a unos pocos nanómetros uno del otro.
"Hemos desarrollado un sistema de retroalimentación activa que nos permite colocar la imagen de una sola molécula fluorescente en cualquier lugar de la matriz CCD con una precisión de subpíxeles, lo que a su vez nos permite trabajar en una región más pequeña que la típica escala de longitud de tres píxeles de la no uniformidad del CCD, "dice Pertsinidis, quien es el autor principal del artículo de Nature. "Con este sistema de retroalimentación más el uso de haces ópticos adicionales para estabilizar el sistema del microscopio, podemos crear una región calibrada en la matriz de silicio donde el error debido a la falta de uniformidad se reduce a 0,5 nanómetros. Al colocar las moléculas que queremos medir en el centro de esta región podemos obtener una resolución subnanométrica utilizando un microscopio óptico convencional que se puede encontrar en cualquier laboratorio de biología ".
Chu dice que la capacidad de mover la platina de un microscopio a distancias pequeñas y calcular el centro geométrico (centroide) de la imagen hace posible no solo medir la no uniformidad de la foto-respuesta entre píxeles, sino también para medir la falta de uniformidad dentro de cada píxel individual.
"Conocer esta falta de uniformidad nos permite hacer correcciones entre la posición aparente y la posición real del centroide de la imagen, "dice Chu." Dado que esta respuesta no uniforme está integrada en la matriz CCD y no cambia de un día a otro, nuestro sistema de retroalimentación activa nos permite tomar imágenes repetidamente en la misma posición de la matriz CCD ".
Pertsinidis continúa trabajando con Chu y otros en el grupo en el desarrollo y aplicación de esta técnica de superresolución. Además del sistema de ARN polimerasa II humana, él y el grupo lo están utilizando para determinar la estructura de las moléculas de cadherina epitelial que son responsables de la adhesión de célula a célula que mantiene unidos los tejidos y otros materiales biológicos. Pertsinidis, Zhang, y otro postdoctorado en el grupo de investigación de Chu, Parque Sang Ryul, también están utilizando esta técnica para crear mediciones en 3D de la organización molecular dentro de las células cerebrales.
"La idea es determinar la estructura y dinámica del proceso de fusión de vesículas que libera las moléculas de neurotransmisores que utilizan las neuronas para comunicarse entre sí," ", Dice Pertsinidis." En este momento estamos obteniendo mediciones in situ con una resolución de unos 10 nanómetros, pero creemos que podemos llevar esta resolución a dos nanómetros ".
En colaboración con Joe Gray, Director Asociado de Ciencias de la Vida de Berkeley Lab y un destacado investigador del cáncer, Los postdoctorados en el grupo de investigación de Chu también están utilizando la técnica de superresolución para estudiar la unión de moléculas de señalización en la proteína RAS, que se ha relacionado con varios cánceres, incluidos los del pecho, páncreas, pulmón y colon. Esta investigación podría ayudar a explicar por qué las terapias contra el cáncer que funcionan bien en algunos pacientes son ineficaces en otros.
Además de sus aplicaciones biológicas, Pertsinidis, Zhang y Chu en su Naturaleza El documento dice que su técnica de superresolución también debería resultar valiosa para caracterizar y diseñar sistemas de imágenes fotométricas de precisión en física atómica o astronomía. y permitir nuevas herramientas en litografía óptica y nanometrología.