Las proteínas intrínsecamente desordenadas (IDP) se encuentran ampliamente en los proteomas de los eucariotas y desempeñan funciones clave en procesos vitales como la transcripción de información genética y la señalización. Además de ser habitualmente muy repetitivos, hidrófilos y cargados eléctricamente, así como de codificar secuencias simples de genes, los desplazados internos también se distinguen por su abundancia natural y sus aspectos estructurales, que se convierten en la base del "paradigma de función desordenada" de las proteínas.
Durante las últimas dos décadas, se ha estudiado activamente el papel de los desplazados internos en las enfermedades humanas y como objetivos farmacológicos, mientras que cómo caracterizar las conformaciones altamente flexibles y heterogéneas de los desplazados internos en alta resolución sigue siendo una cuestión clave en este campo.
Más del 15% de las moléculas de IDP están unidas a membranas en las células, y su dinámica interna y movimientos generales (de traslación y rotación) dentro de las bicapas de fosfolípidos están estrechamente relacionados con sus propiedades fisicoquímicas y funciones biológicas, pero estos procesos dinámicos son difíciles de capturar y caracterizado cuantitativamente por métodos convencionales de análisis estructural.
Un grupo de investigadores dirigido por el Prof. Long Dong de la Universidad de Ciencia y Tecnología de China (USTC) ha desarrollado un método de espectroscopia IDP basado en la técnica de mejora de la relajación paramagnética de membrana (mPRE), que ha logrado con éxito un modelado de alta precisión de la conformaciones internas, orientaciones y profundidades de inmersión de los desplazados internos. Los resultados han sido publicados en el Journal of the American Chemical Society .
En este trabajo, los investigadores exploraron en detalle la flexibilidad y movilidad de las moléculas de la sonda de espín dentro de la membrana para una interpretación precisa de los datos espectrales mPRE, y propusieron un modelo de cuadrícula tridimensional (3D) ponderado basado en simulaciones de todos los átomos para cuantificar que representa el efecto de la dinámica de la sonda de espín en la tasa de mejora de la relajación paramagnética de la membrana.
Aprovechando la alta eficiencia computacional del modelo, los investigadores desarrollaron un algoritmo que se detalla en la Información de respaldo (SI) para optimizar las orientaciones de los grados de libertad de movimiento global e interno de los desplazados internos unidos a membranas mediante la superposición de z- solo coordenadas, diseñado para el análisis de datos mPRE, construyendo un modelo de conjunto de átomos de IDP en un entorno de membrana implícito.
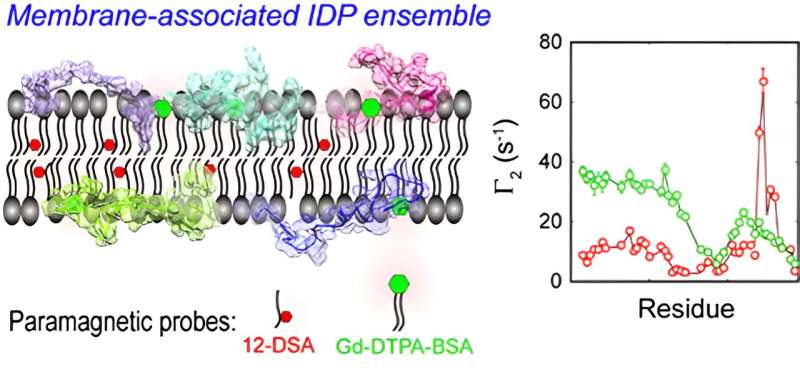
CD3ε es un componente del complejo del receptor de células T (TCR) responsable del reconocimiento del antígeno de las células T. El CD3ε dominio citoplasmático (CD3εCD ) contiene motivos de activación basados en tirosina (ITAM) de inmunorreceptores y forma un complejo difuso con bicapas lipídicas en un estado intrínsecamente desordenado, y regula la actividad de señalización del complejo difuso mediante el uso de protección de membrana dinámica de sitios clave de tirosina.
Los investigadores resolvieron el conjunto basándose en la dinámica molecular de CD3εCD en bicapas lipídicas mediante la aplicación de una solución modelo de conjunto de átomos de IDP en un entorno de membrana implícito.
El conjunto de parámetros experimentales mPRE mapea la distribución dinámica de CD3εCD en diferentes regiones de la membrana a nivel atómico y revela diferencias clave en las interacciones de membrana de diferentes sitios de tirosina en ITAM, proporcionando una nueva explicación mecanicista para el patrón de monofosforilación de ITAM.
Se espera que el método de análisis espectroscópico mPRE establecido en este trabajo facilite ampliamente los estudios de resolución atómica de varios IDP de membrana funcionales.
Más información: Hong Jin et al, Interpretación cuantitativa conjunta de la mejora de la relajación paramagnética de la membrana (mPRE) para estudiar proteínas intrínsecamente desordenadas asociadas a la membrana, Revista de la Sociedad Química Estadounidense (2023). DOI:10.1021/jacs.3c10847
Información de la revista: Revista de la Sociedad Química Estadounidense
Proporcionado por la Universidad de Ciencia y Tecnología de China