Alcanzar objetivos incrustados dentro de la membrana celular ha sido durante mucho tiempo difícil para los desarrolladores de fármacos debido a las desafiantes propiedades bioquímicas de la membrana. Ahora, los químicos de Scripps Research han demostrado nuevas proteínas diseñadas a medida que pueden alcanzar eficientemente estos objetivos "intramembranosos".
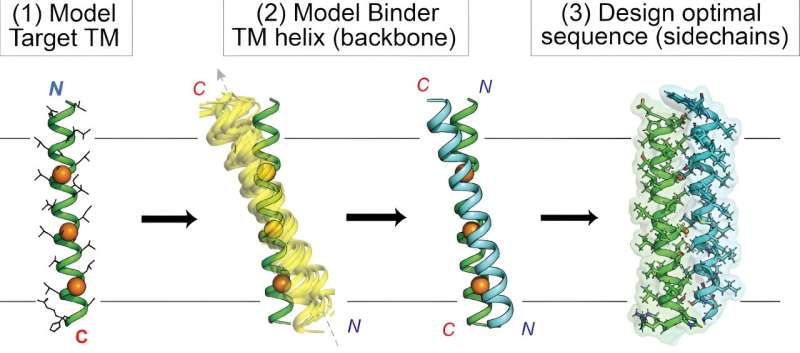
En su estudio, "Proteínas transmembrana de novo diseñadas para unirse e inhibir un receptor de citoquinas" publicado en Nature Chemical Biology , los investigadores utilizaron un enfoque informático único para diseñar nuevas proteínas dirigidas a la región que atraviesa la membrana del receptor de eritropoyetina (EPO), que controla la producción de glóbulos rojos y puede fallar en los cánceres. Además de estas nuevas moléculas dirigidas a EPO, el estudio arrojó un proceso computacional general, o "flujo de trabajo", para agilizar el diseño personalizado flexible de proteínas dirigidas a objetivos intramembrana.
Los investigadores ahora están utilizando su enfoque para desarrollar nuevos tratamientos potenciales dirigidos a la intramembrana en una amplia gama de enfermedades.
"Este trabajo abre muchas posibilidades nuevas para la modulación de objetivos dentro de la membrana celular, incluso para aplicaciones terapéuticas y la comprensión de los mecanismos de señalización en la biología celular", dice el coautor del estudio, Marco Mravic, Ph.D., profesor asistente en la Departamento de Biología Computacional y Estructural Integrativa de Scripps Research.
Los otros coautores correspondientes del estudio fueron Daniel DiMaio, MD, Ph.D., de la Facultad de Medicina de Yale; y William DeGrado, Ph.D., de la Facultad de Farmacia de San Francisco de la Universidad de California, donde Mravic anteriormente obtuvo un doctorado. estudiante.
"Un objetivo importante de la biología sintética es diseñar proteínas con actividad biológica; aquí informamos el diseño y las pruebas de una pequeña proteína que perturba específicamente la actividad de un receptor proteico mucho más grande involucrado en el crecimiento y la diferenciación de las células sanguíneas", dice DiMaio, quien es profesor de Genética y de Biofísica y Bioquímica Molecular, y subdirector del Yale Cancer Center.
"Logramos esto apuntando al segmento del receptor que cruza la membrana celular. Debido a que muchas proteínas celulares contienen segmentos que atraviesan la membrana estructuralmente conservados, este enfoque general puede ser aplicable a muchos otros objetivos proteicos y proporciona una nueva herramienta para modular el comportamiento de células."
Alcanzar objetivos intramembrana ha sido durante mucho tiempo un objetivo importante en biomedicina, ya que muchas proteínas de las células (especialmente las proteínas receptoras) tienen dominios funcionalmente importantes dentro de la membrana. Estas proteínas desempeñan funciones destacadas en casi todos los ámbitos de la salud y la enfermedad.
Sin embargo, los objetivos intramembrana no son objetivos ordinarios. Las membranas celulares generalmente están formadas por dos capas de moléculas de "lípidos" relacionadas con las grasas, estrechamente espaciadas, que repelen el agua y tienen otras propiedades únicas y complejas. Esto hace que el espacio intramembrana sea un objetivo mucho más difícil para los diseñadores de fármacos, en comparación con las zonas acuosas de las superficies o interiores de las células.
"Ha habido muy pocos ejemplos exitosos de medicamentos dirigidos a este espacio dentro de la membrana", dice Mravic.
Esos pocos éxitos, que incluyen tratamientos para trastornos de plaquetas bajas y fibrosis quística, provienen de exámenes ciegos de grandes bibliotecas de compuestos o de una estrecha imitación de proteínas que se sabe que interactúan con proteínas asociadas dentro de la membrana celular.
Por el contrario, Mravic y sus colegas se propusieron diseñar proteínas completamente nuevas (pequeñas llamadas péptidos) para alcanzar objetivos proteicos intramembrana de formas nuevas y diversas. Para hacerlo, tuvieron que ampliar la frontera de los métodos computacionales, combinando la "extracción de datos" de interacciones conocidas entre proteínas en membranas con predicciones tradicionales basadas en la física sobre interacciones entre proteínas.
Finalmente, Mravic y sus colegas diseñaron las primeras proteínas que se unen a la porción que atraviesa la membrana del receptor de EPO de una manera nueva, no vista en la naturaleza. El equipo demostró que estas proteínas bloquean de forma muy específica y potente la función del receptor, a diferencia de enfoques anteriores.
Los resultados pueden ser de interés inmediato para los investigadores que buscan nuevas formas de inhibir el receptor de EPO, que a menudo es activado de forma anormal por las células tumorales para mantener su crecimiento y supervivencia. Pero para Mravic y sus colegas, el estudio representa sobre todo una prueba de principio de un enfoque nuevo y más flexible para la focalización intramembrana.
"Tenemos la intención de utilizar este enfoque para atacar las proteínas de membrana en múltiples procesos biológicos y áreas de enfermedades, incluidos cánceres, trastornos inmunológicos y dolor", afirma Mravic.
También espera que el flujo de trabajo computacional que diseñó para el proyecto sea un acelerador general del diseño de fármacos dirigidos a membranas.
"Antes, el proceso básicamente implicaba que dos personas en una habitación oscura miraran la pantalla de una computadora y dijeran:'Sí, creo que esto se ve mejor que aquello'", dice Mravic. "Ahora hemos automatizado gran parte del proceso de diseño de moléculas y la toma de decisiones en la computadora. Ser más modular, flexible y optimizado permite que el método sea más accesible para una gama más amplia de científicos".
Mravic y sus colegas han publicado sus herramientas computacionales para uso público en Github.
Más información: Proteínas transmembrana de novo diseñadas para unirse e inhibir un receptor de citoquinas, Biología química natural (2024). DOI:10.1038/s41589-024-01562-z, www.nature.com/articles/s41589-024-01562-z
Proporcionado por el Instituto de Investigación Scripps