Las simulaciones de dinámica molecular (MD) se han convertido en una herramienta poderosa en los campos en constante crecimiento de la biología molecular y el desarrollo de fármacos. Si bien existen muchas técnicas de simulación de MD, la MD de selección en cascada paralela (PaCS-MD) es particularmente útil cuando se estudia el plegamiento (o "conformación") de proteínas o las interacciones entre proteínas y ligandos.
El quid de este método consiste en ejecutar múltiples simulaciones MD en paralelo, explorando así diferentes conformaciones posibles simultáneamente. Utilizando criterios de selección cuidadosamente elaborados, las conformaciones prometedoras pueden detectarse automáticamente en "instantáneas temporales" e investigarse más a fondo. Esta estrategia acelera enormemente el descubrimiento de interacciones moleculares clave y procesos dinámicos que ayudan a los científicos a comprender los movimientos funcionales de las proteínas.
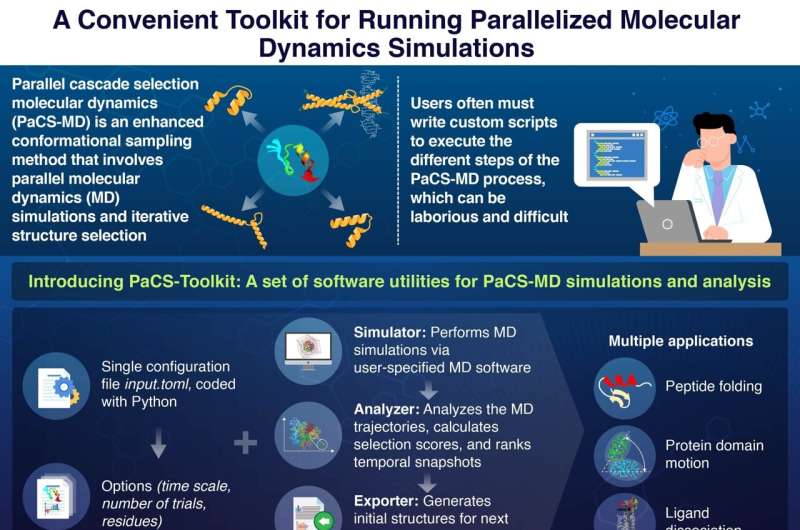
Sin embargo, uno de los obstáculos de PaCS-MD es que los usuarios deben escribir scripts personalizados para ejecutar las simulaciones MD que deseen. En estos scripts, deben especificar las condiciones iniciales y las características objetivo, seleccionar el software MD a utilizar, implementar un procedimiento de clasificación instantánea y preparar las estructuras iniciales para el siguiente ciclo de simulación. Este proceso, que puede ser bastante complejo y propenso a errores, crea una barrera de entrada considerable para los científicos interesados en utilizar PaCS-MD.
Afortunadamente, un equipo de investigadores de la Escuela de Ciencias de la Vida y Tecnología del Instituto de Tecnología de Tokio (Japón) se propuso recientemente abordar este problema. En su último estudio, publicado en The Journal of Physical Chemistry B y dirigido por el profesor Akio Kitao, el equipo desarrolló un paquete de software llamado PaCS-Toolkit para hacer que PaCS-MD sea más accesible y cómodo de usar.
Una ventaja notable de PaCS-Toolkit es que todo el proceso de simulación se configura a través de un único archivo de configuración. En este archivo, los usuarios especifican parámetros importantes para las simulaciones, incluido el tipo de PaCS-MD, el número de simulaciones de MD que se ejecutarán en paralelo y los residuos o átomos de proteínas para realizar un seguimiento como criterio de selección para ramas paralelas. P>
PaCS-Toolkit toma este archivo de configuración, junto con los archivos de entrada MD estándar, y ejecuta simulaciones PaCS-MD de acuerdo con el software MD especificado. Vale la pena señalar que, dado que el paquete es un software abierto escrito en Python, un lenguaje de programación popular, los usuarios pueden ayudar a mejorar PaCS-Toolkit y ampliar sus funcionalidades.
"Nuestro conjunto de herramientas mantiene la flexibilidad para que se puedan agregar nuevas características, bibliotecas y software MD mediante la introducción de clases responsables en Python. Los usuarios que pueden programar en este lenguaje deberían poder modificar el código de PaCS-Toolkit e implementar nuevos métodos según sea necesario. " dice el profesor Kitao.
Otra ventaja crucial de PaCS-Toolkit radica en su optimización y compatibilidad con diferentes entornos informáticos, ya sea una serie de supercomputadoras con una interfaz de paso de mensajes (MPI), servidores equipados con múltiples tarjetas gráficas (GPU) o computadoras personales como computadoras portátiles.
"PaCS-Toolkit incorpora en gran medida la paralelización mediante MPI, GPU y el paquete multiprocesamiento de Python, lo que permite optimizar el tiempo de cálculo en función de los recursos computacionales disponibles", explica el profesor Kitao.
Para mostrar el potencial de su conjunto de herramientas, los investigadores realizaron simulaciones de PaCS-MD adaptadas a tres aplicaciones diferentes. Estos incluyeron el plegamiento de la miniproteína chignolina, el movimiento de dominios proteicos en una enzima del SARS-CoV-2 y la disociación del ligando de un importante receptor de adenosina.
"En conjunto, nuestros resultados indican que PaCS-Toolkit se puede utilizar fácilmente para simular una amplia variedad de dinámicas para diferentes tipos de sistemas moleculares", concluye el profesor Kitao.
Este conjunto de herramientas podría ayudar a desbloquear el verdadero potencial de las simulaciones de PaCS-MD en diversos campos, permitiendo a los investigadores interesados arrojar luz sobre procesos moleculares complejos y acelerar el descubrimiento de fármacos.
Más información: Shinji Ikizawa et al, PaCS-Toolkit:Utilidades de software optimizadas para simulaciones de dinámica molecular de selección en cascada paralela (PaCS-MD) y análisis posteriores, The Journal of Physical Chemistry B (2024). DOI:10.1021/acs.jpcb.4c01271
Información de la revista: Revista de Química Física B
Proporcionado por el Instituto de Tecnología de Tokio