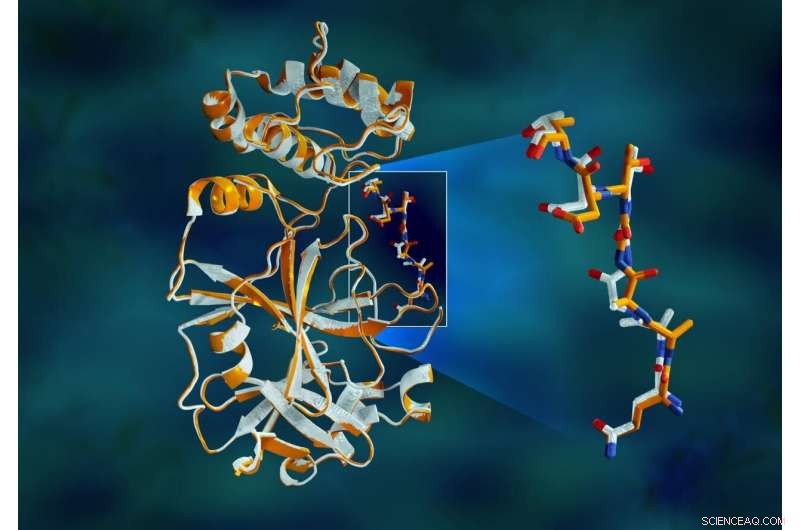
La superposición de datos de rayos X de la proteasa principal del SARS-CoV-2 muestra diferencias estructurales entre la proteína a temperatura ambiente (naranja) y la estructura congelada criogénicamente (blanco). Crédito:Jill Hemman / ORNL, Departamento de Energía de EE. UU.
Un equipo de investigadores de los laboratorios nacionales de Oak Ridge y Argonne del Departamento de Energía ha realizado las primeras mediciones de rayos X a temperatura ambiente en la proteasa principal SARS-CoV-2, la enzima que permite que el virus se reproduzca.
Las mediciones de rayos X marcan un primer paso importante en el objetivo final de los investigadores de construir un modelo 3D integral de la proteína enzimática. El modelo se utilizará para avanzar en las simulaciones de supercomputación destinadas a encontrar inhibidores de fármacos para bloquear el mecanismo de replicación del virus y ayudar a poner fin a la pandemia de COVID-19. Los resultados de su investigación están a disposición del público y se han publicado en la revista Comunicaciones de la naturaleza .
El SARS-CoV-2 es el virus que causa la enfermedad COVID-19. El virus se reproduce expresando largas cadenas de proteínas que la enzima proteasa debe cortar en trozos más pequeños.
"La proteasa es indispensable para el ciclo de vida del virus. La proteína tiene la forma de un corazón de San Valentín, pero realmente es el corazón del virus lo que le permite replicarse y propagarse. Si inhibe la proteasa y detiene el corazón, el virus no puede producir las proteínas que son esenciales para su replicación. Es por eso que la proteasa se considera un objetivo farmacológico tan importante, "dijo Andrey Kovalevsky de ORNL, Autor correspondiente. Si bien la estructura se conoce a partir de cristales conservados criogénicamente, "Esta es la primera vez que se mide la estructura de esta enzima a temperatura ambiente, lo cual es significativo porque está cerca de la temperatura fisiológica donde operan las células ".
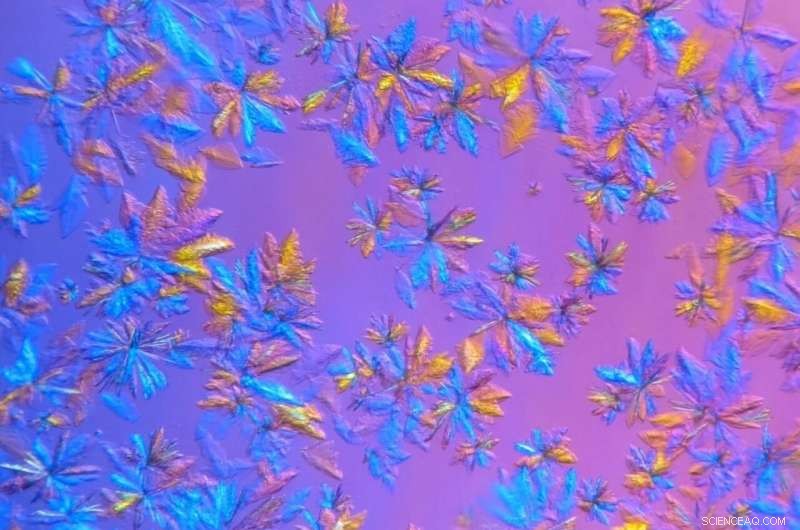
Cristales de proteasa SARS-CoV-2, cultivado en el laboratorio de cristalización y caracterización de proteínas de ORNL y representado en una vista microscópica, se utilizará en experimentos de dispersión de rayos X. Crédito:Daniel Kneller / ORNL, Departamento de Energía de EE. UU.
La construcción de un modelo completo de la estructura de la proteína requiere identificar cada elemento dentro de la estructura y cómo están organizados. Los rayos X son ideales para detectar elementos pesados como el carbono, átomos de nitrógeno y oxígeno. Debido a la intensidad de los haces de rayos X en la mayoría de las instalaciones de sincrotrón a gran escala, Por lo general, las muestras biológicas deben congelarse criogénicamente a unos 100 K, o aproximadamente menos 280 grados Fahrenheit, para resistir la radiación el tiempo suficiente para que se recopilen los datos.
Para extender la vida útil de las muestras de proteína cristalizada y medirlas a temperatura ambiente, Los investigadores de ORNL cultivaron cristales más grandes de lo necesario para los crioestudios de sincrotrón y utilizaron una máquina de rayos X interna que presenta un haz menos intenso.
"El cultivo de cristales de proteínas y la recopilación de datos es un proceso tedioso y que requiere mucho tiempo. En el tiempo que suele llevar preparar y enviar la muestra a un sincrotrón, pudimos hacer crecer los cristales, tomar las medidas y comenzar a analizar los datos, "dijo Daniel Kneller de ORNL, el primer autor del estudio. "Y, cuando hay una pandemia con muchos científicos movilizándose para estudiar este problema, no hay un día libre ".
La enzima proteasa consta de cadenas de aminoácidos con un patrón repetido de átomos de nitrógeno-carbono-carbono que forman la columna vertebral de la proteína. Grupos laterales de los bloques de construcción de aminoácidos, o "residuos, "se extienden desde cada uno de los átomos de carbono de la columna vertebral central. La enzima se pliega en una forma tridimensional específica, creando bolsas especiales donde se adheriría una molécula de fármaco.
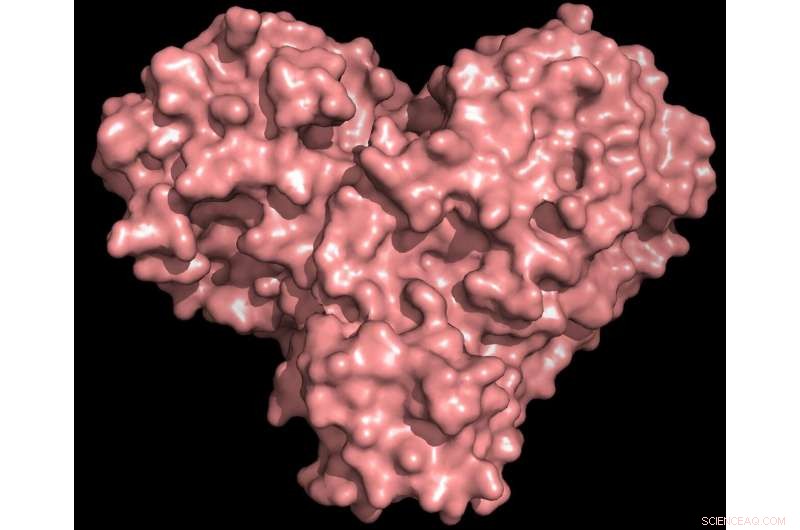
La proteína proteasa tiene la forma de un corazón y funciona como uno solo, permitiendo que el virus se replique y se propague. La inhibición de la proteasa bloquearía la reproducción del virus. Crédito:Crédito:Andrey Kovalevsky / ORNL, Departamento de Energía de EE. UU.
El estudio reveló disparidades estructurales significativas entre las orientaciones de la columna vertebral y algunos de los residuos en las muestras criogénicas y a temperatura ambiente. La investigación sugiere que congelar los cristales puede introducir artefactos estructurales que podrían resultar en una comprensión menos precisa de la estructura de la proteasa.
Los resultados del equipo se están compartiendo con los investigadores, dirigido por Jeremy Smith, presidente del gobernador de la Universidad de Tennessee ORNL, que están realizando simulaciones de acoplamiento de drogas utilizando Summit en ORNL, la supercomputadora más rápida del país.
"Lo que los investigadores están haciendo en Summit es tomar compuestos de fármacos conocidos y tratar de unirlos computacionalmente a la proteasa principal para la reutilización de fármacos, además de buscar nuevas pistas sobre otros posibles candidatos a fármacos, ", dijo el autor correspondiente de ORNL, Leighton Coates." Nuestros datos de temperatura ambiente se están utilizando para construir un modelo más preciso para esas simulaciones y mejorar las actividades de diseño de fármacos ".
El siguiente paso de los investigadores para completar el modelo 3-D de la proteasa principal SARS-CoV-2 es utilizar la dispersión de neutrones en el reactor de isótopos de alto flujo de ORNL y la fuente de neutrones de espalación. Los neutrones son esenciales para localizar los átomos de hidrógeno, que desempeñan un papel fundamental en muchas de las funciones catalíticas y los esfuerzos de diseño de fármacos.
El ADN del plásmido de proteasa utilizado para producir la enzima fue proporcionado por el Centro de Biología Estructural de Argonne en Advanced Photon Source. La cristalización de las proteínas utilizadas en los experimentos de dispersión de rayos X se realizó en el Centro de Biología Estructural y Molecular de ORNL.