
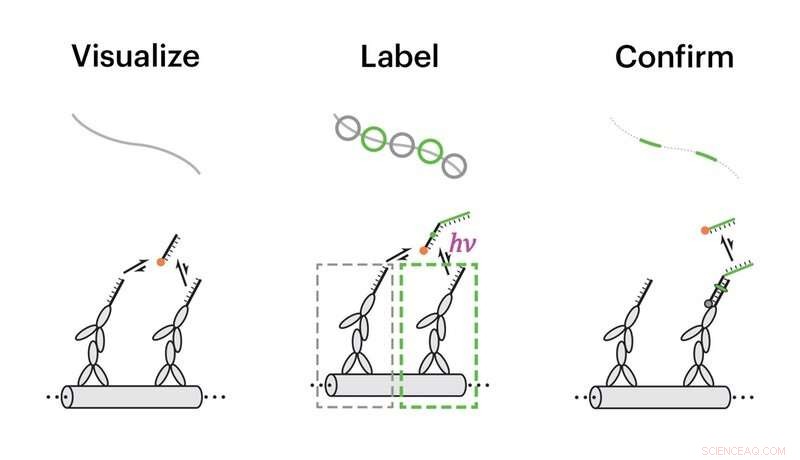
El equipo demostró las capacidades de Action-PAINT en nanoestructuras de ADN sintético que exponen sitios de acoplamiento cercanos idénticos para cadenas de imágenes. En un primer paso, los sitios de acoplamiento se visualizaron mediante microscopía de superresolución (izquierda), luego, con la ayuda de un paquete de software especial, Los sitios de acoplamiento individuales seleccionados manualmente se etiquetaron reticulando las hebras del generador de imágenes con radiación UV (centro), y finalmente, Los eventos de etiquetado exitosos se verificaron mediante una ronda adicional de microscopía de superresolución (derecha). Crédito:Instituto Wyss de la Universidad de Harvard
Para comprender cómo las moléculas individuales desempeñan su papel en los procesos biológicos dentro de las células en las que se sintetizan, Los investigadores han desarrollado métodos de microscopía de superresolución para visualizarlos a nivel de una sola molécula. Sin embargo, para investigar sus funciones, por último, también les gustaría poder modificarlos individualmente a esta alta resolución. Si bien la visualización de moléculas individuales ha avanzado mucho en los últimos años, Hasta ahora ha sido un desafío modificarlos directamente de forma controlada, moda molécula a molécula.
Ahora, reportado en Química de la naturaleza , investigadores del Instituto Wyss de Harvard para la Ingeniería de Inspiración Biológica y la Escuela de Medicina de Harvard (HMS), han desarrollado "Action-PAINT, "un método que combina el enfoque de microscopía de superresolución DNA-PAINT en tiempo real del equipo con una estrategia de etiquetado de una sola molécula en una ubicación deseada dentro de nanoestructuras sintéticas o células intactas. Este enfoque podría desarrollarse más para permitir a los investigadores estimular o inhibir el funciones de moléculas individuales y estudiar las consecuencias de los procesos biológicos y relacionados con enfermedades normales en tiempo real y en superresolución.
"Los métodos de obtención de imágenes de superresolución nos han permitido 'ver lo que antes era invisible". Al acoplar nuestro método de microscopía de superresolución DNA-PAINT con un enfoque de reticulación, ahora también podemos 'tocar lo que antes era inaccesible' colocando un mango físico en moléculas observables individualmente justo en el momento de la visualización, ", dijo Peng Yin, miembro de la facultad principal del Instituto Wyss, Doctor., quien dirigió el estudio. Yin también es co-líder de la Iniciativa de Robótica Molecular del Instituto, y profesor de Biología de Sistemas en HMS.
El equipo de Yin desarrolló un enfoque de dos pasos que primero visualiza proteínas individuales u otras moléculas en superresolución, y luego transfiere una etiqueta molecular al sitio objetivo deseado. Opcionalmente, los investigadores pueden confirmar transferencias exitosas con una ronda adicional de imágenes de súper resolución.
El primer paso de la obtención de imágenes de una sola molécula se basa en el método DNA-PAINT que permitió al equipo determinar primero la localización precisa de moléculas o características moleculares que están separadas espacialmente por distancias muy por debajo del límite de difracción de la luz. y por lo tanto invisible para la mayoría de los microscopios. Los investigadores primero unieron una "hebra de acoplamiento" corta de ADN al objetivo que sirve como sitio de unión para una "hebra de imagen" complementaria que lleva un tinte fluorescente. Debido a que la hebra del generador de imágenes se une con una tasa de encendido y apagado programable, produce eventos de "parpadeo" definidos que se pueden observar usando microscopios estándar. Para adjuntar una etiqueta física al objetivo, los investigadores llevaron a cabo su primer paso de obtención de imágenes de superresolución con una cadena de generación de imágenes un poco más compleja que también incluía un reticulador fotoinducible, que es capaz de vincular químicamente la hebra de acoplamiento y el generador de imágenes entre sí cuando se expone a la radiación UV, y una secuencia informadora adicional.
Para etiquetar objetivos de proteínas individuales en células intactas con Action-PAINT, Las cadenas de acoplamiento se unen a moléculas de anticuerpos que se unen a proteínas diana con alta especificidad. Todos los eventos de unión de anticuerpos en las proteínas diana en un área definida se visualizan con hebras de imágenes fluorescentes complementarias usando microscopía de superresolución (izquierda), y las proteínas diana en áreas seleccionadas a mano que se muestran como cuadrángulos verdes (centro) están marcadas por eventos de reticulación. Finalmente, Los eventos de entrecruzamiento exitosos se confirman mediante el uso de cadenas de imágenes secundarias e imágenes de superresolución (derecha). Crédito:Instituto Wyss de la Universidad de Harvard
"Un componente crítico de nuestro nuevo método es el control preciso del láser de reticulación, que funciona en sincronía con la secuencia de parpadeo de superresolución. Esto realmente convierte nuestra microscopía de súper resolución DNA-PAINT de un método de imagen pasivo a uno activo, permitiendo la interacción en tiempo real entre el investigador y los objetivos moleculares individuales, "dijo el co-primer y co-autor correspondiente Mingjie Dai.
Para habilitar esta nueva capacidad, el equipo desarrolló un paquete de software que les permitió trazar primero las ubicaciones precisas de todos los objetivos moleculares en un área de interés utilizando el método DNA-PAINT, y luego sincronice estrechamente el parpadeo de un UV con los eventos de parpadeo subsiguientes. "De esta manera, podríamos unir químicamente la hebra del generador de imágenes a objetivos moleculares seleccionados de forma personalizada, uno a uno, con resolución molecular, como un pintor pone su color, parche por parche en el estilo puntillismo, "dijo Dai, Doctor., un ex becario postdoctoral en el equipo de Yin, quien actualmente es miembro del Departamento de Biología de Sistemas y Desarrollo de Tecnología de HMS en el Instituto Wyss.
Para demostrar la efectividad y selectividad de su enfoque, El equipo utilizó por primera vez nanoestructuras de ADN sintético que exponían los sitios de acoplamiento para las hebras del generador de imágenes en patrones definidos. "Poniendo Action-PAINT a trabajar, Comenzamos demostrando su efectividad al etiquetar objetivos de una sola molécula a una distancia de solo 30 a 70 nm de sus vecinos idénticos con altas eficiencias en el objetivo, "dijo el co-primer autor Ninning Liu, Doctor., "y luego validó nuestro método en celdas fijas, donde seleccionamos y etiquetamos proteínas de microtúbulos a lo largo de filamentos citoesqueléticos, con diferentes patrones personalizados ". Liu es un becario postdoctoral en el equipo de Yin.
Los autores prevén que Action-PAINT podría desarrollarse aún más en una herramienta de amplia aplicación que, por ejemplo, podría ayudar a modificar directamente las actividades de los receptores de membrana única en la superficie de las células desde donde dirigen el comportamiento celular, o de canales iónicos que controlan la función de las células neuronales. Además, los métodos podrían permitir la transferencia de asas moleculares a proteínas individuales que pueden permitir su extracción y purificación junto con otras proteínas a las que se unen de forma natural.
"Action-PAINT agrega otro nivel de funcionalidad a las capacidades desarrolladas por el equipo de Peng Yin que pueden permitir tanto el mapeo de interacción de proteínas funcionales basado en la posición molecular dentro de las células individuales, y análisis bioquímico de estas interacciones una vez que estas moléculas están aisladas, que en el futuro podrían ayudar a identificar y / o validar nuevos objetivos farmacológicos, "dijo el director fundador del Instituto Wyss, Donald Ingber, MARYLAND., Doctor.