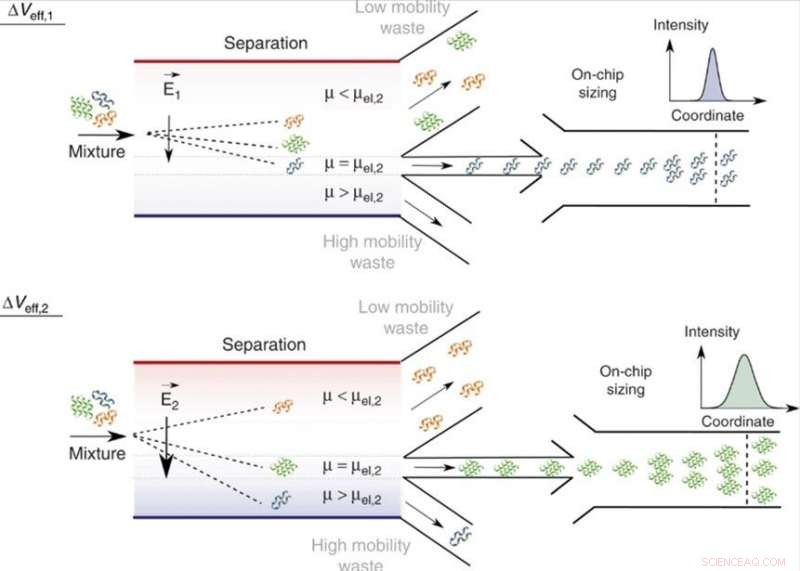
Separación en chip y análisis de mezclas en fase líquida. Ajustando el voltaje efectivo, ΔVeff, moléculas de una movilidad electroforética específica, μ =μel, y por lo tanto de una carga específica a una relación de radio hidrodinámico, qRh, pueden dirigirse al área de análisis con los valores de movilidad menores (μ <μel) y mayores (μ> μel) dirigidos a los canales de recogida de residuos. Como el voltaje aplicado se puede variar, solo se requiere una unidad de análisis y el ancho de la cámara de separación se puede mantener restringido, permitiendo que el dispositivo retenga una eficiencia de alto voltaje y una velocidad de procesamiento rápida. En el área de análisis, las fracciones se dimensionan a través del dimensionamiento por difusión de microfluidos (MDS) controlando su movimiento espacio-temporal en condiciones de flujo laminar. Crédito:Microsistemas y Nanoingeniería, doi:10.1038 / s41378-019-0072-3
Los sistemas de microfluidos se utilizan en biología molecular, bioquímica y biotecnología para analizar rápidamente mezclas biomoleculares heterogéneas con altas tasas de recuperación y volúmenes de muestra diminutos. Sin embargo, Es un desafío combinar procesos preparativos y analíticos en un solo dispositivo para un análisis integrado rápido. En un estudio reciente ahora publicado en Microsistemas y nanoingeniería , Kadi L. Saar y colaboradores de los departamentos interdisciplinarios de química, física, y Fluidic Analytics Limited en Cambridge, REINO UNIDO., han desarrollado un chip que combina los dos pasos de preparación y análisis.
Inicialmente, utilizaron voltaje para separar moléculas de proteína dentro de una mezcla binaria de biomoléculas de igual tamaño, indiscernibles mediante técnicas convencionales de resolución o dimensionamiento. Después de eso, el equipo de investigación utilizó el nuevo dispositivo para obtener una huella digital bidimensional de una mezcla de proteínas heterogénea. Los resultados abrirán nuevas posibilidades para adquirir datos multiparamétricos rápidos en sistemas biomoleculares en una escala de tiempo corta.
Las técnicas de microfluídica son atractivas para analizar muestras biológicas debido a los requisitos de muestra muy bajos y una alta tasa de recuperación. Las plataformas pueden proporcionar una velocidad de análisis insuperable a nivel de unidades de operación individuales o proporcionar varias unidades con un flujo de trabajo combinado directamente. sin transferencia de muestra entre las unidades. Tales transferencias ocurren a través de conectores o tubos e introducen dispersión a la muestra, afectando el rendimiento del sistema. El flujo de trabajo propuesto en este documento puede separar mezclas heterogéneas para determinar los componentes de interés y reducir las complejidades para el procesamiento posterior de la mezcla para su purificación.
Los investigadores habían introducido previamente una variedad de estrategias de separación molecular basadas en flujo continuo a escala micrométrica, incluida la electroforesis de flujo libre, dielectroforesis, magnetoforesis y separación acústoforética. Estrategias de detección como fluorescencia inducida por láser o LED (LIF), Los enfoques de quimioluminiscencia o electroquímicos pueden arraigarse en paralelo dentro de tales plataformas de separación de microfluidos. La información analítica sobre los compuestos separados se puede lograr con estrategias fuera de línea como la espectroscopia de masas o SDS-PAGE, pero las técnicas pueden limitar la velocidad de procesamiento en un solo dispositivo, causando pérdida de muestra o contaminación.
Saar y col. por lo tanto, desarrolló una separación y caracterización cuantitativa totalmente integrada de muestras biomoleculares heterogéneas en un solo dispositivo de microfluidos para superar los límites existentes al acoplar directamente la separación en el chip al análisis en el chip y el tamaño molecular. La característica de diseño permitió el análisis de una fracción específica ajustando la intensidad de campo aplicada. Diseñaron el dispositivo para identificar fracciones separadas similares a los métodos SEC-MALS (cromatografía de exclusión por tamaño con dispersión de luz de múltiples ángulos) o LC (chip) -MS ((en chip) -cromatografía líquida-espectrometría de masas).
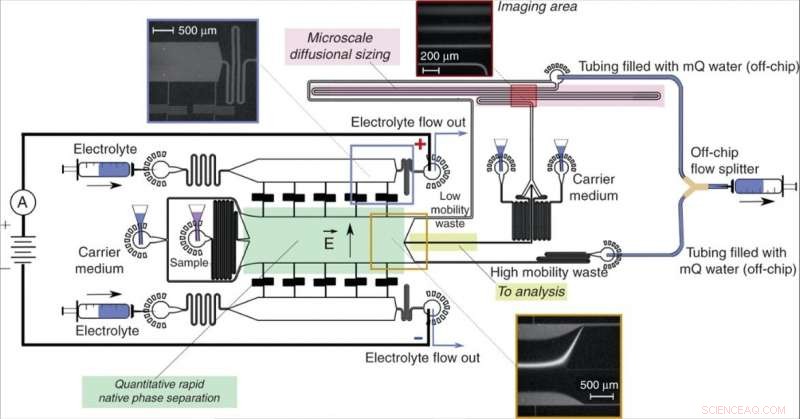
Diseño y operación de dispositivos. Una unidad de electroforesis de flujo libre (verde), permitiendo una rápida separación de las moléculas de analito en su fase nativa y proporcionando información cuantitativa sobre el proceso de separación, Dirigió las fracciones de la muestra (amarillo) a un proceso de análisis posterior que implica el dimensionamiento por difusión de microfluidos (MDS; rosa). La muestra se caracterizó mediante el seguimiento de un único marco de imágenes (recuadro superior derecho) para extraer simultáneamente los tamaños y las cargas de las fracciones separadas. El dispositivo se hizo funcionar aplicando una presión negativa en su salida con un divisor de flujo fuera de chip en forma de Y que mantenía separadas las soluciones de los canales de "desechos de baja movilidad" y "desechos de alta movilidad" para evitar un cortocircuito parcial del dispositivo. El potencial eléctrico se aplicó desde las salidas de la solución de electrolito empleando una solución de electrolito que fluye como se describió anteriormente13. El flujo de la solución de electrolitos, que incluía un trazador fluorescente para su visualización, se eligió de modo que el electrolito llegara a su salida en lugar de ser retirado a la cámara de separación sin llegar a ella (recuadro superior izquierdo), sin embargo, se filtraría en la cámara a una distancia controlada, dejando suficiente espacio para desviar el haz de muestra (recuadro inferior). Crédito:Microsistemas y Nanoingeniería, doi:10.1038 / s41378-019-0072-3
El dispositivo tenía el beneficio adicional de realizar todo el proceso completamente arraigado para la separación electroforética en solución libre, permitiendo a los investigadores obtener un mapa cuantitativo en pocos minutos, mucho más rápido que las técnicas convencionales. La mezcla no se vio afectada por el medio de soporte, y los investigadores pudieron estudiar interacciones moleculares débiles y no covalentes.
Saar y col. diseñó el dispositivo utilizando una unidad de electroforesis cuantitativa de fase nativa conectada a una unidad de tamaño de dispositivo de difusión de microfluidos (MDS). La plataforma combinada permitió componentes de movilidad electroforética específica (µ e1 ) para análisis descendente en chip, en función de la intensidad del campo eléctrico aplicado. Diseñaron tres canales aguas abajo de la unidad de electroforesis para mantener los productos de electrólisis alejados del chip sin entrar en el dispositivo. Minimizaron la cantidad de unidades individuales que impulsaron el flujo en el dispositivo, acoplado a un funcionamiento estable del dispositivo para la caracterización cuantitativa de muestras. Los científicos mantuvieron las salidas del electrolito separadas del dispositivo combinado para aplicar potencial eléctrico a través del dispositivo sin generar un cortocircuito eléctrico. y permitir la eliminación eficiente de cualquier producto de electrólisis sin acumulación para evitar fluctuaciones de presión.
El equipo de investigación aplicó el potencial eléctrico en conectores metálicos para generar una interfaz de metal y fluido fuera del chip de acuerdo con el prototipo de dispositivo diseñado por el mismo equipo. En este trabajo, Saar y col. diseñó un divisor de flujo en forma de Y y mantuvo las corrientes separadas hasta que llegaron al divisor para evitar un cortocircuito parcial. Calcularon la tasa de flujo del electrolito en el dispositivo para tener un efecto en el rendimiento del dispositivo.
(a) El voltaje aplicado a través de la cámara de electroforesis se ajustó en pasos lineales para dirigir fracciones específicas al análisis. (b) A continuación, se dimensionaron las fracciones formando imágenes de la unidad de dimensionamiento por difusión que consta de cuatro canales en los que se controló la extensión de la difusión de las moléculas de analito en su tampón portador circundante. (c) El tamaño promedio de las moléculas de analitos en cada una de las fracciones se encontró ajustando los perfiles fluorescentes observados para diferentes radios hidrodinámicos (Rh) y minimizando el mínimo error cuadrático medio entre el ajuste (línea punteada roja) y los datos (azul línea continua). (d) La intensidad de fluorescencia en el área de análisis varió dependiendo de la concentración de los analitos en cada una de las fracciones (arriba) y se encontró que los tamaños de los componentes en una mezcla binaria de lisozima y albúmina de suero bovino eran Rh =1.9 nm. y Rh =3,6 nm (abajo). (e) Esta mezcla binaria no pudo caracterizarse utilizando técnicas de dimensionamiento que no impliquen etapas de fraccionamiento previo, como la dispersión dinámica de la luz (DLS; línea de puntos azul claro). Su dimensionamiento fue posible utilizando el dispositivo descrito en este trabajo (línea continua azul oscuro). Crédito:Microsistemas y Nanoingeniería, doi:10.1038 / s41378-019-0072-3 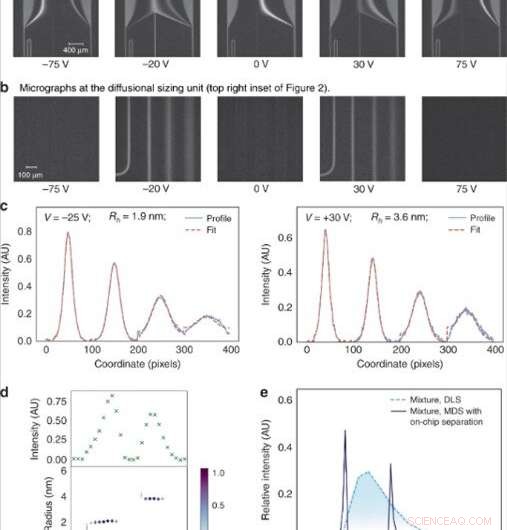
Obtuvieron imágenes de la boquilla en la que la muestra se encontraba con el medio portador como punto de referencia en el movimiento de partículas. Los científicos ajustaron el canal de tamaño de difusión o la velocidad de flujo para dimensionar con precisión las moléculas de analito, órdenes de magnitudes mayores o menores. Dado que diseñaron la plataforma de microfluidos con poli (dimetilsiloxano) (PDMS), los científicos eliminaron cualquier autofluorescencia en la configuración antes de analizar los datos de la imagen.
Luego utilizaron el dispositivo para analizar una mezcla binaria de proteínas de muestra; albúmina de suero bovino y lisozima humana. Para preservar los estados nativos de las moléculas de proteína, tomaron imágenes de las muestras sin etiquetas con un microscopio de longitud de onda ultravioleta de fabricación casera y cuantificaron la fluorescencia intrínseca de la muestra. Saar y col. confirmó la capacidad de separar la mezcla en sus componentes aplicando primero un conjunto de voltajes para registrar los perfiles fluorescentes. Luego registraron las movilidades electroforéticas de las proteínas (µ e1 ) combinado con la velocidad de flujo en el dispositivo para caracterizar la mayoría de las proteínas y sus complejos. Los científicos alteraron el caudal o el voltaje aplicado para analizar biomoléculas con diversos parámetros biofísicos.
Usando la plataforma, caracterizaron rápidamente mezclas de moléculas a nanoescala, donde los analitos individuales mostraron tamaños similares pero diversas propiedades electroforéticas. Según el histograma resultante, el equipo de investigación confirmó la presencia de dos muestras distintas. Relativamente, en un enfoque de separación convencional fuera del chip, el último paso requirió el fraccionamiento mediante la transferencia de muestras de una herramienta analítica a otra a través de tubos interconectados, limitar el rendimiento del dispositivo. La concentración de proteína total en el estudio se aproximó a 100 µM y los científicos detectaron con precisión el límite de sensibilidad a aproximadamente 100 nM. en relación con la fluorescencia intrínseca de las fracciones de proteínas. Para compuestos ópticamente inactivos, Saar y col. sugieren una estrategia alternativa de detección y caracterización, como la detección de masa seca.
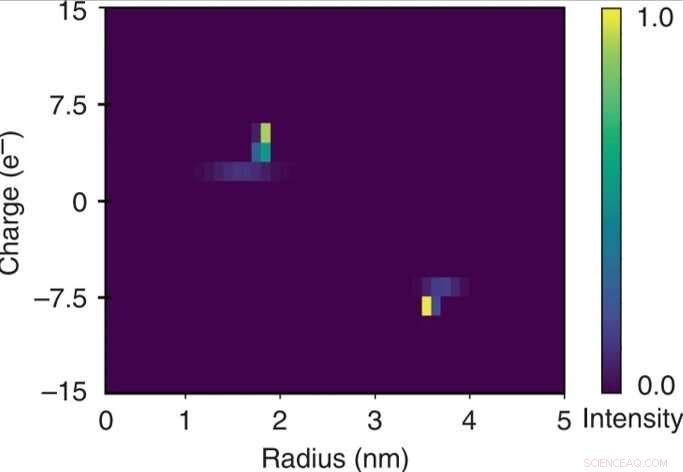
Caracterización bidimensional rápida de una mezcla de albúmina de suero bovino y lisozima humana. La naturaleza cuantitativa de la unidad de separación electroforética permitió relacionar el potencial aplicado con la movilidad electroforética μel de cada una de las fracciones. Estos datos se utilizaron en combinación con los radios hidrodinámicos extraídos (Rh) para estimar las cargas efectivas (q) de las moléculas en cada una de las fracciones y para construir un mapa bidimensional q vs Rh de la mezcla en una escala de tiempo de unos pocos minutos por monitoreando el dispositivo solo en un único marco de imagen. Crédito:Microsistemas y Nanoingeniería, doi:10.1038 / s41378-019-0072-3
Saar y col. utilizó la estrategia para obtener mapas característicos bidimensionales (2-D) de la mezcla de proteínas como prueba de concepto. Extrajeron información cuantitativa del paso de separación y relacionaron los potenciales aplicados con las movilidades electroforéticas de las especies para estimar la eficacia del dispositivo. Registraron la corriente que fluye en el sistema durante el funcionamiento normal y cuando la cámara de separación estaba en cortocircuito para estimar la resistencia eléctrica total del dispositivo y los electrodos.
Los investigadores calcularon la movilidad electroforética como el movimiento de una partícula en un campo eléctrico para cada una de las fracciones. Basado en los datos experimentales, el mapa de características 2-D construido incluyó la carga efectiva (q) y el radio hidrodinámico (Rh) de la mezcla. Las unidades de carga elemental resultantes de las proteínas específicas coincidían con los valores estimados en otros lugares. Obtuvieron el mapa bidimensional completo al monitorear solo un único marco de imagen para un análisis rápido de la solución.
El tiempo analítico del dispositivo de microfluidos desde la separación hasta el dimensionamiento por difusión y la obtención de imágenes fue de aproximadamente 14 segundos. Los científicos construyeron el mapa 2-D experimental usando solo 3 µL de muestra durante siete minutos en total, órdenes de magnitud más rápido que la escala de tiempo para realizar geles de proteínas 2-D convencionales. El equipo de investigación llevó a cabo una amplia gama de interacciones biomoleculares, en solución, directamente en condiciones nativas que anteriormente eran difíciles de realizar en el laboratorio.
De este modo, Kadi L. Saar y sus colaboradores desarrollaron un dispositivo de microfluidos que combina la separación en el chip con el análisis directo en el chip para reemplazar los enfoques de microescala convencionales existentes. Usando el dispositivo, analizaron rápidamente una mezcla binaria de proteínas que no podían identificarse como componentes individuales mediante los enfoques de dimensionamiento de soluciones existentes. Construyeron un mapa característico 2-D de la mezcla heterogénea en una escala de tiempo rápida para abrir la posibilidad de caracterización de proteínas en solución con una resolución de tiempo sin precedentes en comparación con las técnicas biofísicas existentes.
© 2019 Science X Network