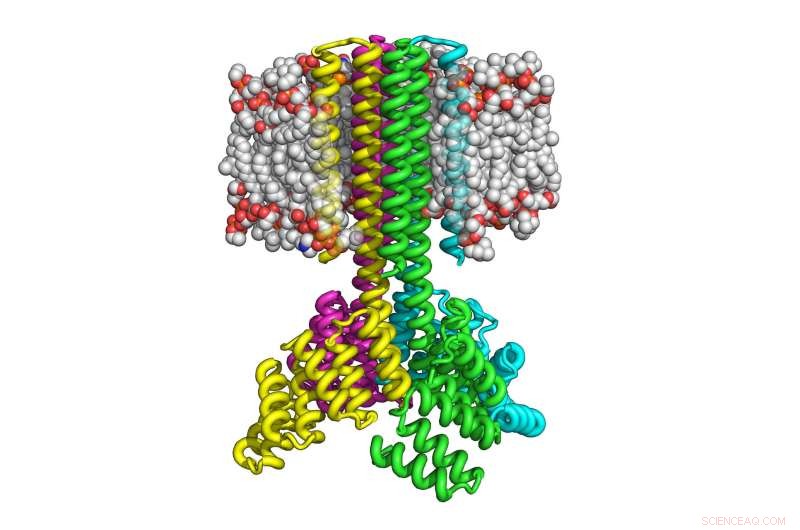
Esta ilustración muestra cómo cuatro copias de proteína transmembrana diseñada por computadora se ensamblaron para formar un tetrámero en forma de cohete con una base citoplasmática ancha que se canaliza en ocho hélices transmembrana y que se orienta correctamente en la membrana. Crédito:Instituto de Diseño de Proteínas de la Universidad de Washington
Ahora es posible crear complejos, proteínas transmembrana diseñadas a medida desde cero, los científicos informan esta semana. El avance, dirigido por ingenieros moleculares del Instituto de Diseño de Proteínas de la Universidad de Washington, permitirá a los investigadores crear proteínas transmembrana que no se encuentran en la naturaleza para realizar tareas específicas.
En el mundo viviente Las proteínas transmembrana se encuentran incrustadas en la membrana de todas las células y orgánulos celulares. Son fundamentales para que funcionen con normalidad. Por ejemplo, muchas proteínas transmembrana naturales actúan como puertas de entrada para el movimiento de sustancias específicas a través de una membrana biológica. Algunas proteínas transmembrana reciben o transmiten señales celulares. Debido a esos roles, muchos medicamentos están diseñados para atacar proteínas transmembrana y alterar su función.
"Nuestros resultados allanan el camino para el diseño de proteínas de membrana multicapa que podrían imitar las proteínas que se encuentran en la naturaleza o tener una estructura completamente nueva". función y usos, "dijo David Baker, un profesor de bioquímica de la Facultad de Medicina de la Universidad de Washington y director del Instituto de Diseño de Proteínas de la Universidad de Washington, quien dirigió el proyecto. La investigación aparece en la edición del 1 de marzo de la revista. Ciencias . Peilong Lu, un investigador senior en el laboratorio Baker, es el autor principal del artículo.
Pero comprender cómo se unen las proteínas transmembrana y cómo funcionan ha resultado ser un desafío. Debido a que actúan mientras están incrustados dentro de la membrana celular, Las proteínas transmembrana han demostrado ser más difíciles de estudiar que las proteínas que operan en la solución acuosa que forma el citoplasma de las células o en el líquido extracelular.
En el nuevo estudio, Lu y sus compañeros de trabajo usaron un programa de computadora, desarrollado en el laboratorio Baker y llamado Rosetta, que puede predecir la estructura en la que se plegará una proteína después de haber sido sintetizada. La arquitectura de una proteína es crucial porque la estructura de una proteína determina su función.
La forma de una proteína se forma a partir de interacciones complejas entre los aminoácidos que forman la cadena de proteínas y entre los aminoácidos y el entorno circundante. Por último, la proteína asume la forma que mejor equilibra todos estos factores para que la proteína alcance el estado energético más bajo posible.
El programa Rosetta utilizado por Lu y sus colegas puede predecir la estructura de una proteína teniendo en cuenta estas interacciones y calculando el estado energético general más bajo. No es inusual que el programa cree decenas de miles de estructuras modelo para una secuencia de aminoácidos y luego identifique las que tienen el estado de energía más bajo. Se ha demostrado que los modelos resultantes representan con precisión la estructura que probablemente asumirá la secuencia en la naturaleza.
Determinar la estructura de las proteínas transmembrana es difícil porque porciones de proteínas transmembrana deben pasar a través del interior de la membrana, que está hecho de grasas aceitosas llamadas lípidos.
En fluidos acuosos, Los residuos de aminoácidos que tienen cadenas laterales polares (componentes que pueden tener una carga bajo ciertas condiciones fisiológicas o que participan en el enlace de hidrógeno) tienden a estar ubicados en la superficie de la proteína, donde pueden interactuar con el agua. que tiene cargas laterales negativas y positivas a su molécula. Como resultado, Los residuos polares en las proteínas se denominan hidrófilos. o "amante del agua".
Residuos no polares, por otra parte, tienden a encontrarse empaquetados dentro del núcleo de proteína lejos del fluido acuoso polar. Dichos residuos se denominan hidrófobos o "temerosos del agua". Como resultado, la interacción entre los residuos de la proteína amantes y temerosos del agua y los fluidos acuosos circundantes ayuda a impulsar el plegamiento de la proteína y estabiliza la estructura final de la proteína.
En membranas, sin embargo, El plegamiento de proteínas es más complicado porque el interior lipídico de la membrana es no polar, es decir, no tiene separación de cargas eléctricas. Esto significa que para ser estable, la proteína debe colocarse no polar, residuos temerosos del agua en su superficie, y empacar su polar, Residuos amantes del agua en el interior. Luego, debe encontrar una manera de estabilizar su estructura creando enlaces entre los residuos hidrófilos dentro de su núcleo.
La clave para resolver el problema dice Lu, era aplicar un método desarrollado por el laboratorio Baker para diseñar proteínas de modo que la polar, Los residuos hidrofílicos encajan de tal manera que suficientes formarían interacciones polar-polares que pueden unir la proteína desde adentro.
"Reunir estas 'redes de enlaces de hidrógeno enterradas' fue como armar un rompecabezas, "Dijo Baker.
Con este enfoque, Lu y sus colegas pudieron fabricar las proteínas transmembrana diseñadas dentro de bacterias y células de mamíferos utilizando hasta 215 aminoácidos. Las proteínas resultantes demostraron ser muy estables térmicamente y capaces de orientarse correctamente sobre la membrana. Al igual que las proteínas transmembrana naturales, las proteínas son multipaso, lo que significa que atraviesan la membrana varias veces, y se ensamblan en complejos de múltiples proteínas estables, como los dímeros, trímeros y tetrámeros.
"Hemos demostrado que ahora es posible diseñar con precisión complejos, proteínas transmembrana multipaso que se pueden expresar en las células. Esto permitirá a los investigadores diseñar proteínas transmembrana con estructuras y funciones completamente nuevas, "dijo Lu.