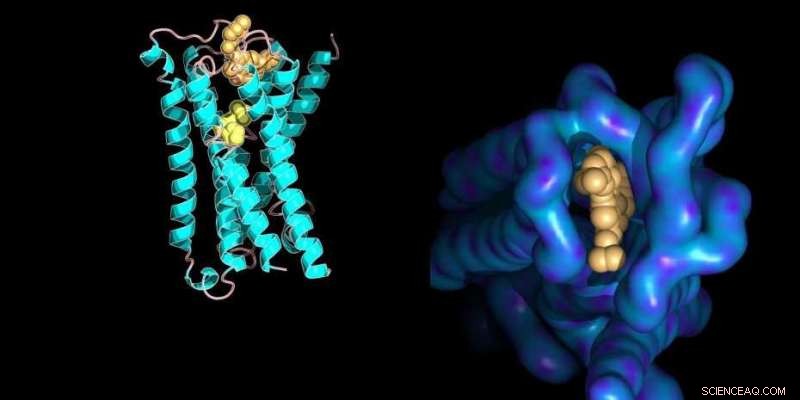
Modelo molecular de un receptor de membrana de proteína unido a sus ligandos de molécula pequeña con detalles estructurales del sitio de unión alostérico (derecha). Crédito:Ilaria Piazza / ETH Zurich / PDB, entrada de la base de datos 4MQT
Los investigadores de ETH Zurich han utilizado un nuevo enfoque para descubrir interacciones previamente desconocidas entre proteínas y pequeñas moléculas metabólicas en células bacterianas. La técnica también se puede utilizar para probar el efecto de los medicamentos.
La ciencia ahora tiene una nueva "ómica":una interactómica, que se ocupa de las interacciones entre proteínas y moléculas pequeñas. Habiéndose centrado previamente en genómica o proteómica, Los biólogos de sistemas ahora pueden agregar interactómica proteína-metabolito a la mezcla. La genómica se ocupa del análisis sistemático de todos los genes de un organismo, mientras que la proteómica se ocupa de todo el conjunto de proteínas dentro de una unidad biológica.
Con interactómica proteína-metabolito, el grupo liderado por Paola Picotti, Profesor de Biología de sistemas moleculares en ETH Zurich, ahora ha agregado otro nivel a los "ómicos" existentes. Los investigadores publicaron recientemente un estudio en la revista Cell, en el que analizaron y cuantificaron sistemáticamente las interacciones de todas las proteínas con metabolitos (pequeñas moléculas metabólicas) a nivel de todo el proteoma por primera vez, estableciendo las relaciones entre ellos.
Diferentes sitios de corte
Los investigadores han demostrado cuántas de las proteínas y enzimas presentes en una célula bacteriana de E. coli interactúan con los metabolitos. Para hacerlo utilizaron un enfoque conocido como proteólisis limitada (LiP), combinado con análisis de espectrometría de masas.
Los investigadores extrajeron fluido celular, que contiene proteínas, de las células bacterianas. Luego agregaron un metabolito a cada muestra y le permitieron interactuar con las proteínas. Finalmente, cortan las proteínas en pedazos más pequeños (péptidos) usando "tijeras moleculares". En total, los investigadores probaron 20 metabolitos diferentes y sus interacciones con proteínas de esta manera.
Cuando una proteína interactúa con un metabolito, ya sea que se asiente en el sitio activo de la proteína o se adhiera a otro sitio, la estructura de la proteína está alterada. Las "tijeras moleculares" luego lo cortan en diferentes sitios de la estructura original, resultando en un conjunto diferente de péptidos.
Usando el espectrómetro de masas, los investigadores midieron todas las piezas presentes en la muestra y alimentaron los datos obtenidos en una computadora para reconstruir las diferencias y cambios estructurales, y en qué parte de la proteína se localizan.
Cientos de nuevas interacciones descubiertas
Conocimiento sobre el interactoma proteína-metabolito (las interacciones entre proteínas y metabolitos y las redes moleculares (de señales) que les pertenecen), anteriormente era muy limitado en comparación con lo que se sabía sobre las interacciones entre diferentes proteínas o entre proteínas y ADN o ARN. El estudio ahora ha aumentado drásticamente este conocimiento.
Usando este enfoque con E. coli, Picotti y su equipo descubrieron alrededor de 1, 650 interacciones diferentes proteína-metabolito, de los cuales más de 1, 400 eran previamente desconocidos. También salieron a la luz miles de sitios de unión en proteínas a las que se pueden unir los metabolitos. "Aunque el metabolismo de E. coli y moléculas asociadas ya es muy conocido, logramos descubrir muchas interacciones nuevas y los sitios de unión correspondientes, "dice Picotti. Esto demuestra el enorme potencial del método:" Los datos que producimos con esta técnica ayudarán a identificar nuevos mecanismos reguladores, enzimas desconocidas y nuevas reacciones metabólicas en la célula ".
El cambio estructural regula la actividad
En su estudio, los investigadores también muestran que las pequeñas moléculas metabólicas prefieren unirse (y así regular) proteínas cuya concentración es más o menos constante en el tiempo. Esto sugiere que la unión de metabolitos a proteínas y los cambios en las concentraciones de proteínas son dos vías complementarias por las cuales las células regulan la actividad de las proteínas.
Las proteínas pueden activarse o desactivarse con relativa rapidez mediante un cambio estructural mediado por metabolitos. "Este tipo de cambio estructural se puede revertir más rápidamente, "explica Picotti. Desde la perspectiva de la célula, esto a menudo tiene sentido, porque el uso de la vía que implica un cambio en la concentración significa que la célula debe desmantelar o reconstruir las proteínas, que le cuesta más tiempo, energía y recursos.
Picotti y sus colegas también han podido demostrar que muchas enzimas son menos exigentes de lo que se pensaba anteriormente:aparentemente pueden unirse y alterar químicamente varios metabolitos diferentes. Hasta ahora, se asumió que las enzimas eran en su mayoría específicas de unas pocas moléculas muy similares.
La industria farmacéutica está muy interesada en el nuevo enfoque, ya que se puede utilizar para probar la interacción de fármacos con proteínas celulares e identificar los objetivos de un fármaco. Los investigadores podrían investigar a qué proteínas y a qué sitios se une este fármaco, cómo altera su estructura y, por tanto, influye en su actividad. Esto facilitaría y aceleraría las pruebas y el desarrollo de nuevos fármacos.