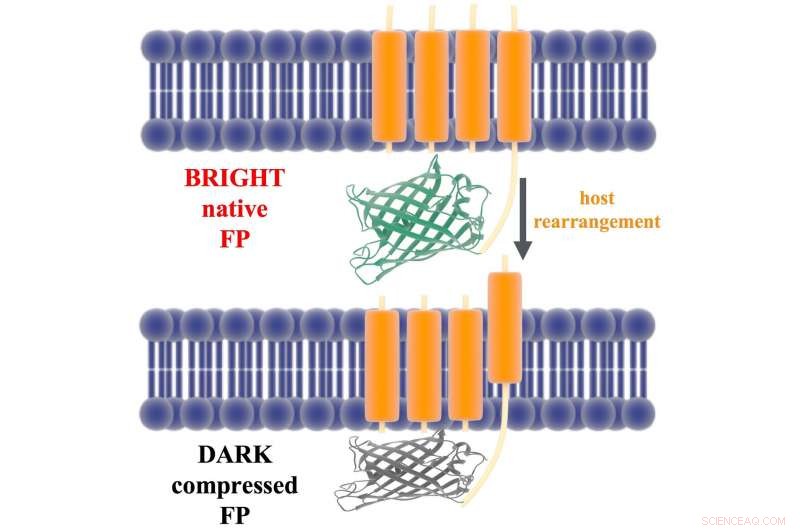
Una ilustración muestra la correa que une el dominio fluorescente de una proteína ArcLight (verde y gris) con su dominio sensor de voltaje (naranja) en la membrana de una neurona. Los científicos de Rice sugieren en un nuevo estudio que la despolarización de la membrana mueve el dominio de detección de voltaje, que a su vez comprime la proteína fluorescente y la apaga. Crédito:Lena Simine
Los científicos de la Universidad de Rice han apagado efectivamente un debate sobre el mecanismo detrás de un biosensor fluorescente que monitorea las neuronas al detectar cambios en el voltaje.
El trabajo dirigido por el químico teórico de Rice Peter Rossky y la investigadora postdoctoral Lena Simine confirmó a través de simulaciones por computadora su teoría de que un proceso mecánico controla la extinción de la fluorescencia en ArcLight. un indicador de voltaje sintético colocado dentro de las proteínas que recubren las membranas internas de las neuronas.
A través de sus modelos, los investigadores acoplaron tanto el mecanismo como la fluorescencia a la fuerza de los campos eléctricos que observaron a través del cromóforo, la parte fluorescente de la proteína. Sus resultados mostraron que una simple medida del campo en una simulación podría usarse para predecir si los nuevos sensores fluorescentes se comportarán antes de que los investigadores los sinteticen y qué tan bien se comportarán. Dijo Rossky.
El estudio aparece en el Revista de la Sociedad Química Estadounidense .
ArcLight, desarrollado por el neurocientífico Vincent Pieribone de Yale en 2012, es una proteína indicadora de voltaje de fluorescencia codificada genéticamente. Contiene una mutación que hace que la señal de fluorescencia se atenúe cuando aumenta el voltaje y se ilumine cuando el voltaje cae. Eso lo hace útil para rastrear señales en el sistema nervioso al expresar la proteína en las neuronas y ver cómo se iluminan.
La proteína está unida a la pared celular de la neurona por un componente sensor de voltaje que se mueve unos angstroms cuando una señal de otra neurona cambia la carga eléctrica en la membrana. Los investigadores de Rice teorizaron que el movimiento empuja a la proteína contra la membrana, comprimiéndolo y apagando la fluorescencia.
Rossky dijo que cambiar la forma de la proteína acerca dos residuos un nanómetro entre sí. Eso es suficiente para dictar cómo el cromóforo se deshace de la energía, ya sea como luz (renunciando a fotones y emitiendo fluorescencia) o como calor.
"Hicimos la hipótesis de qué cambio de geometría ocurre en la proteína como resultado de la respuesta de la membrana, Rossky dijo. Y luego preguntamos:'¿Esto cambia la fluorescencia?' Y descubrimos que sí. Además, demostramos que monitorear una calidad mucho más simple, el campo eléctrico a lo largo de dos ejes de donde proviene la fluorescencia, es suficiente para describir completamente la respuesta ".
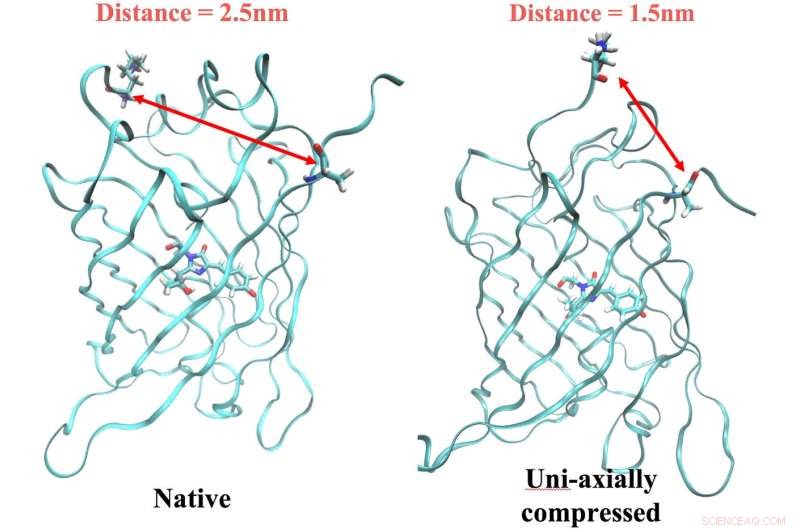
La proteína ArcLight nativa a la izquierda, con residuos de anclaje separados por 2,5 nanómetros, emite fluorescencia cuando se activa con luz en la frecuencia correcta. Pero la fluorescencia se apaga cuando la proteína se comprime, lo que acerca las anclas un nanómetro. Los investigadores de Rice encontraron un vínculo entre el mecanismo y una señal eléctrica en la proteína que se puede utilizar como marcador al simular nuevas proteínas fluorescentes con modelos informáticos. Crédito:Lena Simine
ArcLight demostró ser un buen modelo. Pieribone, un colaborador de Rice, dijo a los asistentes a una conferencia de 2014 en Rice que ni siquiera él sabía exactamente cómo funcionaba. La conferencia inspiró a Simine, que acababa de llegar a Rice, para embarcarse en un estudio del mecanismo.
"Pensé, 'Eso suena como un buen proyecto para mí, '" ella dijo.
Trabajar con investigadores del grupo de José Onuchic en el Centro de Física Biológica Teórica (CTBP) de Rice permitió a Simine, un físico químico de formación, para aprovechar la experiencia del centro en la simulación de proteínas para realizar pruebas.
Ella dijo que un debate de una década entre científicos no pudo determinar si las propiedades mecánicas o eléctricas de las proteínas causaban su fluorescencia. Resultó ser un poco de ambos.
"Un artículo reciente dio evidencia computacional de que es predominantemente electrostático, y tiene sentido porque la proteína es muy suave, "Dijo Simine." También pensamos que esas mutaciones se adhieren a la membrana, y cuando lo hagan, la orientación de la proteína permite que la proteína se comprima ". Descubrió que los cambios electrostáticos en la membrana neuronal desencadenan el cambio físico que apaga la fluorescencia, pero también dejó un rastro eléctrico en la proteína que se pudo observar en la simulación.
"Pensamos un poco y se nos ocurrió una coordenada de reacción, ", dijo." Podemos tomar cualquier mutación de la secuencia de esta proteína y traducirla en dos números que son las entradas para este modelo, los campos electrostáticos alrededor del cromóforo. Es agradable, elegante teoría fenomenológica ".
El laboratorio planea probar su técnica en proteínas fluorescentes sintetizadas a medida y simulaciones coincidentes para ver si su teoría y experimentación continúan alineándose. Si lo hacen, esperan que sus modelos sean de gran utilidad para los biólogos sintéticos que fabrican nuevas clases de marcadores fluorescentes.
"Si desea conocer la fluorescencia de una molécula determinada, tu haces el experimento, ", Dijo Rossky." Pero si quieres saber por qué funciona, estos cálculos son increíblemente valiosos ".