La concepción de un artista del poder del diseño computacional para explorar e iluminar péptidos estructurados en el vasto paisaje energético. Crédito:Vikram Mulligan / Instituto de Diseño de Proteínas de la Universidad de Washington
Nuevas estrategias computacionales informadas esta semana en Ciencias podría ayudar a hacer realidad la promesa de los medicamentos basados en péptidos. Los péptidos son similares a las moléculas de proteínas, pero difieren en su tamaño más pequeño, estructura y funciones.
Los péptidos macrocíclicos han despertado el interés de la industria farmacéutica, porque tienen ciertas propiedades físicas y químicas que podrían convertirse en la base de una nueva generación de medicamentos.
Los péptidos pequeños tienen los beneficios de los fármacos de moléculas pequeñas, como una aspirina, y grandes terapias de anticuerpos, como rituximab, con menos inconvenientes. Son estables como moléculas pequeñas y potentes y selectivos como anticuerpos.
Un ejemplo de una historia de éxito con un fármaco de péptidos macrocíclicos es la ciclosporina, un inmunosupresor para trasplantes de órganos y algunos trastornos autoinmunes.
Antes del trabajo descrito en el Ciencias papel, No había forma de diseñar sistemáticamente macrociclos de péptidos ordenados como la ciclosporina.
Péptidos naturales que pueden servir como puntos de partida fiables, o andamios, son pocos. Igual de frustrante es que a menudo no funcionan como se esperaba cuando se reutilizan. En lugar de, los investigadores habían recurrido a la detección de grandes bibliotecas de compuestos generadas aleatoriamente con la esperanza de encontrar lo que necesitaban.
Los métodos cubiertos en el informe, El "diseño computacional completo de macrociclos de péptidos ordenados" ahora resuelve estos problemas.
Esta ilustración conceptual muestra cómo se pueden programar heteropolímeros de péptidos a partir de componentes más pequeños para explorar un panorama energético diverso. Crédito:Ahmad Hosseinzadeh y Khosro Khosravi
Los autores principales son Parisa Hossienzadeh, Gaurav Bhardwaj y Vikram Mulligan, del Departamento de Bioquímica de la Facultad de Medicina de la Universidad de Washington y del Instituto de Diseño de Proteínas de la Universidad de Washington. El autor principal es David Baker, profesor de bioquímica y director del instituto. Baker también es investigador del Instituto Médico Howard Hughes.
"En nuestro periódico, "señalaron los investigadores, "Describimos estrategias computacionales para diseñar péptidos que adopten diversas formas con una precisión muy alta y para proporcionar una cobertura completa de las estructuras que pueden formarse con péptidos cortos".
Señalaron las ventajas de este nuevo enfoque computacional:
Primero, pudieron diseñar y compilar una biblioteca de muchos andamios de péptidos estables nuevos que pueden proporcionar las plataformas básicas para la arquitectura de candidatos a fármacos. Sus métodos también se pueden utilizar para diseñar péptidos personalizados adicionales con formas arbitrarias bajo demanda.
"Tomamos muestras del paisaje diverso de formas que pueden formar los péptidos, como guía para diseñar la próxima generación de medicamentos, ", dijeron los investigadores.
La clave para el control de la geometría y química de las moléculas fue el diseño de péptidos con aminoácidos naturales, llamados L-aminoácidos, y sus opuestos espejo que contienen D-aminoácidos. (La L y D significan palabras latinas para rotar hacia la izquierda o hacia la derecha, ya que algunas estructuras moleculares pueden tener quiralidad o ser diestros o zurdos).
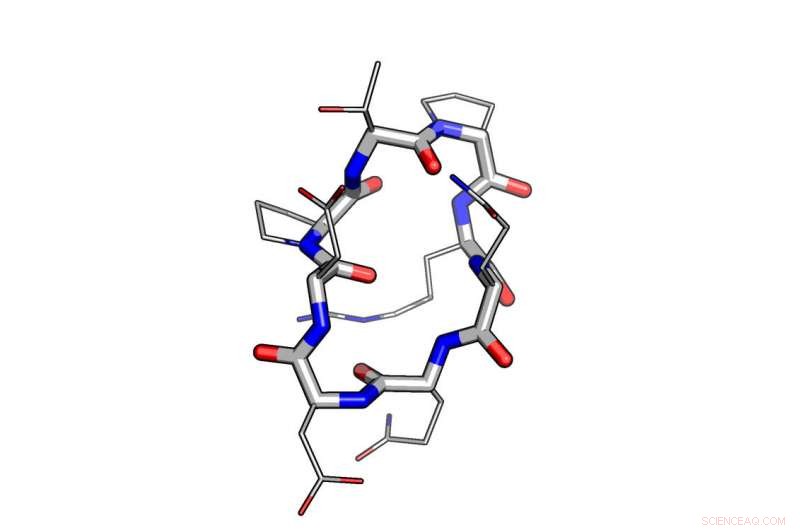
Un ejemplo del uso del diseño computacional en el modelado de un péptido. Crédito:Instituto de Diseño de Proteínas de la Universidad de Washington
Los D-aminoácidos mejoraron las propiedades farmacológicas al aumentar la resistencia a las enzimas naturales que descomponen los péptidos. La inclusión de D-aminoácidos en los diseños también permite una gama más diversa de formas.
Diseñar péptidos requiere mucho poder informático, resultando en cálculos costosos. Los investigadores dieron crédito a un grupo de científicos ciudadanos y voluntarios que donaron los minutos de su teléfono inteligente celular y tiempo de computadora. La supercomputadora Hyak de la Universidad de Washington también ejecutó algunos de los programas.
Los investigadores señalaron direcciones futuras para sus enfoques de diseño computacional de péptidos. Esperan diseñar péptidos que puedan penetrar las membranas celulares y entrar en las células vivas.
En otros aspectos, planean agregar nuevas funcionalidades a las estructuras de péptidos estabilizando los motivos de unión en las interfaces proteína-proteína para estudios de ciencias básicas. Para aplicaciones clínicas, anticipan el uso de sus métodos y andamios para desarrollar fármacos basados en péptidos.