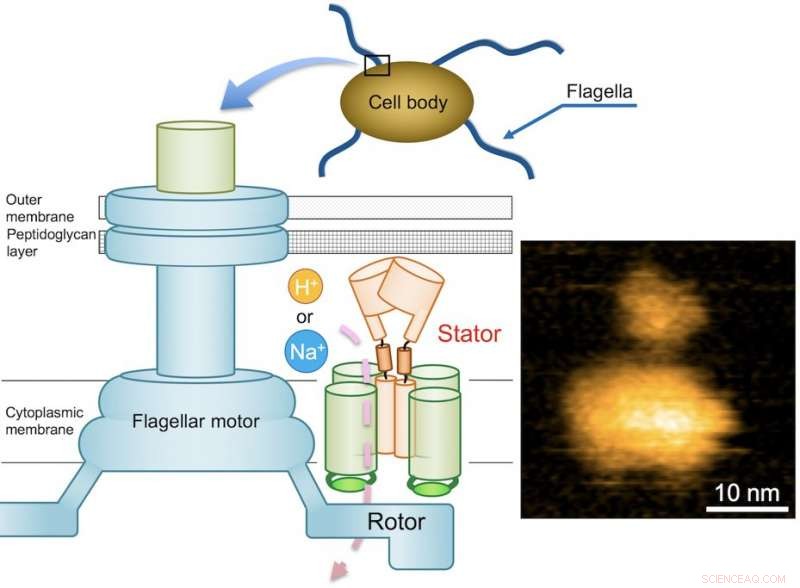
Fig. 1. Diagrama esquemático del motor flagelar bacteriano y una imagen AFM típica del complejo MotPS purificado. Crédito:Universidad de Osaka
Las bacterias nadan de muchas formas diferentes, y los motores que impulsan su natación son muy variados, lo que implica una respuesta adaptativa a un entorno. Uno de los motores más comúnmente identificados es el flagelo. Aunque proporcionar motilidad es una característica principal de los flagelos, También se les han atribuido propiedades adhesivas y otras.
El motor flagelar consta de un rotor y una docena de unidades de estator, y es impulsado por la energía creada por los iones cuando migran a través de la membrana celular. Mientras que el motor flagelar de algunas bacterias entéricas y de agua dulce, como Salmonella, funciona con protones (iones de hidrógeno, H + ), que consta de cuatro copias de la proteína MotA y dos copias de MotB, Los motores de otras bacterias que viven en ambientes de agua salada funcionan con iones de sodio (Na + ).
En el caso de Bacillus subtilis (B. subtilis), una bacteria que se encuentra comúnmente en el suelo, el motor flagelar tiene dos tipos distintos de unidades de estator:MotAB tipo H + y MotPS tipo Na +. Mientras se identifican los componentes, el mecanismo que impulsa el motor flagelar sigue siendo desconocido. Este misterio se ve agravado por el hecho de que los flagelos no son claramente visibles bajo el microscopio óptico. Estos desafíos pusieron a un equipo de investigadores centrados en la Universidad de Osaka en una búsqueda para dilucidar el mecanismo desconocido. Informaron sus hallazgos en Avances de la ciencia .
"Los análisis precisos de la dinámica estructural del motor flagelar utilizando microscopía de fuerza atómica de alta velocidad (HS-AFM) combinada con el análisis mutacional proporcionaron información sobre el mecanismo de generación de par del motor flagelar, "Dice el primer autor del estudio Naoya Terahara." Hicimos observaciones en tiempo real de los cambios estructurales inducidos por Na + en el dominio de MotS que se une al peptidoglicano, un polímero que consta de azúcares y aminoácidos en bacterias ".
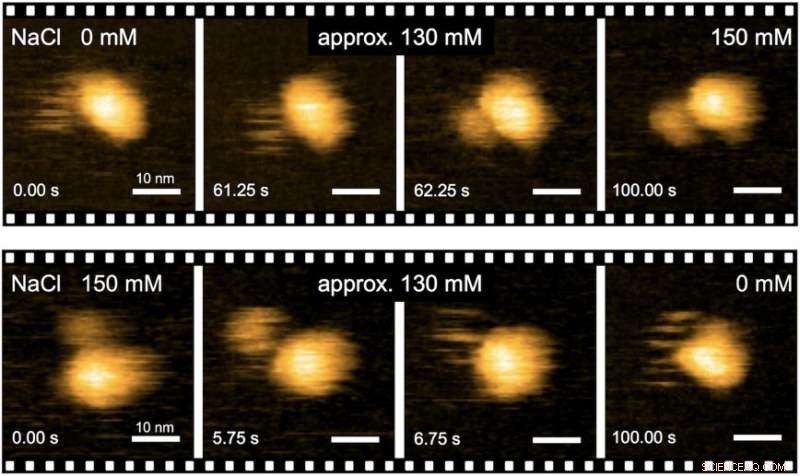
Fig. 2. Imágenes en tiempo real de transiciones estructurales inducidas por Na + del complejo MotPS por HS-AFM. Crédito:Universidad de Osaka
Para comprender cómo responde el complejo de proteínas MotPS a los cambios en el medio ambiente, los investigadores primero caracterizaron la relación par-velocidad del motor B. subtilis en un amplio rango de concentraciones de Na +. La velocidad máxima del motor se redujo de aproximadamente 200 a 80 Hz cuando la concentración externa de Na + se incrementó de 0 a 200 mM, aunque el par de pérdida no se modificó en absoluto.
Notablemente, cuando el dominio de MotB que se une al peptidoglicano fue reemplazado por el de MotS, el motor quimérico mantuvo la misma curva de par-velocidad que el motor MotAB en presencia de iones Na + pero no en ausencia. Esto significa que Na + es el principal impulsor del ensamblaje y activación del motor flagelar, independientemente de su composición.
"En ausencia de iones de sodio, MotPS existe como una forma inactiva en la membrana citoplásmica porque MotSc, el dominio periplásmico C-terminal de MotS, Adopta una conformación desplegada. Cuando aumenta la concentración de iones de sodio, la unión de los iones a MotSc induce el plegamiento del dominio PGB y facilita su dimerización de una manera altamente cooperativa, ", dice el autor correspondiente Tohru Minamino.
A través de este estudio, el equipo también ha demostrado la viabilidad de usar HS-AFM combinado con análisis mutacional para estudiar estructuras bacterianas intrincadas, que puede proporcionar información valiosa para la producción industrial de productos comercialmente viables, como proteasas y terapéuticas, como en el caso de B. subtilis.
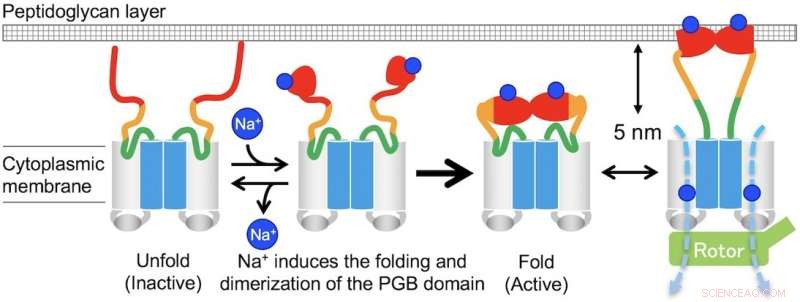
Fig. 3. Modelo para el montaje y desmontaje del complejo de estator MotPS inducido por Na +. Crédito:Universidad de Osaka