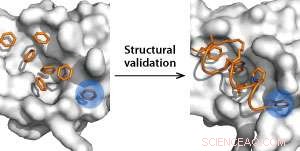
Detección de bolsas de unión usando moléculas de benceno como sondas (izquierda) y validación estructural usando péptidos grapados (derecha). Crédito:Y. S. Tan et al.
En la búsqueda de nuevas terapias contra el cáncer, Los investigadores de A * STAR han ideado una estrategia computacional que descubre cualquier sitio de unión previamente desconocido o "bolsillos" en los objetivos de los fármacos.
Es probable que surjan tratamientos más eficaces contra el cáncer a partir de la línea de desarrollo de fármacos. El descubrimiento de fármacos contra el cáncer depende de la identificación y caracterización de las bolsas de unión en las proteínas diana. Típicamente, esta evaluación utiliza técnicas computacionales que se basan en estructuras proteicas estáticas. Sin embargo, las proteínas tienen una flexibilidad inherente que provoca una tendencia a cambiar de forma al entrar en contacto con los fármacos. Ciertos bolsillos de unión permanecen indetectables a menos que interactúen con una sustancia apropiada y, por lo tanto, se pasan por alto en las simulaciones convencionales. Estos bolsillos ocultos sin embargo, Suelen ser sitios hidrófobos o hidrófobos que solo se abren cuando hay sustancias de baja polaridad.
Para abordar esto, Yaw Sing Tan y Chandra Verma del Instituto de Bioinformática han desarrollado un método basado en sondas llamado dinámica molecular de mapeo de ligandos (LMMD). Utilizaron esta técnica para buscar bolsas de unión ocultas en la proteína diana anticancerígena MDM2. Las predicciones resultantes fueron validadas experimentalmente por colaboradores de larga data del Laboratorio p53 de A * STAR y el Instituto de Ciencias Químicas e Ingeniería, así como por biólogos estructurales de la Universidad de Newcastle. REINO UNIDO.
Tan explica que inicialmente había diseñado este método basado en sondas para otra proteína objetivo y lo usó con éxito para encontrar un bolsillo de unión oculto que permanecía cerrado en las simulaciones convencionales. "Luego decidimos aplicar este enfoque a MDM2 para ver si podíamos descubrir sitios de unión previamente desconocidos que pudieran mejorar la potencia de los inhibidores de MDM2 existentes". " él añade.
Usando moléculas de benceno como sondas de detección de bolsas hidrófobas, los investigadores identificaron computacionalmente dos nuevos sitios de unión en MDM2. "Estábamos emocionados de ver que estos sitios se encuentran muy cerca del bolsillo de unión de la proteína supresora de tumores p53, "dice Tan.
Es más, los investigadores esperan que los sitios recién descubiertos conduzcan a péptidos grapados más potentes, que son hélices de aminoácidos estabilizadas químicamente por una cadena de hidrocarburos que han surgido recientemente como poderosos activadores de p53. Como consecuencia, crearon péptidos grapados a partir de análogos que se sabe que se unen estrechamente a MDM2 y reactivan p53, y determinó la afinidad de estos péptidos por MDM2. Sus simulaciones mostraron que los péptidos se unían a MDM2 con más fuerza que p53 en los bolsillos y coincidían con experimentos de cristalografía de rayos X y biofísicos.
"Este método podría utilizarse para interrogar a otros objetivos proteicos contra el cáncer para descubrir nuevos sitios de unión que podrían ser el objetivo de la inhibición". ", dice Tan. El equipo ahora está trabajando para ampliar el alcance de las sondas LMMD a otros tipos de ligandos.