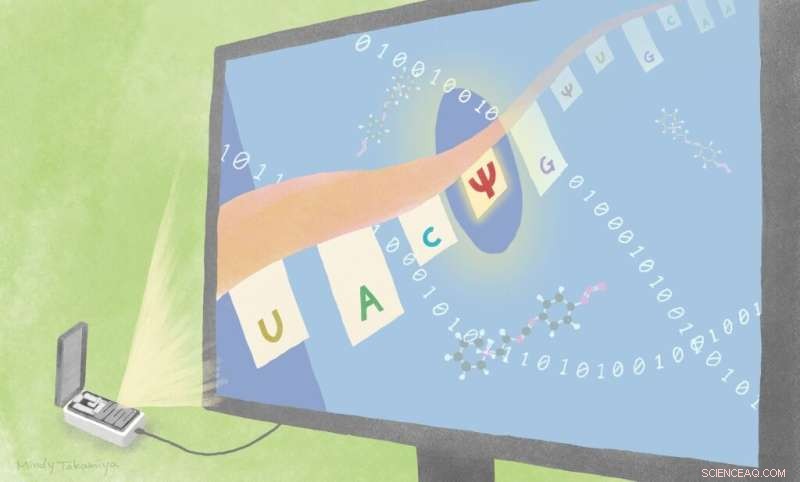
Los científicos encontraron dos nuevas formas de identificar sustituciones de uracilo a pseudouridina, combinando la tecnología de secuenciación existente con diferentes enfoques. Uno es con cálculo basado en algoritmos y el otro es con una sonda química CMC. Crédito:Mindy Takamiya/iCeMS de la Universidad de Kyoto
Dos nuevos enfoques podrían ayudar a los científicos a utilizar la tecnología de secuenciación existente para distinguir mejor los cambios en el ARN que afectan la forma en que se lee el código genético.
Los científicos de la Universidad de Kyoto están cada vez más cerca de encontrar formas de identificar cambios en las secuencias de ARN que afectan la formación de proteínas y pueden causar enfermedades. Su enfoque, publicado en la revista Genomics , utiliza algoritmos de probabilidad junto con un dispositivo de secuenciación de bolsillo ya disponible.
"Las modificaciones que se encuentran en todos los tipos de ARN biológico influyen en la regulación génica, que finalmente decide cómo funcionan las diferentes células en nuestro cuerpo", explica Ganesh Pandian Namasivayam del Instituto de Ciencia Integrada de Material Celular (WPI-iCeMS) de la Universidad de Kyoto. "Las anomalías en estas modificaciones pueden provocar enfermedades graves, como diabetes, trastornos del neurodesarrollo y cáncer. Saber cómo y dónde están estas modificaciones del ARN es de suma importancia desde un punto de vista clínico", añade Soundhar Ramaswamy, primer autor del estudio.
Ya existen formas de identificar las modificaciones del ARN, pero son insuficientes. Los enfoques biofísicos como la cromatografía y la espectrometría de masas solo pueden procesar pequeñas cantidades de ARN a la vez. Los métodos de secuenciación de alto rendimiento, que pueden procesar grandes cantidades de ARN, implican una preparación de muestras laboriosa, no pueden mapear múltiples modificaciones simultáneamente y son propensos a errores.
Namasivayam, Hiroshi Sugiyama y sus colegas de la Universidad de Kyoto probaron y encontraron dos enfoques que pueden distinguir con relativo éxito una modificación de ARN abundante y bien conocida que involucra el reemplazo de la base de nucleótidos uracilo con otra llamada pseudouridina.
Similar al ADN, el ARN está formado por una hebra de combinaciones variables de cuatro bases de nucleótidos diferentes:uracilo, citosina, adenina y guanina. La forma en que se organizan estas bases determina el código que indica qué proteína se debe producir. Cuando la pseudouridina reemplaza al uracilo en la columna vertebral del ARN, puede conducir a una mayor producción de proteínas o a cambiar el código de uno que señala la interrupción de la traducción de la información a uno que señala la formación de aminoácidos.
El enfoque del equipo implica el uso de una plataforma de secuenciación directa de ARN ya disponible desarrollada por Oxford Nanopore Technologies. En esta plataforma, las hebras de ARN pasan a través de pequeños poros en una membrana. Se producen interrupciones en la corriente que se mueve a través de la membrana según el orden de las diferentes bases de ARN. Esto permite a los científicos "leer" la secuencia. Pero a los científicos que utilizan este enfoque a menudo les resulta difícil distinguir los diferentes tipos de modificaciones entre sí.
Shubham Mishra, primer autor conjunto de este estudio, desarrolló algoritmos para identificar una alta probabilidad de existencia de una sustitución de pseudouridina en comparación con la posibilidad de que fuera un tipo diferente de cambio de base.
Una de sus estrategias compara series cortas de ARN de cinco bases de nucleótidos en las que el uracilo, la pseudouridina o la citosina están rodeados por ambos lados por las mismas bases. Luego, las lecturas pasan por algoritmos que calculan la probabilidad de que la base del medio sea una de las tres. Usaron su estrategia, llamada Indo-Compare (Indo-C), en secuencias de ARN modificadas y luego en levadura y ARN humano y descubrieron que era bueno para distinguir las sustituciones de pseudouridina de las demás.
También pudieron identificar sustituciones de pseudouridina mezclando una sonda química con muestras de ARN, que luego se adhiere selectivamente a ellas. Esto cambió las lecturas de secuencia de una manera que identifica la modificación.
"Creemos que nuestro trabajo hará que los métodos basados en secuenciación de nanoporos sean menos laboriosos para detectar modificaciones de ARN y más capaces de caracterizar los impactos de estas modificaciones en el desarrollo y la enfermedad", dice Namasivayam.
A continuación, el equipo tiene como objetivo optimizar el uso de ambos enfoques juntos para identificar con mayor precisión las modificaciones de ARN y ADN. Esto implicará la fabricación de nuevas sondas químicas que correspondan a cambios específicos. También planean seguir desarrollando algoritmos avanzados de aprendizaje automático que complementen los enfoques de secuenciación directa de ARN basados en sondas químicas. Secuenciación simultánea de múltiples modificaciones de bases de ARN:una nueva era en la biología del ARN