Gráficamente abstracto. Crédito:célula molecular (2022). DOI:10.1016/j.molcel.2022.06.023
Utilizando una técnica nueva e innovadora, los científicos de la Facultad de Medicina de Duke-NUS y sus colaboradores han identificado miles de secuencias de ADN previamente desconocidas en el genoma humano que codifican microproteínas y péptidos potencialmente críticos para la salud y las enfermedades humanas.
"Gran parte de lo que entendemos sobre el 2 % conocido del genoma que codifica proteínas proviene de la búsqueda de cadenas largas de secuencias de nucleótidos que codifican proteínas, o marcos de lectura largos y abiertos", explicó la bióloga computacional Dra. Sonia Chothani, investigadora del Programa de Trastornos Cardiovasculares y Metabólicos (CVMD) de Duke-NUS y primer autor del estudio. "Recientemente, sin embargo, los científicos han descubierto pequeños marcos de lectura abiertos (smORF) que también pueden traducirse del ARN a pequeños péptidos, que tienen funciones en la reparación del ADN, la formación de músculos y la regulación genética".
Los científicos han estado tratando de identificar los smORF y los pequeños péptidos que codifican, ya que la interrupción de estos smORF puede causar enfermedades. Sin embargo, los enfoques disponibles actualmente son muy limitados.
"Gran parte de los conjuntos de datos actuales no brindan información lo suficientemente detallada como para identificar smORF en el ARN", agregó el Dr. Chothani. "La mayoría también proviene de análisis de células humanas inmortalizadas que se propagan, a veces durante décadas, para estudiar la fisiología, la función y la enfermedad de las células. Sin embargo, estas líneas celulares no siempre son representaciones precisas de la fisiología humana".
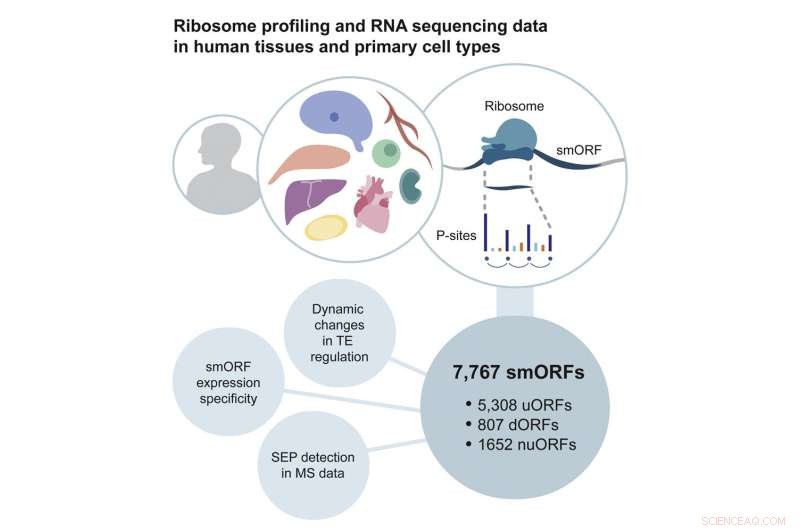
Publicación en Molecular Cell , Chothani y sus colegas en Singapur, Alemania, el Reino Unido y Australia describen una metodología que desarrollaron para abordar estos problemas. Examinaron los conjuntos de datos de perfiles de ribosomas actualmente disponibles en busca de cadenas cortas de ARN con secciones periódicas de tres bases, que cubren más del 60% de la longitud del ARN. Luego realizaron su propia secuenciación de ARN y perfiles de ribosomas para generar un recurso de datos combinados de seis tipos de células y cinco tipos de tejido, como del corazón y el cerebro, derivados de cientos de pacientes.
Los análisis de estos datos identificaron casi 8000 smORF. Curiosamente, eran muy específicos de los tejidos en los que se encontraron, lo que significa que estos smORF pueden realizar una función específica para su entorno. El equipo también identificó 603 microproteínas codificadas por algunos de estos smORF.
"El genoma está plagado de smORF", dijo el profesor asistente Owen Rackham, autor principal del estudio del Programa CVMD. "Nuestro mapa integral y espacialmente resuelto de smORF humanos destaca los componentes funcionales del genoma que se pasan por alto, señala nuevos actores en la salud y la enfermedad y proporciona un recurso para la comunidad científica como plataforma para acelerar los descubrimientos".
El profesor Patrick Casey, vicedecano senior de investigación de Duke-NUS, dijo:"Con el sistema de atención médica en evolución no solo para tratar enfermedades sino también para prevenirlas, la identificación de nuevos objetivos potenciales para la investigación de enfermedades y el desarrollo de fármacos podría abrir caminos a nuevas soluciones". Esta investigación de la Dra. Chothani y su equipo, publicada como un recurso para la comunidad científica, aporta información importante al campo". Encontrar los genes más pequeños podría generar grandes beneficios