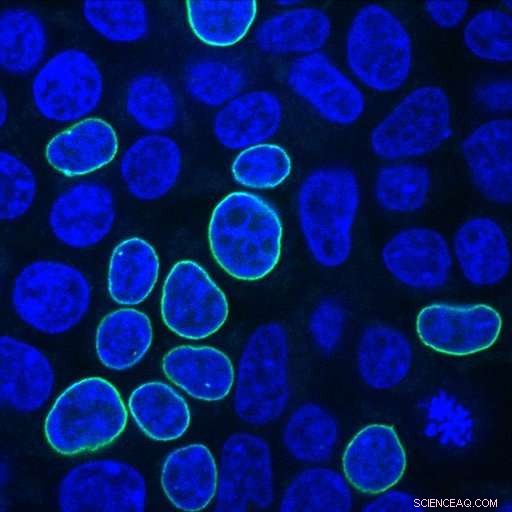
Las células renales embrionarias humanas brillan en verde después de la reparación de una rotura de ADN inducida por CRISPR con un fragmento de PCR que codifica una proteína fluorescente y brazos de homología con 33 nucleótidos. Crédito:Alexandre Paix
Los científicos de Johns Hopkins han desarrollado un método simplificado y "reglas" de eficiencia que lo acompañan para introducir nuevas secuencias de ADN en las células después de utilizar la herramienta de corte de genes conocida como CRISPR. Los científicos dicen que el método que se basaron en pruebas con embriones de ratón y miles de células humanas, podría mejorar la coherencia y la eficiencia de la edición del genoma.
El nuevo método y su desarrollo se describen en línea en el 28 de noviembre en el procedimientos de la Academia Nacional de Ciencias .
"CRISPR es una herramienta para ayudar a los científicos a modificar el genoma, predecir el resultado de ciertos rasgos y estudiarlos, pero la herramienta en sí solo crea rupturas en el genoma. No controla cómo se inserta una nueva secuencia de ADN en el genoma, "dice Geraldine Seydoux, Doctor., el profesor Huntington Sheldon de Descubrimiento Médico en el Departamento de Biología Molecular y Genética y vicedecano de investigación básica en la Facultad de Medicina de la Universidad Johns Hopkins, e investigador del Instituto Médico Howard Hughes.
"Nos propusimos estudiar cómo las células reparan las roturas inducidas por CRISPR con el objetivo de utilizar el proceso de reparación del ADN natural de la célula para introducir nuevas secuencias en el genoma. Nos sorprendió descubrir que las células copiarían fácilmente secuencias de ADN extraño para reparar las roturas del ADN. , siempre que los ADN extraños sean lineales, "Agrega Seydoux." Al estudiar cómo se copian los fragmentos de ADN extraños durante el proceso de reparación, Creamos algunas reglas simples para hacer que la edición del genoma sea lo más eficiente posible, optimizar la herramienta, y hazlo con confianza ".
CRISPR, que significa repetición palindrómica corta agrupada regularmente interespaciada, ha ganado popularidad entre los científicos en los últimos cinco años como una herramienta para cortar ADN de manera eficiente. Fue adaptado para su uso en células de mamíferos a partir de un proceso de defensa viral natural en células bacterianas que implica la creación de cortes letales en el ADN viral. Esencialmente, la herramienta es un conjunto simplificado de "tijeras" moleculares.
La creencia predominante entre los científicos, es que las células reparan las roturas del ADN insertando un conjunto aleatorio de nucleótidos, los componentes químicos del ADN. Esto generalmente destruye cualquier gen que se encuentre en el lugar donde se rompe el ADN.
También es bien sabido por los científicos que, de vez en cuando, las células utilizan una fuente diferente:una secuencia de otro fragmento de ADN, o ADN "donante":para sellar la ruptura del ADN. Sin embargo, la nueva secuencia "donante" no puede insertarse por sí sola en un espacio vacío del genoma.
En lugar de, el ADN del nuevo donante necesita una especie de cinta en cada extremo para ayudarlo a adherirse al espacio abierto por el corte. Los científicos se refieren a esta cinta como los brazos de "homología" del ADN del donante.
Los brazos de homología consisten en nucleótidos que se superponen a las porciones intactas del ADN con el código genético correspondiente. Esto ayuda a que el ADN del donante "se adhiera" al ADN intacto.
Todavía, Los científicos consideraron que el uso de ADN de un donante era una forma ineficaz de reparar el genoma. asumiendo que requirió largos brazos de homología, especialmente al insertar una secuencia de ADN larga, y ADN monocatenario o circular, que son difíciles de preparar en tamaños largos.
A medida que los científicos adquirieron más experiencia con CRISPR, Seydoux dice:"Surgieron preguntas sobre las reglas de diseño óptimas para el ADN del donante y la longitud de los brazos de homología".
Buscando respuestas a estas preguntas, los científicos de Johns Hopkins insertaron varias combinaciones de ADN de donantes en células renales embrionarias humanas, conocidos por su capacidad para crecer bien y por su uso frecuente en la investigación del cáncer. Los científicos utilizaron ADN de un donante con un gen que codifica una proteína fluorescente, que se ilumina en verde en la membrana nuclear de la célula cuando la inserción del gen es exitosa.
El investigador asociado de Johns Hopkins, Alexandre Paix, descubrió que los fragmentos lineales de ADN funcionan muy bien como donantes, y son de dos a cinco veces más eficientes que los ADN circulares (conocidos como plásmidos) en las células humanas. "El ADN lineal es muy fácil de preparar en el laboratorio, usando PCR, "dice Paix, refiriéndose a las herramientas de reacción en cadena de la polimerasa, que se utilizan para amplificar el ADN.
Paix también probó varias longitudes de brazos de homología. Encontró que el punto óptimo para los brazos de homología es de unos 35 nucleótidos de longitud, mucho más corto de lo que suelen utilizar los científicos.
Específicamente, Se encontró que los brazos de homología de 33 a 38 nucleótidos de longitud eran tan exitosos como los de 518 nucleótidos, produciendo entre un 10 y un 20 por ciento de ediciones exitosas en condiciones óptimas. A diferencia de, cuando los científicos probaron brazos de homología de 15 y 16 nucleótidos de longitud, las tasas de éxito de la inserción se redujeron a la mitad. Repitieron estos resultados en tres lugares diferentes del genoma humano.
También encontraron que la secuencia recién insertada, sin contar los brazos de homología, puede ser hasta 1, 000 nucleótidos de longitud.
El equipo logró tasas de éxito de entre el 10 y el 50 por ciento con inserciones de entre 57 y 993 nucleótidos de longitud. Las secuencias más cortas se insertaron con más éxito que las más largas. Por ejemplo, nuevas secuencias que eran 57, 714 y 993 nucleótidos de largo se insertaron con éxito 45,4, 23,5 y 17,9 por ciento del tiempo, respectivamente. Más allá de 1, 000 nucleótidos, nuevas inserciones con 1, 122 y 2, 229 nucleótidos tuvieron poco éxito, alrededor del 0,5 por ciento de las veces. "En ese tamaño, resulta muy difícil introducir la cantidad de ADN del donante necesaria para la edición. Las células tienden a 'ahogarse' con tanto ADN, "dice Seydoux.
Finalmente, el equipo también encontró que la tasa de éxito de la edición alcanza su punto máximo cuando la nueva secuencia se coloca dentro de los 30 nucleótidos del sitio de corte CRISPR. "Más allá de 30 nucleótidos, la inserción no es viable, "dice Seydoux.
"Estos parámetros deberían adaptarse a la mayoría de los genes que los científicos buscan editar. De hecho, la mayoría de los experimentos implican editar solo dos o tres nucleótidos cerca del sitio de corte CRISPR, "agrega Seydoux.
El equipo de investigación también probó si el mismo enfoque podría funcionar en embriones de ratón. Usando un fragmento de PCR con brazos de homología de 36 nucleótidos, el equipo insertó con éxito una secuencia de 739 nucleótidos de longitud que codificaba una proteína fluorescente en 27 de 87 (31 por ciento) embriones de ratón.
El equipo de investigación de Seydoux ya está utilizando las reglas de reparación para estudiar el ADN en Caenorhabditis elegans, una especie de gusano, y los investigadores están estudiando si las reglas de reparación se aplican a otros tipos de células humanas.
Antes de que las directrices sean ampliamente adoptadas, Seydoux dice que deberían probarse en más tipos de células humanas y otros organismos.