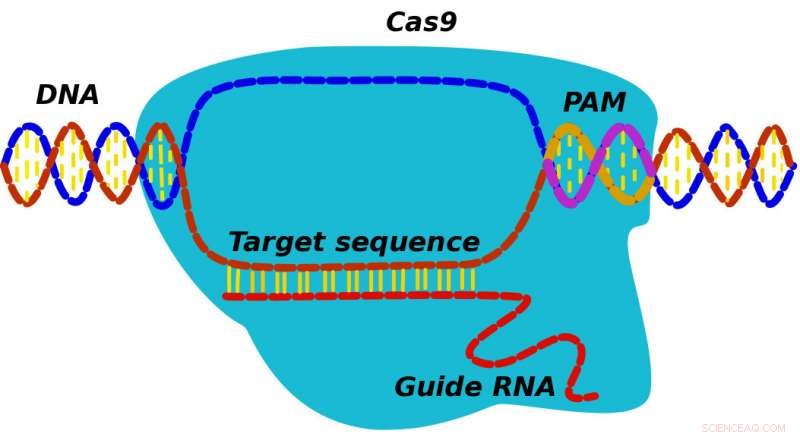
Cuando una proteína CRISPR-Cas9 encuentra su objetivo, primero encuentra una secuencia espaciadora conocida como PAM y luego busca el ADN adyacente para ver si coincide con el ARN guía de Cas9. Un nuevo modelo desarrollado por investigadores de la Universidad de Rice podría ayudar a descubrir detalles sobre el mecanismo por el cual CRISPR-Cas9 puede reemplazar mutaciones con ADN correcto. Crédito:Alexey Shvets / Rice University
Los investigadores de la Universidad de Rice han desarrollado un modelo computacional para cuantificar el mecanismo por el cual las proteínas CRISPR-Cas9 encuentran sus objetivos de edición del genoma.
Anatoly Kolomeisky, un profesor de Rice de química e ingeniería química y biomolecular, y el alumno Alexey Shvets adaptaron un sistema que desarrollaron anteriormente para mostrar cómo las proteínas generalmente encuentran sus objetivos biológicos. Esperan que el modelo revisado ayude a desvelar los misterios restantes de CRISPR.
En su estado natural, CRISPR, que significa "repeticiones palindrómicas cortas agrupadas regularmente interespaciadas, Es el mecanismo biológico por el cual las bacterias se protegen de las infecciones virales. Las bacterias incorporan una copia del ADN extraño y construyen un registro de todas las que invaden. Se refieren a ese registro cuando se detectan nuevos invasores y lo utilizan para destruirlos.
En años recientes, Los investigadores han comenzado a adaptar el mecanismo para su uso en la edición del genoma, que tiene el potencial de curar enfermedades y mejorar los organismos, incluidos los humanos. Pero un obstáculo ha sido el riesgo de que las proteínas CRISPR-Cas9, uno de los sistemas que utilizan el enfoque CRISPR, cortará y reemplazará las secuencias de destino incorrectas, introduciendo mutaciones.
El modelo de Rice descrito en el Revista biofísica descubrió que es probable que CRISPR-Cas9 localice buenos objetivos de manera más eficiente cuando se permite que se produzcan estas ediciones fuera del objetivo, porque las proteínas no pierden el tiempo en disociarse de los objetivos externos para seguir buscando.
Eso puede ser bueno o no, pero sin duda merece la pena estudiarlo, Dijo Kolomeisky.
"La tasa de error (corte fuera del objetivo) es a veces del 10 al 20 por ciento, ", dijo." Tenemos dos ideas sobre esto:una es que los virus mutan muy rápidamente y tal vez las bacterias están tratando de cortar objetivos que sólo están levemente mutados como una forma de ser más flexibles. La otra es que hay proteínas que pueden corregir errores, así que si no hay muchos cortes incorrectos, el sistema puede tolerarlos.
Kolomeisky dijo que su modelo es un simple paso hacia el descubrimiento de la dinámica de la edición CRISPR. "CRISPR-Cas9 es la variación más popular porque tiene una sola proteína y es más fácil, biológicamente, trabajar con, " él dijo.
El laboratorio de Rice desarrolló su modelo original para aprender cómo las proteínas se deslizan a lo largo del ADN para encontrar objetivos y desencadenar procesos como la transcripción de genes. Kolomeisky señaló que la pionera de CRISPR, Jennifer Doudna, descubrió que CRISPR-Cas9 no busca de la misma manera. "Descubrió que no se desliza en ninguna parte del ADN, " él dijo.
En lugar de, según Doudna y su equipo, la proteína reconoce inicialmente secuencias PAM de tres nucleótidos (para motivo adyacente al protoespaciador) que marcan la ubicación de los objetivos potenciales. "CRISPR encuentra y se une a PAM y luego su ARN asociado explora el ADN adyacente para ver si este es el objetivo, "Dijo Kolomeisky." Si lo es, la proteína comienza a cortarse. Que no, se disocia y busca en otra parte ".
En los experimentos posteriores de Doudna con las secuencias PAM eliminadas, Las proteínas CRISPR-Cas9 no pudieron encontrar sus objetivos en absoluto. Por lo tanto, los PAM tienen un papel importante y no son solo un espaciador genérico, él dijo. "Tan pronto como leo esto, Comprendí que también podríamos usar nuestro modelo aquí ".
El modelo teórico analiza los procesos de primer paso:aquellos que suceden cuando un sistema cruza un umbral físico o químico, como encontrar un PAM relevante, para rastrear las proteínas CRISPR-Cas9 insertadas en una célula mientras examinan primero las secuencias de PAM y luego, mientras está vinculado a PAM, busque el objetivo de ADN que coincida con el ARN de Cas9.
Descubrieron que los CRISPR que evitan los cortes fuera del objetivo al disociarse del ADN "incorrecto" tardan más en asentarse que uno que simplemente corta el objetivo. "Ir al PAM incorrecto lleva tiempo, "Dijo Kolomeisky." Nuestro cálculo muestra que CRISPR puede encontrar objetivos reales más rápido cuando a veces corta en los lugares equivocados. La fracción que va a los objetivos correctos puede ser menor, pero eventualmente los cortarás.
"Es un modelo simple y con una solución exacta, "Dijo Kolomeisky." Si alguien quiere hacer una prueba, el modelo puede proporcionar predicciones específicas y, en algunos casos, ofrecer tendencias de lo que se debe observar ". Lo que falta en el modelo es la capacidad de ver si la clave de ARN reconoce su objetivo simultáneamente, uniéndose al ADN de una vez, o secuencialmente, nucleótido por nucleótido.
"Lo más impresionante de CRISPR no es el descubrimiento de un sistema inmunológico en las bacterias, sino el hecho de que ha creado una revolución en la biotecnología. porque significa que en cualquier célula podemos cortar cualquier ADN en una ubicación específica, muy precisamente, "Dijo Kolomeisky." Espero que nuestro trabajo estimule más estudios fundamentales, porque me gusta mucho el método CRISPR. Pero no estoy contento cuando la gente lo aplica sin entender cómo funciona a nivel molecular ".
Shvets es ahora investigador postdoctoral en el Instituto de Tecnología de Massachusetts. Kolomeisky es profesor de química y de ingeniería química y biomolecular.