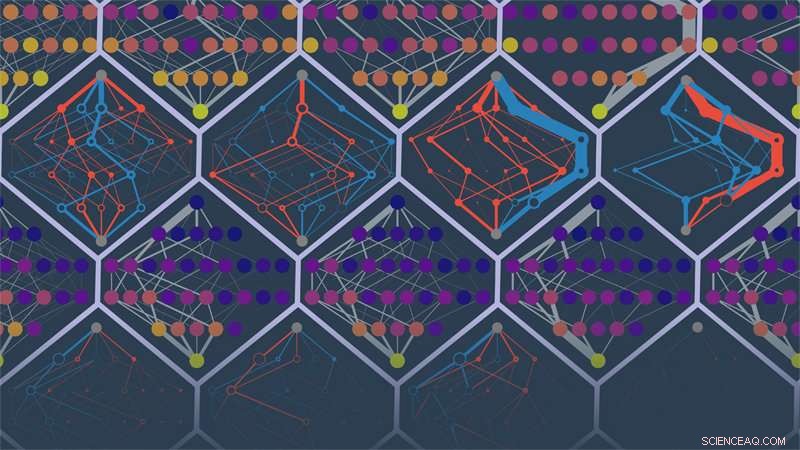
Los investigadores del laboratorio de Michael Harms de la Universidad de Oregon predijeron los caminos evolutivos de diferentes proteínas simuladas. Los puntos de la imagen son secuencias de proteínas. Los bordes denotan probabilidades de trayectorias. Los colores indican una condición física de baja a alta (púrpura a amarillo) o rutas falsamente inaccesibles o falsamente accesibles (rojo y azul). Crédito:Michael Harms y Zach Sailer
Los científicos de la Universidad de Oregón teorizaron que podrían manipular una proteína una mutación a la vez y predecir su evolución. Intentaron probarlo. Y falló.
Ellos piensan sin embargo, que han encontrado una verdad fundamental que subyace a la imprevisibilidad en un sistema biológico. Las limitaciones físicas básicas hacen que la incertidumbre sea la norma, informaron en un artículo publicado en línea el 23 de octubre en el procedimientos de la Academia Nacional de Ciencias .
"Si bien obtuvimos un resultado negativo sorprendente, pudimos decir por qué, "dijo Michael J. Harms, profesor del Departamento de Química y Bioquímica de la UO y científico del Instituto de Biología Molecular. "Eso es positivo. Nuestro simple estudio proporciona la confirmación de lo que muchas personas en el campo han observado repetidamente:la imprevisibilidad. Parece que es universal".
La investigación fue un asunto digital, realizado con simulaciones por ordenador diseñadas por el estudiante de doctorado de la UO Zachary R. Sailer. Él y Harms crearon una proteína de celosía simple, utilizando un enfoque creado previamente en el laboratorio de Harms, con una secuencia aleatoria de 12 aminoácidos. Luego ejecutaron simulaciones evolutivas para optimizar la estabilidad, una propiedad física de la proteína.
El objetivo era utilizar los efectos de las 228 mutaciones que se sabe están asociadas con la proteína inicial para predecir estas trayectorias simuladas:qué mutación ocurriría, cuando, tiempo extraordinario. La capacidad de proyectar hacia adelante se desvaneció rápidamente después de las dos primeras mutaciones. Después, las trayectorias anticipadas se desviaron en medio de un número creciente de probabilidades de desvío.
"La calidad de su información en realidad decae con el tiempo, ", Dijo Sailer." A medida que se acumulan las mutaciones, los efectos de las mutaciones que midió comienzan a cambiar, por lo que no puede predecir hacia dónde se dirige ".
En su papel Sailer y Harms sugieren que la física, particularmente termodinámica, está en juego. Cada mutación altera la proteína en una pequeña, pero de forma no lineal. Esto significa que el efecto de cada mutación depende de todas las mutaciones que ocurrieron antes.
"Creo que lo que mostramos, fundamentalmente, es que aunque sepa mucho sobre un sistema, sobre una proteína, no se puede predecir cómo evoluciona debido a la física del sistema, "Hay reglas físicas que limitan la evolución y su previsibilidad", dijo Harms.
Cómo evolucionan las proteínas es una cuestión fundamental en la biología evolutiva, tanto desde una perspectiva filosófica, para aprender más sobre la maquinaria de los sistemas biológicos, y en busca de pistas que puedan conducir a medicamentos mejorados o mejores.
"Prácticamente, "Harms dijo, "Nuestra investigación puede ayudarnos a aprender cómo prevenir la evolución de la resistencia a los antibióticos en las bacterias". Casi todas las infecciones transmitidas por bacterias están desarrollando resistencia a los antibióticos, creando una preocupación de salud pública líder en todo el mundo.
"En lugar de estudiar los efectos de todas las mutaciones individuales, "Sailer agregó, "Quizás deberíamos estudiar combinaciones aleatorias de muchas mutaciones. Tal enfoque podría ayudarnos a predecir la evolución de la resistencia".
Se está trabajando en el laboratorio de Harms para probar esta posibilidad en proteínas reales. "Estamos creando herramientas computacionales que nos permiten analizar conjuntos de datos de resistencia a los antibióticos, y recibimos indicios de que un enfoque combinatorio funciona, "Dijo Harms." Es más complicado que estudiar mutaciones individuales, pero nuestro trabajo muestra que es poco probable que el enfoque individual funcione ".