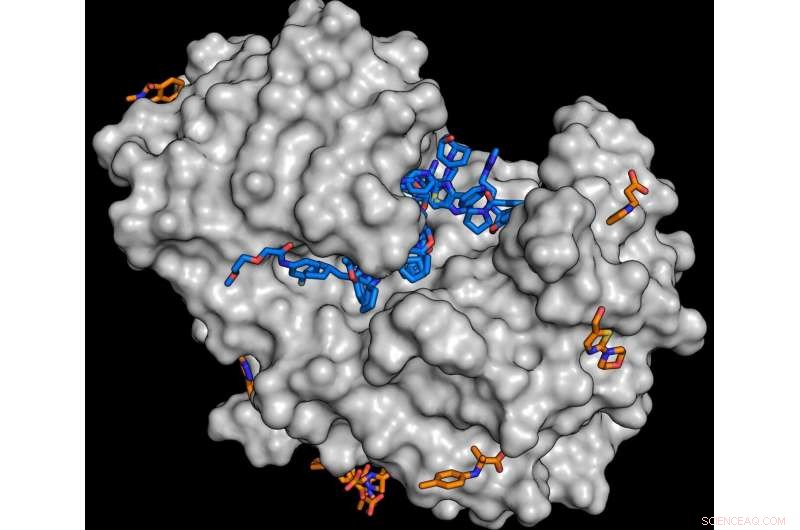
Para el estudio, la enzima endotiapepsina (gris) se combinó con moléculas de la biblioteca de fragmentos. El análisis muestra que numerosas sustancias pueden acoplarse a la enzima (moléculas azul y naranja). Cada sustancia encontrada es un punto de partida potencial para el desarrollo de moléculas más grandes. Crédito:J. Wollenhaupt / HZB
Para acelerar el desarrollo sistemático de medicamentos, el equipo MX de Helmholtz-Zentrum Berlin (HZB) y el Grupo de Diseño de Fármacos de la Universidad de Marburg han establecido una nueva biblioteca de sustancias. Consiste en 1103 moléculas orgánicas que podrían usarse como bloques de construcción para nuevos medicamentos. El equipo MX ahora ha validado esta biblioteca en colaboración con el grupo FragMAX en MAX IV. La biblioteca de sustancias del HZB está disponible para la investigación en todo el mundo y también desempeña un papel en la búsqueda de sustancias activas contra el SARS-CoV-19.
Para que las drogas sean efectivas, habitualmente deben acoplarse a las proteínas en el organismo. Como una llave en una cerradura parte de la molécula del fármaco debe encajar en los huecos o cavidades de la proteína diana. Desde hace varios años, Por lo tanto, el equipo del Departamento de Cristalografía Macromolecular (MX) en HZB dirigido por el Dr. Manfred Weiss junto con el Grupo de Diseño de Medicamentos dirigido por el Prof. Gerhard Klebe (Universidad de Marburg) ha estado trabajando en la construcción de lo que se conoce como bibliotecas de fragmentos. Estos consisten en pequeñas moléculas orgánicas (fragmentos) con las que se pueden sondear y mapear las cavidades funcionalmente importantes en la superficie de las proteínas. Los cristales de proteína se saturan con los fragmentos y luego se analizan con una potente luz de rayos X. Esto permite obtener información estructural tridimensional a niveles de resolución atómica. Entre otras cosas, es posible averiguar qué tan bien se acopla un fragmento de molécula específico a la proteína diana. El desarrollo de estas bibliotecas de sustancias se llevó a cabo como parte del proyecto de investigación conjunto Frag4Lead y fue financiado por el Ministerio Federal de Educación e Investigación de Alemania (BMBF).
El equipo MX (MX significa Cristalografía Macromolecular) ha publicado ahora el diseño de una biblioteca de fragmentos químicamente diversos llamada biblioteca "F2X-Universal", que consta de 1, 103 compuestos. Se ha extraído una selección representativa de 96 compuestos de esta biblioteca, que se conoce como la pantalla de entrada F2X. En el curso de la publicación de la biblioteca, esta selección ha sido probada y validada con éxito por el equipo MX del HZB en la fuente de rayos X MAX IV en Lund, Suecia y en BESSY II.
En el estudio, Los equipos de HZB y MAX IV verificaron la eficacia de la biblioteca de entrada F2X mediante el cribado de endotiapepsina y el complejo proteico Aar2 / RnaseH como enzimas diana. En el siguiente paso, el equipo MX utilizará toda la biblioteca universal.
"Para el estudio actual, los expertos en detección de fragmentos de HZB — BESSY II trabajaron muy de cerca con el equipo del proyecto FragMAX en MAX IV ", dijo el Dr. Uwe Müller del equipo MX en HZB, quien ayudó a configurar las tres líneas de luz MX en BESSY II, así como la línea de luz BioMAX en MAX IV. "Esto permitió a ambos socios desarrollar aún más sus propias plataformas tecnológicas y utilizarlas para obtener imágenes de las superficies funcionales de diferentes proteínas. Esta será una base excelente para la futura colaboración entre MAX IV y HZB".