Los componentes moleculares de las computadoras podrían representar una nueva revolución informática y ayudarnos a crear computadoras más baratas, más rápidas, más pequeñas y más potentes. Sin embargo, los investigadores luchan por encontrar formas de ensamblarlos de manera más confiable y eficiente.
Para ayudar a lograrlo, científicos del Instituto de Física de la Academia Checa de Ciencias investigaron las posibilidades del autoensamblaje de máquinas moleculares basándose en soluciones perfeccionadas por la evolución natural y utilizando sinergia con la fabricación de chips actual.
Existe un límite para la miniaturización de los actuales chips informáticos basados en silicio. La electrónica molecular, que utiliza interruptores y memorias del tamaño de una sola molécula, podría proporcionar una revolución en el tamaño, la velocidad y las capacidades de las computadoras y al mismo tiempo reducir su creciente consumo de energía, pero su producción en masa es un desafío. La nanofabricación y el ensamblaje de los componentes a gran escala, con pocos defectos y accesibles siguen siendo difíciles de alcanzar. La inspiración extraída de la naturaleza viva podría cambiar este status quo.
Actualmente se están produciendo pequeños prototipos de circuitos moleculares compuestos por un par de moléculas mediante microscopía de sonda de barrido, que los manipula una molécula a la vez mediante un voladizo macroscópico lento y pesado.
Prokop Hapala, quien dirigió el estudio publicado en ACS Nano , lo compara con la construcción de un delicado mosaico utilizando una enorme grúa, un azulejo a la vez. El autoensamblaje podría resolver este problema, pero crea otros desafíos. Por ejemplo, ¿cómo producimos una variedad de estructuras cuando sólo una pequeña cantidad de información estructural puede codificarse en interacciones entre unos pocos grupos funcionales?
Los investigadores del Instituto de Física de la Academia Checa de Ciencias se inspiraron en la naturaleza, donde los componentes funcionales y estructurales están desacoplados en plantillas poliméricas como el ADN o el ARN. Allí, los azúcares-fosfato representan el andamiaje y las nucleobases, unidas por enlaces de hidrógeno, proporcionan el almacenamiento de información.
Gracias a estos enlaces, estos polímeros informativos pueden autoensamblarse en formas complejas e impulsar la autorreplicación o la síntesis de otras moléculas más pequeñas. Este enfoque ya se ha utilizado en el "origami de ADN", que puede producir moléculas complejas con las formas y funciones deseadas. Pero, ¿cómo podemos ampliar el proceso y lograr una mayor variedad?
"Los pares de bases de ADN conocidos, que ingenuamente se podría pensar que serían la mejor opción, no se pueden utilizar tal como están", explica Paolo Nicolini, uno de los autores. "Funcionan muy bien en la célula, pero esto se debe al entorno y al resto de la maquinaria celular. En condiciones compatibles con la nanofabricación, simplemente no son lo suficientemente selectivos".
Mithun Manikandan, Paolo Nicolini y Prokop Hapala decidieron combinar las posibilidades que ofrecen el origami de ADN y la fotolitografía para diseñar estructuras complejas de chips contemporáneos. Esto podría allanar el camino para la producción en masa de circuitos moleculares revolucionarios integrados con la tecnología contemporánea de fabricación de chips, algo que podría permitir una transición sin problemas de la maquinaria informática actual al siguiente nivel.
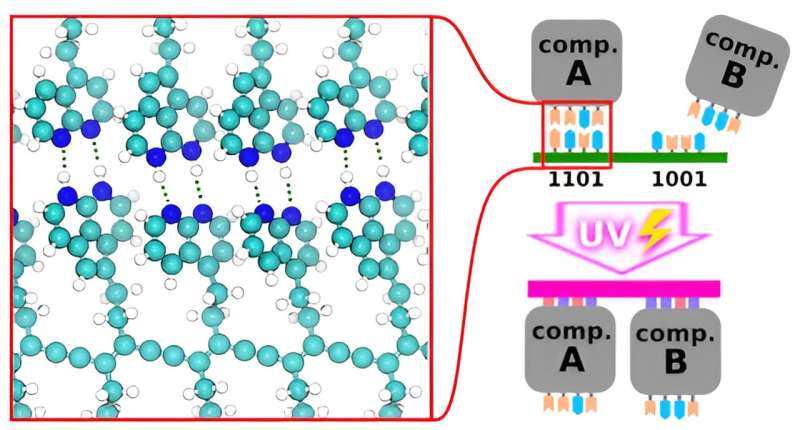
Para permitir esto, los investigadores propusieron reemplazar la columna vertebral de azúcar-fosfatos con diacetileno fotosensible. Utilizaron simulaciones detalladas para detectar grupos finales complementarios unidos por enlaces de hidrógeno que impulsarían el autoensamblaje en una red en las condiciones utilizadas en la producción de chips.
Se utilizaron derivados de diacetileno como columna vertebral porque pueden polimerizarse eficientemente en estas condiciones cuando se ceban con luz ultravioleta o inyección de electrones, y se investigaron in silico unidades análogas a las bases de ADN/ARN (las "letras" del código genético) como grupos terminales. conducir el conjunto de componentes a las formas previstas.
El objetivo era encontrar pares complementarios, donde dos unidades se unen de manera confiable entre sí y no con otras unidades; este rasgo, nuevamente análogo a cómo funciona el ADN, permitiría la creación de patrones de circuitos complejos deterministas. Los investigadores descubrieron que las unidades que contienen grupos terminales donantes de hidrógeno puro eran especialmente adecuadas. Se encontraron dieciséis unidades candidatas prometedoras, lo que allanó el camino para la investigación experimental y eventuales aplicaciones industriales.
Los resultados tienen implicaciones interesantes para la computación del ADN y los análogos artificiales del ADN. Los alfabetos de cuatro letras más viables encontrados en la selección se produjeron en una región muy estrecha de energías de unión de 15 a 25 kcal/mol, y todos dependían de un pequeño subconjunto de los grupos finales analizados.
Aunque sólo se pudo probar con alta precisión un pequeño subconjunto del posible espacio de letras, esto sugiere que el alfabeto de ADN puede no ser simplemente el resultado de un "accidente congelado en el tiempo", sino que podría haber sido una opción estable y energéticamente favorable. No se encontraron alfabetos de seis letras en el espacio probado, pero nuevos mecanismos de selectividad y enlaces no covalentes distintos de los enlaces de hidrógeno (como los enlaces de halógeno) podrían habilitarlos. De manera similar, se podrían probar las posibilidades de los análogos de ADN terapéuticos y farmacéuticos.
Este trabajo mejorará aún más la disponibilidad sintética de las moléculas y superará las limitaciones experimentales. Si bien es probable que la mayoría de nosotros estemos leyendo esto en máquinas que dependen de transistores basados en silicio, pronto podríamos comenzar sin problemas a realizar una transición a máquinas que utilicen parcialmente nanoelectrónica molecular. Este trabajo representa un paso más hacia ese futuro.
Más información: Mithun Manikandan et al, Diseño computacional de plantillas de polímeros fotosensibles para impulsar la nanofabricación molecular, ACS Nano (2024). DOI:10.1021/acsnano.3c10575
Información de la revista: ACS Nano
Proporcionado por la Academia Checa de Ciencias