
Marianne Liebi, investigador postdoctoral en el grupo Coherent X-ray Scattering de PSI, Colocación de un pequeño trozo de hueso para nano imágenes tridimensionales. Crédito:Instituto Paul Scherrer / Markus Fischer
Los huesos están formados por pequeñas fibras que son aproximadamente mil veces más finas que un cabello humano. Una característica importante de estas llamadas fibrillas de colágeno es que están ordenadas y alineadas de manera diferente según la parte del hueso en la que se encuentran. Aunque este orden es decisivo para la estabilidad mecánica del hueso, La tomografía computarizada (TC) tradicional solo se puede utilizar para determinar la densidad, pero no la orientación local de la nanoestructura subyacente. Los investigadores del Instituto Paul Scherrer PSI han superado esta limitación gracias a un algoritmo innovador basado en computadora. Aplicaron el método a las mediciones de una pieza de hueso obtenida con el Swiss Light Source SLS. Su enfoque les permitió determinar el orden localizado y la alineación de las fibrillas de colágeno dentro del hueso en tres dimensiones. Aparte del hueso, el método se puede aplicar a una amplia variedad de muestras biológicas y de ciencias de los materiales.
Los investigadores publicaron el resultado de su estudio en la revista Naturaleza .
La disposición de la nanoestructura de un objeto tridimensional ahora se puede visualizar gracias a un nuevo método desarrollado por investigadores del Instituto Paul Scherrer PSI. Los investigadores demostraron este nuevo enfoque en colaboración con expertos en biomecánica ósea de ETH Zurich y la Universidad de Southampton. REINO UNIDO, utilizando un pequeño trozo de una vértebra humana que tenía aproximadamente dos milímetros y medio de largo. El hueso consta de fibras diminutas que se denominan fibrillas de colágeno. Su alineación y orden tridimensional local, que juega un papel central en la determinación de las propiedades mecánicas de un hueso, ahora se ha visualizado a lo largo de toda la pieza de hueso. Este nuevo enfoque de imágenes proporciona información importante que podría ayudar, por ejemplo, el estudio de enfermedades óseas degenerativas como la osteoporosis. En general, El nuevo método es adecuado no solo para examinar objetos biológicos sino también para desarrollar nuevos materiales prometedores.
Los datos se obtuvieron de Swiss Light Source SLS de PSI, donde se proyectó el trozo de hueso con un haz de rayos X extremadamente fino e intenso. Este rayo se escanea a través de la muestra, registro de datos punto por punto. La interacción de los rayos X con la muestra proporciona información sobre la nanoestructura local en cada punto de medición.
El paso crucial de 2D a 3D
Hasta ahora, de esta forma sólo se podían escanear y examinar muestras bidimensionales. Tradicionalmente, los objetos examinados se cortan así en rodajas muy finas. Pero no todos los objetos se pueden cortar tan finos como quisiera, explica el supervisor del proyecto Manuel Guizar-Sicairos. Y a veces cuando lo cortas destruyes o perturbas la propia nanoestructura que querías examinar. En general, es preferible un método no destructivo, dejando el objeto intacto para investigaciones posteriores.
Para poder obtener imágenes de objetos tridimensionales, los investigadores de PSI escanearon su muestra repetidamente, girándolo un pequeño ángulo entre cada escaneo. De esta manera, obtuvieron datos de medición para todas las orientaciones que les permitieron reconstruir posteriormente el objeto tridimensional, incluyendo su nanoestructura, en la computadora.

Manuel Guizar-Sicairos y Marianne Liebi trabajaron juntos para investigar un trozo de hueso y desarrollar el complejo algoritmo para la evaluación de sus datos. Crédito:Instituto Paul Scherrer / Markus Fischer
El nuevo método de medición utilizado por los investigadores de PSI se basa en un principio básico de la tomografía computarizada (TC). La TC también implica primero tomar muchas imágenes de rayos X de un paciente u objeto desde diferentes ángulos y luego combinarlas para formar las imágenes deseadas por medio de un cálculo por computadora. Sin embargo, La tomografía computarizada tradicional no utiliza un haz de rayos X fino. En lugar de, el objeto se irradia como un todo.
Si bien la tomografía computarizada puede representar la densidad variable del material, no captura detalles como el orden y la alineación de la nanoestructura subyacente. Esto último solo es posible a través de la medición precisa de la interacción entre la muestra y los rayos X que es posible gracias a la estrecha, haz de rayos X intenso del SLS junto con detectores de última generación.
Las imágenes surgen gracias a algoritmos matemáticos
El paso más complejo fue compilar una imagen de computadora de la muestra tridimensional a partir de la gran cantidad de datos. Para hacer esto, los investigadores desarrollaron su propio algoritmo matemático sofisticado. El haz de rayos X siempre penetra en toda la profundidad de la muestra y solo vemos el resultado final, explica Marianne Liebi, autor principal del estudio. Cómo se ve realmente la estructura tridimensional es algo que tenemos que averiguar más adelante.
Para cada punto del interior de la muestra, El algoritmo de Liebi busca la estructura que mejor se corresponda con todos los datos medidos. En el algoritmo, los investigadores aprovecharon que podían asumir una cierta simetría en la disposición de las fibrillas de colágeno en el hueso, reduciendo así sus datos a un nivel manejable. Sin embargo, aún quedaban por encontrar 2,2 millones de parámetros. Estos se optimizaron utilizando un programa de computadora que prueba cada vez mejores soluciones hasta que encuentra una que puede explicar mejor todas las mediciones.
Me asombró que después de tanta matemática pura, surgió una imagen que realmente parecía un hueso, dijo Liebi. Los detalles en él fueron plausibles de inmediato.
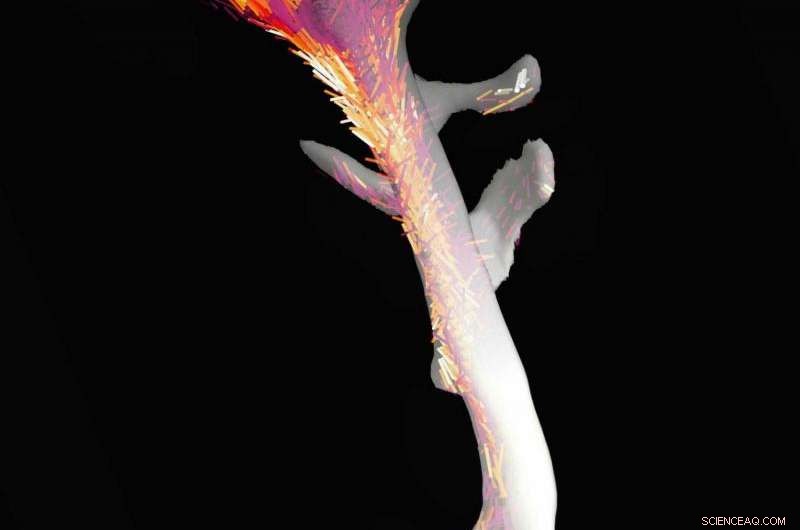
El hueso y su nanoestructura:gracias a su algoritmo recientemente desarrollado, Los investigadores de PSI lograron mapear el orden y la alineación de las diminutas fibrillas de colágeno en todo este fragmento de hueso de aproximadamente dos milímetros y medio de longitud. Crédito:Instituto Paul Scherrer / Marianne Liebi
Como un mapa de las zonas de vegetación
Mientras que la tomografía computarizada clásica genera imágenes en escala de grises, el nuevo método proporciona imágenes en color con mucha más información:los cilindros multicolores muestran la orientación en la nanoescala e incluso proporcionan información sobre el grado de orientación, que es alta si las fibrillas de colágeno adyacentes tienen todas la misma orientación y baja si están orientadas al azar.
No podemos obtener imágenes de cada fibrilla de colágeno individual directamente, pero eso no es necesario de todos modos, explica Guizar-Sicairos. Nuestra técnica de imágenes es similar a un mapa de zonas de vegetación. Ahí también, uno promedia en ciertas áreas, afirmando que una región está dominada por árboles coníferos, otro por árboles de hoja caduca y otro por bosque mixto. De este modo, es posible mapear la vegetación de continentes enteros sin tener que clasificar cada árbol.
Por analogía se puede decir que con los métodos microscópicos y nanoscópicos tradicionales esta representación de árboles individuales era necesaria. Por eso hasta ahora cuanto más pequeña era la estructura de un objeto, más pequeña también tenía que ser la sección de la imagen. Su nuevo método permitió a los investigadores de PSI sortear esta limitación:a partir de un trozo de hueso visible a simple vista, registraron la disposición de la nanoestructura en una sola imagen.
Al mismo tiempo que su publicación, Naturaleza contará con una segunda publicación con una investigación liderada por otro equipo de investigadores con Liebi y Guizar-Sicairos como coautores. Esa publicación presenta un algoritmo alternativo que conduce a un resultado similar:los investigadores pudieron determinar la nanoestructura interna tridimensional de un diente humano.