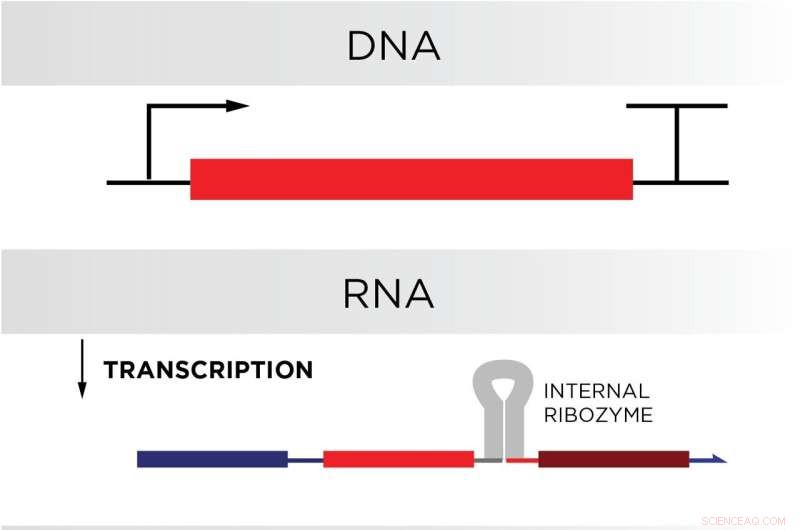
Los investigadores del NIST tienen como objetivo convertir la célula en una fábrica de computadoras biológicas mediante el diseño y la inserción de ADN en el genoma de una célula. Las proteínas celulares producirían ARN basado en el ADN a través de la transcripción. La hebra de ARN luego se plegaría, se uniría a sí misma y se dividiría en dos, gracias a una secuencia especial de ARN autoescindible llamada ribozima. La estructura resultante, una puerta de circuito de ARN, solo se desharía y desencadenaría más reacciones químicas bajo ciertas condiciones. Crédito:N. Hanacek/NIST
Diminutas computadoras biológicas hechas de ADN podrían revolucionar la forma en que diagnosticamos y tratamos una gran cantidad de enfermedades, una vez que la tecnología esté completamente desarrollada. Sin embargo, un obstáculo importante para estos dispositivos basados en ADN, que pueden operar tanto en células como en soluciones líquidas, ha sido su breve duración. Solo un uso y las computadoras están gastadas.
Ahora, los investigadores del Instituto Nacional de Estándares y Tecnología (NIST) pueden haber desarrollado computadoras biológicas de larga duración que podrían persistir dentro de las células. En un artículo publicado en la revista Science Advances , los autores renuncian al enfoque tradicional basado en el ADN y optan por utilizar el ácido nucleico ARN para construir computadoras. Los resultados demuestran que los circuitos de ARN son tan fiables y versátiles como sus equivalentes basados en ADN. Además, las células vivas pueden ser capaces de crear estos circuitos de ARN continuamente, algo que no es posible con los circuitos de ADN, posicionando aún más al ARN como un candidato prometedor para computadoras biológicas poderosas y duraderas.
Al igual que la computadora o el dispositivo inteligente en el que probablemente esté leyendo esto, las computadoras biológicas pueden programarse para realizar diferentes tipos de tareas.
"La diferencia es que, en lugar de codificar con unos y ceros, se escriben cadenas de A, T, C y G, que son las cuatro bases químicas que componen el ADN", dijo Samuel Schaffter, investigador postdoctoral del NIST y autor principal del estudio. .
Al ensamblar una secuencia específica de bases en una hebra de ácido nucleico, los investigadores pueden determinar a qué se une. Se puede diseñar una hebra para que se adhiera a partes específicas de ADN, ARN o algunas proteínas asociadas con una enfermedad, luego desencadene reacciones químicas con otras hebras en el mismo circuito para procesar información química y eventualmente producir algún tipo de resultado útil.
Esa salida podría ser una señal detectable que podría ayudar en el diagnóstico médico, o podría ser un fármaco terapéutico para tratar una enfermedad.
Sin embargo, el ADN no es el material más resistente y puede desmoronarse rápidamente en ciertas condiciones. Las células pueden ser entornos hostiles, ya que a menudo contienen proteínas que fragmentan los ácidos nucleicos. E incluso si las secuencias de ADN permanecen lo suficiente como para detectar su objetivo, los enlaces químicos que forman las vuelven inútiles después.
"No pueden hacer cosas como monitorear continuamente los patrones en la expresión génica. Son de un solo uso, lo que significa que solo te dan una instantánea", dijo Schaffter.
Al ser también un ácido nucleico, el ARN comparte muchos de los problemas del ADN cuando se trata de ser un bloque de construcción de una computadora biológica. Es susceptible a una degradación rápida, y después de que una hebra se une químicamente a una molécula objetivo, esa hebra se acaba. Pero a diferencia del ADN, el ARN podría ser un recurso renovable en las condiciones adecuadas. Para aprovechar esa ventaja, Schaffter y sus colegas primero tenían que demostrar que los circuitos de ARN, que las células teóricamente podrían producir, podían funcionar tan bien como los circuitos basados en ADN.
La ventaja del ARN sobre el ADN proviene de un proceso celular natural llamado transcripción, en el que las proteínas producen ARN de forma continua utilizando el ADN de una célula como plantilla. Si el ADN en el genoma de una célula codificara los componentes del circuito en una computadora biológica, entonces la célula produciría los componentes de la computadora continuamente.
En el proceso de computación biológica, las hebras individuales de ácidos nucleicos en un circuito pueden terminar fácilmente unidas a otras hebras en el mismo circuito, un efecto no deseado que evita que los componentes del circuito se unan a sus objetivos previstos. El diseño de estos circuitos a menudo significa que los diferentes componentes encajarán de forma natural entre sí.
Para evitar la unión no deseada, las secuencias de ADN que forman parte de las computadoras conocidas como circuitos de desplazamiento de hebras generalmente se sintetizan (en máquinas en lugar de células) por separado y en forma de doble hebra. Con cada base química en cada hebra unida a una base en la otra hebra, esta doble hebra actúa como una puerta cerrada que solo se desbloquearía si la secuencia objetivo apareciera y tomara el lugar de una de las hebras.
Schaffter y Elizabeth Strychalski, líder del Grupo de Ingeniería Celular del NIST y coautora del estudio, intentaron imitar esta función de "puerta bloqueada" en su circuito de ARN, teniendo en cuenta que, en última instancia, las células tendrían que producir estas puertas bloqueadas por sí mismas. Para preparar las células para el éxito, los investigadores escribieron las secuencias de modo que la mitad de las hebras pudieran unirse al ras con la otra mitad. Al unirse de esta manera, las secuencias de ARN se plegarían sobre sí mismas como un panecillo de perrito caliente, asegurándose de que estén en un estado bloqueado.
Pero para que funcionen correctamente, las puertas tendrían que ser dos hebras químicamente unidas pero distintas, más parecidas a un pan de hamburguesa o un sándwich que a un pan de perrito caliente. El equipo obtuvo el diseño de doble cadena en sus puertas codificando un tramo de ARN llamado ribozima cerca del punto de plegamiento de las puertas. Esta ribozima en particular, tomada del genoma de un virus de la hepatitis, se cortaría sola después de que la hebra de ARN en la que estaba incrustada se doblara, creando dos hebras separadas.
Los autores probaron si sus circuitos podían realizar operaciones lógicas básicas, como desbloquear sus puertas solo en escenarios específicos, como si una de dos secuencias de ARN específicas estuviera presente o solo si ambas estuvieran al mismo tiempo. También construyeron y examinaron circuitos hechos de varias puertas que realizaban diferentes operaciones lógicas en serie. Solo cuando estos circuitos encontraron la combinación correcta de secuencias, sus puertas se desbloquearían una por una como fichas de dominó.
Los experimentos involucraron la exposición de diferentes circuitos a piezas de ARN, algunos de los cuales, los circuitos fueron diseñados para unirse, y la medición de la salida de los circuitos. En este caso, la salida al final de cada circuito era una molécula indicadora fluorescente que se iluminaba una vez que se desbloqueaba la puerta final.
Los investigadores también rastrearon la velocidad a la que se abrieron las puertas a medida que los circuitos procesaban las entradas y compararon sus medidas con las predicciones de los modelos informáticos.
"Para mí, estos debían funcionar en un tubo de ensayo de forma tan predictiva como la computación de ADN. Lo bueno de los circuitos de ADN es que, la mayor parte del tiempo, puedes simplemente escribir una secuencia en una hoja de papel y funcionará de la manera quieres", dijo Schaffter. "La clave aquí es que descubrimos que los circuitos de ARN eran muy predecibles y programables, mucho más de lo que pensé que serían, en realidad".
Las similitudes en el rendimiento entre los circuitos de ADN y ARN podrían indicar que puede ser beneficioso cambiar a este último, ya que el ARN se puede transcribir para reponer los componentes de un circuito. Y muchos circuitos de ADN existentes que los investigadores ya han desarrollado para realizar diversas tareas teóricamente podrían intercambiarse por versiones de ARN y comportarse de la misma manera. Sin embargo, para estar seguros, los autores del estudio deben impulsar la tecnología aún más.
En este estudio, los autores demostraron que los circuitos transcribibles funcionan, pero aún no los han producido usando la maquinaria celular real de transcripción. En cambio, las máquinas sintetizaron los ácidos nucleicos a través de un proceso similar al que se usa para producir ADN para la investigación. Dar el siguiente paso requeriría insertar ADN en el genoma de un organismo, donde serviría como modelo para los componentes del circuito de ARN.
"Estamos interesados en poner esto en bacterias a continuación. Queremos saber:¿Podemos empaquetar diseños de circuitos en material genético usando nuestra estrategia? ¿Podemos obtener el mismo tipo de rendimiento y comportamiento cuando los circuitos están dentro de las células?" Schaffter dijo. "Tenemos el potencial para hacerlo". Científicos desarrollan nuevos circuitos lógicos de ADN