
Crédito:DeepMind
Los jugadores aplauden, di palabras como Whoo, golpea cuchillos de plástico sobre la mesa y disfruta de los mejores fines de semana con la inteligencia artificial como acto principal, gracias a la IA desatada en los juegos.
REINO UNIDO CON CABLE editor de ciencia, Matt Reynolds, analizó el impacto de DeepMind en los hitos de la IA:"Ha superado a los campeones de Go, superó a los jugadores profesionales de StarCraft y centró su atención en el ajedrez y el shogi ".
Dejemos que los juegos continúen, pero las cosas serias deben brillar seriamente. En breve, Podemos admirar que el uso de la IA con el propósito de realizar un descubrimiento científico se haya vuelto especialmente vivo gracias a la investigación en DeepMind.
Los observadores de tecnología comentaron esta semana sobre trabajos de investigación que muestran las fortalezas de la IA. "A medida que la IA madure como campo (y se quede sin videojuegos para conquistar), probablemente más de sus logros se verán así:mejoras sólidas en importantes dominios de investigación".
Un trabajo de investigación publicado en Naturaleza se convirtió en noticia instantánea como un artículo sobre cómo se puede utilizar la IA para predecir el plegamiento de proteínas. VentureBeat se refirió a desafíos formidables que se acercan cada vez más a las metas.
El artículo es "Mejora de la predicción de la estructura de las proteínas utilizando los potenciales del aprendizaje profundo, "El artículo se publicó en línea el 15 de diciembre en Naturaleza .
El sistema DeepMind se llama AlphaFold. Reynolds tenía una nota interesante sobre AlphaFold y el conjunto de datos:"El campo de plegamiento de proteínas también está bien configurado para entrenar agentes con inteligencia artificial. Tiene un gran conjunto de datos:el Protein Data Bank, un repositorio de la estructura 3-D y la composición genética de 150, 000 proteínas, que se utilizó para entrenar el sistema de predicción de la estructura de proteínas de DeepMind, llamado AlphaFold ".
El documento exploró los logros de DeepMind hasta el momento en el uso de IA para predecir el plegamiento de proteínas. El gran problema del plegamiento de proteínas es que, como Kelsey Piper en Vox remarcó, es un "tema crucial para el desarrollo de nuevos medicamentos".
Tres miembros del equipo de DeepMind, Andrew Senior, John Jumper y Demis Hassabis, publicó una entrada de blog el 15 de enero que explica las estructuras de las proteínas, el desafío del plegamiento de proteínas y lo que querían lograr.
"Como lo demuestra la paradoja de Levinthal, tomaría más tiempo que la edad del universo conocido enumerar al azar todas las configuraciones posibles de una proteína típica antes de alcanzar la verdadera estructura tridimensional; sin embargo, las proteínas mismas se pliegan espontáneamente, en milisegundos. Predecir cómo estas cadenas se plegarán en la intrincada estructura tridimensional de una proteína es lo que se conoce como el 'problema del plegamiento de proteínas', un desafío en el que los científicos han trabajado durante décadas ".
El equipo demostró que podían entrenar una red neuronal para hacer predicciones precisas "de las distancias entre pares de residuos, que transmiten más información sobre la estructura que las predicciones de contacto ".
Hablaron de optimización a través de "un algoritmo de descenso de gradiente simple para generar estructuras sin procedimientos de muestreo complejos".
El gran problema de la negrita de las proteínas es que, dijo Piper, es un "tema crucial para el desarrollo de nuevos medicamentos". ¿Cómo es eso? Y, ¿Por qué son tan importantes las "formas"? "La forma que toman las proteínas predice con qué otras sustancias interactuarán, por lo que comprender el plegamiento de proteínas es crucial para el descubrimiento de fármacos e incluso podría usarse para desarrollar nuevos procesos de fabricación, "Dijo Piper.
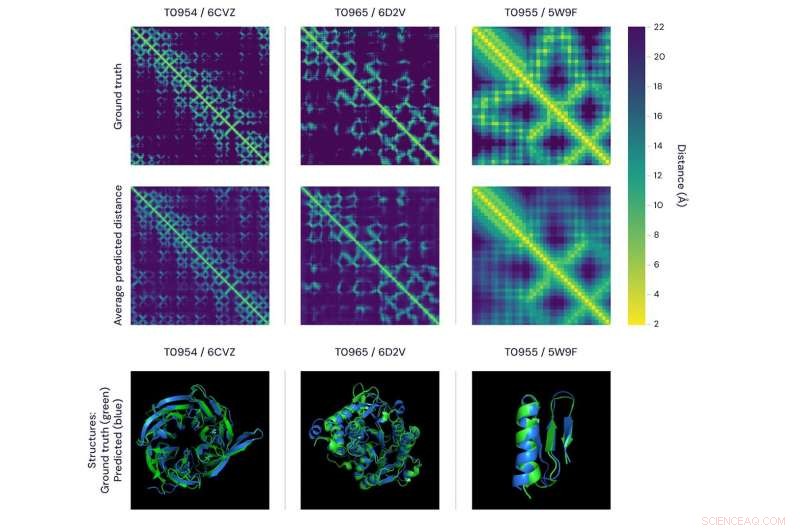
Dos formas de visualizar la precisión de las predicciones de alphafold. la figura superior presenta las matrices de distancia para tres proteínas. El brillo de cada píxel representa la distancia entre los aminoácidos en la secuencia que comprende la proteína:cuanto más brillante es el píxel, cuanto más cerca esté el par. En la fila superior se muestran los reales, distancias determinadas experimentalmente y, en la fila inferior, el promedio de las distribuciones de distancia predichas por AlphaFold. En tono rimbombante, estos encajan bien tanto a escala global como local. los paneles inferiores representan la misma comparación utilizando modelos 3D, con las predicciones de AlphaFold (azul) versus los datos de verdad del terreno (verde) para las mismas tres proteínas. Crédito:DeepMind
El blog DeepMind dijo que "se cree que la forma de una proteína dicta su función. Una vez que se entiende la forma de una proteína, su papel dentro de la célula se puede adivinar, y los científicos pueden desarrollar fármacos que funcionen con la forma única de la proteína ".
AlphaFold podría ayudar a los investigadores a comprender mejor la función y el mal funcionamiento de las proteínas.
Una cita digna de mención en VentureBeat del director del grupo de bioinformática de la UCL, David Jones, quien asesoró al equipo de DeepMind en partes del proyecto:
"Las técnicas experimentales para determinar las estructuras de las proteínas requieren mucho tiempo y son caras, por lo que existe una gran demanda de mejores algoritmos informáticos para calcular las estructuras de las proteínas directamente a partir de las secuencias de genes que las codifican, y el trabajo de DeepMind sobre la aplicación de la IA a este problema de larga data en biología molecular es un avance definitivo ".
¿Qué tipo de técnicas experimentales han consumido mucho tiempo y han sido costosas? El blog mencionó microscopía crioelectrónica, resonancia magnética nuclear y cristalografía de rayos X, cada uno dependiendo de prueba y error, "que puede llevar años de trabajo, y cuestan decenas o cientos de miles de dólares por estructura proteica ".
Kyle Wiggers en VentureBeat :"Con AlphaFold, El equipo de investigación de DeepMind se centró en el problema de modelar formas objetivo desde cero sin utilizar proteínas resueltas como plantillas ".
Cómo define la naturaleza el plegamiento de proteínas:"El plegamiento de proteínas es el proceso mediante el cual las proteínas alcanzan su estructura terciaria funcional madura (nativa), ya menudo comienza co-traduccionalmente. El plegamiento de proteínas requiere acompañantes y, a menudo, implica el establecimiento gradual de estructuras secundarias y supersecundarias regulares. a saber, hélices α y láminas β, que se pliegan rápidamente, estabilizado por puentes de hidrógeno y disulfuro, y luego la estructura terciaria ".
En julio antes de la publicación del artículo de DeepMind, Naturaleza ya había tomado nota de que "la carrera para superar uno de los mayores desafíos de la biología, predecir las estructuras tridimensionales de las proteínas a partir de sus secuencias de aminoácidos, se está intensificando, gracias a los nuevos enfoques de inteligencia artificial (IA) ".
La publicación del blog DeepMind dijo que hicieron que el código estuviera disponible (GitHub), y ofreció el enlace, para cualquiera que desee obtener más información o replicar sus resultados.
En la publicación de su blog, explicaron por qué su sistema es importante:"Nuestro sistema, AlphaFold ... se basa en décadas de investigación previa utilizando grandes conjuntos de datos genómicos para predecir la estructura de las proteínas. Los modelos tridimensionales de proteínas que genera AlphaFold son mucho más precisos que los anteriores ".
Avanzando el blog declaró que una herramienta como AlphaFold podría ayudar a los investigadores de enfermedades raras a predecir la forma de una proteína de interés de manera rápida y económica y, eventualmente, podría ayudarlos a contribuir al descubrimiento eficiente de fármacos. reduciendo al mismo tiempo los costos asociados con la experimentación.
La investigación de enfermedades raras no es la única área que puede beneficiarse de lo que han logrado. El blog hablaba de contaminantes como el plástico y el aceite; Los avances en enzimas biodegradables habilitados por el diseño de proteínas podrían ayudar a descomponer los desechos de maneras más amigables con nuestro medio ambiente.
"De hecho, Los investigadores ya han comenzado a diseñar bacterias para que secreten proteínas que harán que los desechos sean biodegradables. y más fácil de procesar, "según el blog.
© 2020 Science X Network