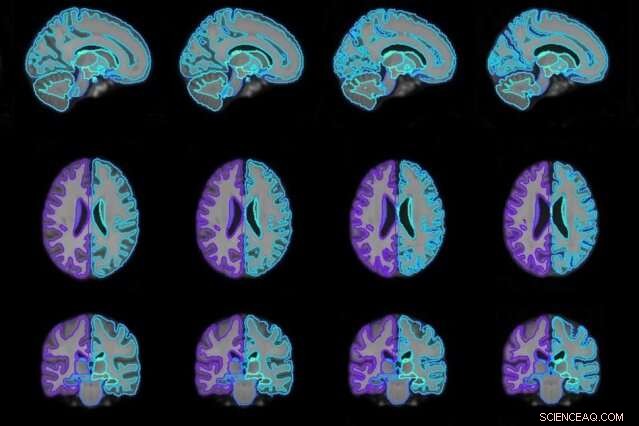
Con su modelo, los investigadores pudieron generar plantillas de escaneo cerebral a pedido de varias edades (en la imagen) que se pueden usar en el análisis de imágenes médicas para guiar el diagnóstico de enfermedades. Crédito:Instituto de Tecnología de Massachusetts
Los investigadores del MIT han ideado un método que acelera el proceso de creación y personalización de plantillas utilizadas en el análisis de imágenes médicas. para orientar el diagnóstico de enfermedades.
Un uso del análisis de imágenes médicas es procesar conjuntos de datos de las imágenes médicas de los pacientes y capturar las relaciones estructurales que pueden indicar la progresión de las enfermedades. En muchos casos, el análisis requiere el uso de una plantilla de imagen común, llamado "atlas, "Esa es una representación promedio de una población de pacientes determinada. Los atlas sirven como referencia para la comparación, por ejemplo, para identificar cambios clínicamente significativos en las estructuras cerebrales a lo largo del tiempo.
Crear una plantilla requiere mucho tiempo, laborioso proceso, a menudo tarda días o semanas en generarse, especialmente cuando se utilizan escáneres cerebrales en 3-D. Para ganar tiempo, Los investigadores a menudo descargan atlas disponibles públicamente previamente generados por grupos de investigación. Pero esos no capturan completamente la diversidad de conjuntos de datos individuales o subpoblaciones específicas, como aquellos con enfermedades nuevas o de niños pequeños. Por último, el atlas no se puede mapear sin problemas en imágenes atípicas, produciendo malos resultados.
En un artículo presentado en la Conferencia sobre sistemas de procesamiento de información neuronal en diciembre, los investigadores describen un modelo de aprendizaje automático automatizado que genera atlas "condicionales" basados en atributos específicos del paciente, como la edad, sexo, y enfermedad. Aprovechando la información compartida de todo un conjunto de datos, el modelo también puede sintetizar atlas de subpoblaciones de pacientes que pueden faltar por completo en el conjunto de datos.
"El mundo necesita más atlas, "dice el primer autor Adrian Dalca, un ex postdoctorado en el Laboratorio de Ciencias de la Computación e Inteligencia Artificial (CSAIL) y ahora miembro de la facultad de radiología en la Escuela de Medicina de Harvard y el Hospital General de Massachusetts. "Los atlas son fundamentales para muchos análisis de imágenes médicas. Este método puede generar muchos más y también generar otros condicionales".
Junto a Dalca en el papel están Marianne Rakic, un investigador visitante en CSAIL; John Guttag, el profesor Dugald C. Jackson de Ciencias de la Computación e Ingeniería Eléctrica y director del Grupo de Inferencia Conducida por Datos de CSAIL; y Mert R. Sabuncu de la Universidad de Cornell.
Alineación y atlas simultáneos
Los métodos tradicionales de construcción de atlas corren mucho tiempo, Procesos de optimización iterativa en todas las imágenes de un conjunto de datos. Se alinean decir, todos los escáneres cerebrales en 3-D a un atlas inicial (a menudo borroso), y calcular una nueva imagen promedio a partir de los escaneos alineados. Repiten este proceso iterativo para todas las imágenes. Esto calcula un atlas final que minimiza la medida en que todos los escaneos en el conjunto de datos deben deformarse para coincidir con el atlas. Hacer este proceso para subpoblaciones de pacientes puede ser complejo e impreciso si no hay suficientes datos disponibles.
La asignación de un atlas a un nuevo escaneo genera un "campo de deformación, "que caracteriza las diferencias entre las dos imágenes. Esto captura variaciones estructurales, que luego se puede analizar más a fondo. En escáneres cerebrales, por ejemplo, las variaciones estructurales pueden deberse a la degeneración del tejido en diferentes etapas de una enfermedad.
En trabajos anteriores, Dalca y otros investigadores desarrollaron una red neuronal para alinear rápidamente estas imágenes. En parte, que ayudó a acelerar el proceso tradicional de construcción de atlas. "Dijimos, "¿Por qué no podemos construir atlas condicionales mientras aprendemos a alinear imágenes al mismo tiempo?", Dice Dalca.
Para hacerlo los investigadores combinaron dos redes neuronales:una red aprende automáticamente un atlas en cada iteración, y otro, adaptado de la investigación anterior, alinea simultáneamente ese atlas con las imágenes de un conjunto de datos.
Entrenando, la red conjunta recibe una imagen aleatoria de un conjunto de datos codificado con los atributos deseados del paciente. A partir de ese, estima un atlas condicional de atributos. La segunda red alinea el atlas estimado con la imagen de entrada, y genera un campo de deformación.
El campo de deformación generado para cada par de imágenes se utiliza para entrenar una "función de pérdida, "un componente de los modelos de aprendizaje automático que ayuda a minimizar las desviaciones de un valor dado. En este caso, la función aprende específicamente a minimizar las distancias entre el atlas aprendido y cada imagen. La red refina continuamente el atlas para alinearlo sin problemas con cualquier imagen dada en el conjunto de datos.
Atlas bajo demanda
El resultado final es una función que aprende cómo atributos específicos, como la edad, se correlacionan con variaciones estructurales en todas las imágenes de un conjunto de datos. Al incorporar nuevos atributos del paciente a la función, Aprovecha toda la información aprendida en el conjunto de datos para sintetizar un atlas bajo demanda, incluso si esos datos de atributos faltan o son escasos en el conjunto de datos.
Digamos que alguien quiere un atlas de escaneo cerebral para una paciente de 45 años de un conjunto de datos con información de pacientes de 30 a 90 años, pero con pocos datos para mujeres de 40 a 50 años. La función analizará patrones de cómo cambia el cerebro entre las edades de 30 a 90 e incorporará los pocos datos que existen para esa edad y sexo. Luego, producirá el atlas más representativo para las mujeres de la edad deseada. En su papel los investigadores verificaron la función generando plantillas condicionales para varios grupos de edad de 15 a 90 años.
Los investigadores esperan que los médicos puedan usar el modelo para construir sus propios atlas rápidamente a partir de los suyos. conjuntos de datos potencialmente pequeños. Dalca ahora colabora con investigadores del Hospital General de Massachusetts, por ejemplo, aprovechar un conjunto de datos de escáneres cerebrales pediátricos para generar atlas condicionales para niños más pequeños, que son difíciles de conseguir.
Un gran sueño es construir una función que pueda generar atlas condicionales para cualquier subpoblación, desde el nacimiento hasta los 90 años. Los investigadores pueden iniciar sesión en una página web, ingrese una edad, sexo, enfermedades y otros parámetros, y obtenga un atlas condicional bajo demanda. "Eso sería maravilloso, porque todos pueden referirse a esta función como una única referencia de atlas universal, "Dice Dalca.
Otra aplicación potencial más allá de las imágenes médicas es el entrenamiento deportivo. Alguien podría entrenar la función para generar un atlas para, decir, movimiento de saque de un jugador de tenis. Luego, el jugador podría comparar nuevos servicios con el atlas para ver exactamente dónde se mantuvieron en forma adecuada o dónde salieron mal las cosas.
"Si miras deportes, Por lo general, los comentaristas dicen que notaron si la forma de alguien estaba fuera de un momento en comparación con otro, "Dice Dalca." Pero puedes imaginar que podría ser mucho más cuantitativo que eso ".
Esta historia se vuelve a publicar por cortesía de MIT News (web.mit.edu/newsoffice/), un sitio popular que cubre noticias sobre la investigación del MIT, innovación y docencia.