Si bien E. coli es uno de los organismos más estudiados, aún no se ha revelado claramente la función del 30% de las proteínas que componen E. coli. Para ello, se utilizó una inteligencia artificial para descubrir 464 tipos de enzimas a partir de proteínas que se desconocían, y los investigadores verificaron las predicciones de tres tipos de proteínas que fueron identificadas con éxito mediante ensayos enzimáticos in vitro.
Un equipo de investigación conjunto, que incluye a Gi Bae Kim, Ji Yeon Kim, el Dr. Jong An Lee y el Distinguido Profesor Sang Yup Lee del Departamento de Ingeniería Química y Biomolecular del KAIST, y el Dr. Charles J. Norsigian y el Profesor Bernhard O. Palsson de El Departamento de Bioingeniería de la UCSD ha desarrollado DeepECtransformer, una inteligencia artificial que puede predecir las funciones enzimáticas a partir de la secuencia de proteínas. Además, el equipo ha establecido un sistema de predicción mediante la utilización de IA para identificar de forma rápida y precisa la función de la enzima.
El trabajo del equipo se describe en el artículo titulado "Anotación funcional de genes codificadores de enzimas mediante aprendizaje profundo con capas transformadoras". El artículo fue publicado el 14 de noviembre en Nature Communications. .
Las enzimas son proteínas que catalizan reacciones biológicas e identificar la función de cada enzima es esencial para comprender las diversas reacciones químicas que existen en los organismos vivos y las características metabólicas de esos organismos.
El número de la Comisión de Enzimas (CE) es un sistema de clasificación de funciones enzimáticas diseñado por la Unión Internacional de Bioquímica y Biología Molecular, y para comprender las características metabólicas de varios organismos, es necesario desarrollar una tecnología que pueda analizar rápidamente las enzimas y los números CE. de las enzimas presentes en el genoma.
Se han desarrollado varias metodologías basadas en el aprendizaje profundo para analizar las características de las secuencias biológicas, incluida la predicción de la función de las proteínas, pero la mayoría de ellas tienen el problema de una caja negra, donde el proceso de inferencia de la IA no se puede interpretar.
También se han informado varios sistemas de predicción que utilizan IA para la predicción de la función enzimática, pero no resuelven este problema de caja negra o no pueden interpretar el proceso de razonamiento a un nivel detallado (por ejemplo, el nivel de residuos de aminoácidos en la secuencia de la enzima). ).
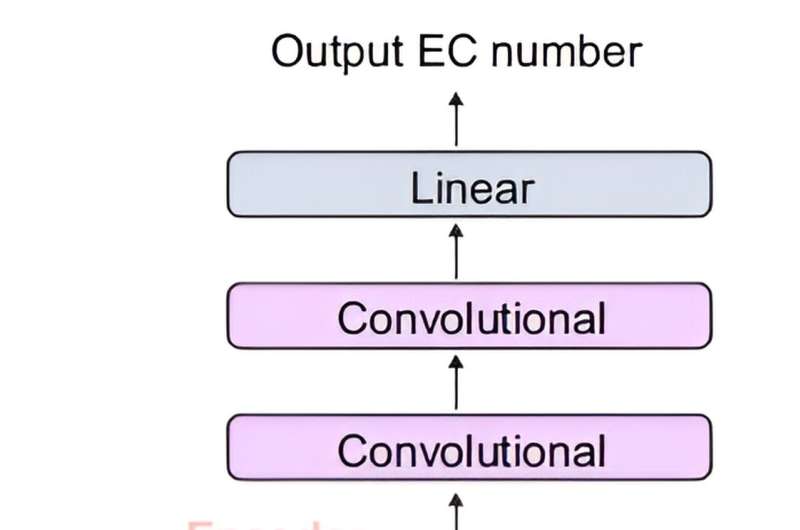
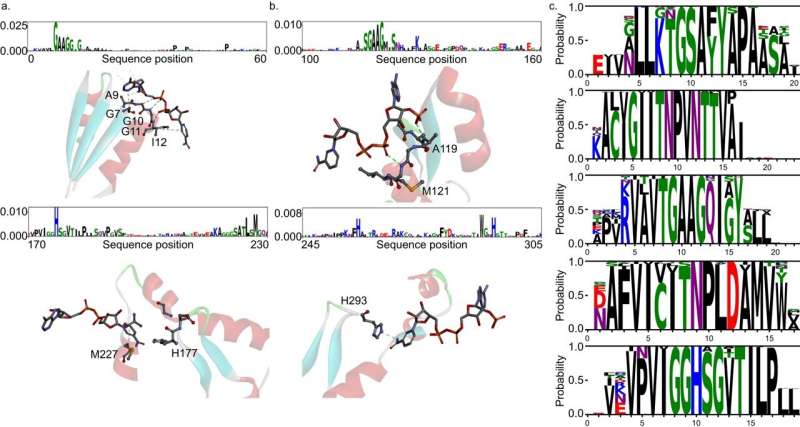
El equipo conjunto desarrolló DeepECtransformer, una IA que utiliza aprendizaje profundo y un módulo de análisis de homología de proteínas para predecir la función enzimática de una secuencia de proteínas determinada.
Para comprender mejor las características de las secuencias de proteínas, se utilizó además la arquitectura transformadora, que se usa comúnmente en el procesamiento del lenguaje natural, para extraer características importantes sobre las funciones de las enzimas en el contexto de toda la secuencia de proteínas, lo que permitió al equipo predecir con precisión la EC. número de la enzima. El transformador DeepEC desarrollado puede predecir un total de 5360 números EC.
El equipo conjunto analizó más a fondo la arquitectura del transformador para comprender el proceso de inferencia de DeepECtransformer y descubrió que en el proceso de inferencia, la IA utiliza información sobre los sitios activos catalíticos y/o los sitios de unión de cofactores que son importantes para la función enzimática. Al analizar la caja negra del transformador DeepEC, se confirmó que la IA fue capaz de identificar por sí sola las características que son importantes para la función enzimática durante el proceso de aprendizaje.
"Al utilizar el sistema de predicción que desarrollamos, pudimos predecir las funciones de enzimas que aún no habían sido identificadas y verificarlas experimentalmente", afirmó Gi Bae Kim, primer autor del artículo.
"Al utilizar DeepECtransformer para identificar enzimas previamente desconocidas en organismos vivos, podremos analizar con mayor precisión varias facetas involucradas en los procesos metabólicos de los organismos, como las enzimas necesarias para biosintetizar varios compuestos útiles o las enzimas necesarias para biodegradar plásticos". añadió.
"DeepECtransformer, que predice de forma rápida y precisa las funciones enzimáticas, es una tecnología clave en genómica funcional, que nos permite analizar la función de enzimas enteras a nivel de sistemas", afirmó el profesor Sang Yup Lee.
Y añadió:"Podremos utilizarlo para desarrollar fábricas microbianas ecológicas basadas en modelos metabólicos integrales a escala del genoma, minimizando potencialmente la información faltante del metabolismo".
Más información: Gi Bae Kim et al, Anotación funcional de genes que codifican enzimas mediante aprendizaje profundo con capas transformadoras, Nature Communications (2023). DOI:10.1038/s41467-023-43216-z
Información de la revista: Comunicaciones sobre la naturaleza
Proporcionado por el Instituto Avanzado de Ciencia y Tecnología de Corea (KAIST)