Un número cada vez mayor de bacterias se ha vuelto resistente a muchos antibióticos de uso común. Investigadores de Bochum han descubierto una nueva oportunidad para una posible molécula activa cuyo predecesor fue rechazado. Al estudiar su interacción con la proteína objetivo bacteriana con mucha precisión en tres dimensiones, identificaron un punto de ataque no detectado previamente al que podría atacar este compuesto.
"Como este punto de ataque sólo se produce en las proteínas bacterianas, el fármaco se volvería mucho más selectivo y se reduciría su nocividad para las células humanas", explica el profesor Raphael Stoll, jefe del grupo de investigación de Espectroscopia de RMN biomolecular de la Facultad de Química y Bioquímica. en la Universidad del Ruhr en Bochum, Alemania.
Los investigadores publicaron sus hallazgos el 4 de abril de 2024 en el Journal of Medicinal Chemistry. .
El estudio se centró en la proteína péptido deformilasa (PDF). Implicado en los procesos de maduración de proteínas en las células, el PDF es esencial para la supervivencia de las bacterias. Sin embargo, se encuentra tanto en bacterias como en células humanas. "Hace unos 20 años se hicieron importantes intentos de combatir la PDF con agentes antibióticos", señala Stoll.
"Sin embargo, el fármaco candidato original, la actinonina, tuvo que ser descartado por varias razones. Uno de los problemas enfrentados fue el PDF humano recientemente descubierto, que estaba potencialmente asociado con efectos secundarios. Aún así, se llevaron a cabo más investigaciones para generar compuestos activos modificados ", añade Hendrik Kirschner.
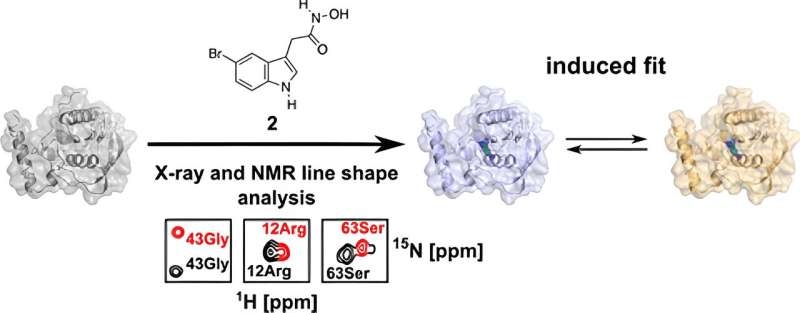
Como parte de su tesis doctoral, examinó el PDF en detalle en el estudio actual y lo analizó desde la perspectiva de la biología estructural utilizando espectroscopia de resonancia magnética nuclear (RMN) biomolecular y cristalografía de rayos X. Estas técnicas le permitieron obtener una estructura 3D resuelta hasta el más mínimo detalle.
"Este es el resultado de muchos años de colaboración con nuestro colega el profesor Eckhard Hofmann", afirma Stoll.
"Podemos utilizar estas técnicas para visualizar superficies y bolsas de unión de biomoléculas y demostrar que la unión de la molécula a esta proteína no es estática, sino dinámica", explica Kirschner.
Los investigadores observaron que hay dos orientaciones de unión diferentes en la proteína para una molécula de fármaco modificada:además de la orientación que también está presente en la PDF humana, hay otra que, en principio, debería ocurrir exclusivamente en la PDF bacteriana.
"Esto nos lleva a modificar la molécula del fármaco para que favorezca la segunda orientación de unión", afirma Stoll.
Como resultado, la molécula del antibiótico podría volverse más selectiva. "Esto podría ofrecer a este fármaco candidato una segunda oportunidad", concluye Kirschner.
Más información: Hendrik Kirschner et al, Hacia inhibidores de antibióticos más selectivos:una visión estructural de la bolsa de unión compleja del péptido deformilasa de E. coli, Journal of Medicinal Chemistry (2024). DOI:10.1021/acs.jmedchem.3c02382
Información de la revista: Revista de Química Medicinal
Proporcionado por Ruhr-Universitaet-Bochum