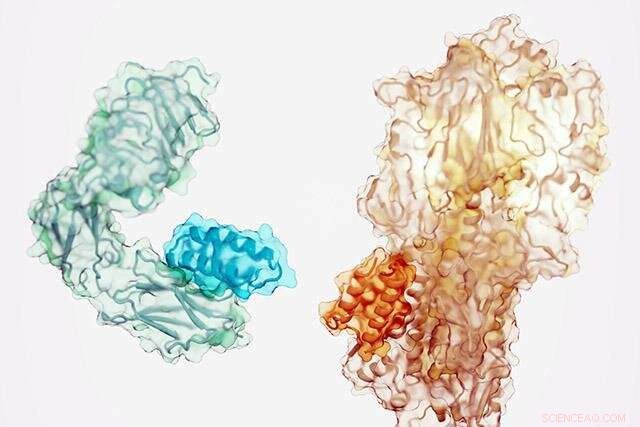
Proteínas pequeñas (sombra más oscura) diseñadas para unirse al receptor de insulina (izquierda) y un componente del virus de la influenza (derecha). Crédito:Ian C. Haydon/Instituto para el Diseño de Proteínas
Un equipo de científicos ha creado un nuevo y poderoso método para generar fármacos proteicos. Usando computadoras, diseñaron moléculas que pueden atacar proteínas importantes en el cuerpo, como el receptor de insulina, así como proteínas vulnerables en la superficie de los virus. Esto resuelve un desafío de larga data en el desarrollo de fármacos y puede conducir a nuevos tratamientos para el cáncer, la diabetes, las infecciones, la inflamación y más.
La investigación, que aparece hoy en la revista Nature , fue dirigido por científicos en el laboratorio de David Baker, profesor de bioquímica en la Facultad de Medicina de la Universidad de Washington y ganador del Premio Breakthrough en Ciencias de la Vida 2021.
"La capacidad de generar nuevas proteínas que se unen fuerte y específicamente a cualquier objetivo molecular que desee es un cambio de paradigma en el desarrollo de fármacos y la biología molecular en general", dijo Baker.
Los anticuerpos son los fármacos basados en proteínas más comunes en la actualidad. Por lo general, funcionan uniéndose a un objetivo molecular específico, que luego se activa o desactiva. Los anticuerpos pueden tratar una amplia gama de trastornos de la salud, incluidos el COVID-19 y el cáncer, pero generar nuevos es un desafío. Los anticuerpos también pueden ser costosos de fabricar.
Un equipo dirigido por dos becarios postdoctorales en el laboratorio de Baker, Longxing Cao y Brian Coventry, combinó avances recientes en el campo del diseño computacional de proteínas para llegar a una estrategia para crear nuevas proteínas que se unen a objetivos moleculares de manera similar a los anticuerpos. Desarrollaron un software que puede escanear una molécula objetivo, identificar sitios de unión potenciales, generar proteínas dirigidas a esos sitios y luego examinar millones de proteínas de unión candidatas para identificar las que tienen más probabilidades de funcionar.
El equipo usó el nuevo software para generar proteínas de unión de alta afinidad contra 12 objetivos moleculares distintos. Estos objetivos incluyen importantes receptores celulares como TrkA, EGFR, Tie2 y el receptor de insulina, así como proteínas en la superficie del virus de la influenza y el SARS-CoV-2 (el virus que causa el COVID-19).
"Cuando se trata de crear nuevos medicamentos, hay objetivos fáciles y objetivos difíciles", dijo Cao, quien ahora es profesor asistente en la Universidad de Westlake. "En este documento, mostramos que incluso los objetivos muy difíciles son susceptibles a este enfoque. Pudimos hacer proteínas de unión a algunos objetivos que no tenían anticuerpos o socios de unión conocidos".
En total, el equipo produjo más de medio millón de proteínas de unión candidatas para las 12 dianas moleculares seleccionadas. Los datos recopilados en este gran conjunto de proteínas de unión candidatas se utilizaron para mejorar el método general.
"Esperamos ver cómo estas moléculas podrían usarse en un contexto clínico y, lo que es más importante, cómo este nuevo método de diseño de fármacos proteicos podría conducir a compuestos aún más prometedores en el futuro", dijo Coventry. Hacia un fármaco para tratar todos los coronavirus