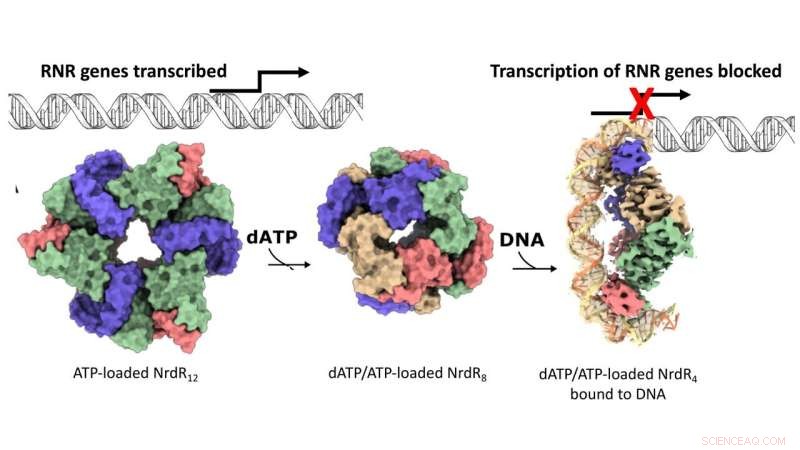
El mecanismo de acción del represor específico de RNR NrdR. Representación superficial de los mapas crio-EM para las estructuras NrdR dodecaméricas, octaméricas y tetraméricas unidas a ADN. Los monómeros NrdR en cada ensamblaje de tetrámero son de color beige, azul, verde y rosa. El ADN se muestra esquemáticamente, excepto por la parte resuelta por crio-EM donde las dos cadenas de ADN antiparalelas son de color naranja y amarillo claro, respectivamente. Crédito:Inna Rozman Grinberg
En un nuevo estudio publicado en Nature Communications , investigadores de la Universidad de Estocolmo muestran por primera vez cómo NrdR se une al ADN para inhibir RNR. El nuevo mecanismo podría ayudar a los científicos a diseñar mejores antibióticos al enfocarse en la capacidad de reproducción de un patógeno.
"Descubrimos NrdR hace más de una década, pero la forma en que funciona era un enigma. En este artículo, combinamos estudios bioquímicos y estructurales para descubrir cómo NrdR se une al ADN", dice Inna Rozman Grinberg, investigadora del Departamento de Bioquímica y Bioquímica. Biofísico de la Universidad de Estocolmo y autor principal del estudio.
La síntesis de ADN es el proceso de creación de nuevas moléculas de ADN que tiene lugar cuando una célula se divide. En prácticamente todos los organismos vivos, la enzima ribonucleótido reductasa (RNR) proporciona elementos básicos para las nuevas cadenas de ADN. En muchas bacterias, incluidos patógenos conocidos como Mycobacterium tuberculosis, la proteína represora NrdR interrumpe la síntesis de RNR. Sin embargo, el mecanismo que sustenta la interacción entre RNR y NrdR seguía siendo esquivo. En general, cuando las células bacterianas se dividen, la RNR se activa para proporcionar componentes básicos para producir nuevo ADN. Mientras tanto, NrdR permanece desconectado. Luego, cuando se completa la síntesis de ADN, NrdR se une al ADN evitando una mayor síntesis de RNR.
Pero, ¿cómo detectan las células cuándo es el momento de apagar RNR? La respuesta radica en la relación de concentración relativa entre dos moléculas clave, a saber, el trifosfato de adenosina (ATP) y el trifosfato de desoxiadenosina (dATP). Los investigadores demostraron que algunos RNR pueden apagarse cuando hay niveles altos de dATP. NrdR, por otro lado, solo se une al ADN e inhibe la síntesis de RNR cuando los niveles de dATP son altos. Este mecanismo de detección, que es utilizado casi exclusivamente por RNR y NrdR, está mediado por un dominio proteico llamado "cono ATP".
"Este mecanismo destaca un aspecto emocionante de la evolución de las proteínas", dice Inna Rozman Grinberg.
Según Inna Rozman Grinberg, dado que NrdR es abundante en bacterias pero está ausente en humanos, el cono de ATP podría usarse como un objetivo antibiótico contra patógenos con un solo RNR, o contra patógenos con NrdR para controlar todos sus RNR. "Hasta donde sabemos, este es el único ejemplo de un dominio de proteína móvil evolutivo que controla tanto la expresión como la actividad de una enzima". El 'cambio' de RNR ofrece esperanza en la lucha contra las bacterias resistentes a los antibióticos