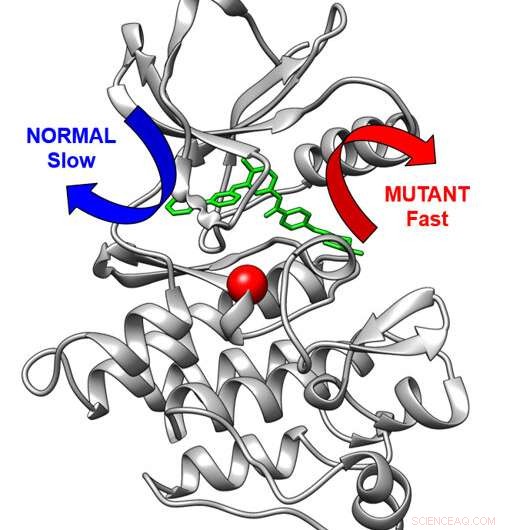
Estructura tridimensional del objetivo del fármaco contra el cáncer Abl quinasa (gris) unida al fármaco contra el cáncer imatinib. Normalmente, el fármaco sale ralentizado por la flecha azul. Una modificación en la quinasa (esfera roja) hace que el fármaco salga por una vía rápida (flecha roja). Crédito:Aziz M. Rangwala
Comprender por qué y cómo se produce la resistencia a la quimioterapia es un paso importante hacia la optimización de los tratamientos contra el cáncer. Un equipo de científicos que incluye a Markus Seeliger, Ph.D., del Stony Brook Cancer Center y Renaissance School of Medicine de la Stony Brook University, cree haber encontrado un nuevo proceso a través del cual ocurre la resistencia a los medicamentos. Están utilizando un modelo de simulación por computadora que los ayuda a comprender exactamente cómo interactúan las moléculas con el medicamento contra el cáncer Imatinib (conocido como Gleevec) en el proceso de resistencia a la quimioterapia. El imatinib trata la leucemia mieloide crónica (LMC) con gran eficacia, pero muchos pacientes en etapa tardía experimentan resistencia al fármaco, lo que hace que el fármaco sea mínimamente efectivo en esa etapa.
La investigación se destaca en un artículo publicado en Angewandte Chemie y se basa en investigaciones anteriores detalladas en 2021 en PNAS .
Imatinib inhibe la proteína quinasa BCR-Abl, una maquinaria de señalización celular demasiado activa en la LMC. En el PNAS estudio, los investigadores demostraron que las variaciones en el plan de construcción de la quinasa pueden dificultar que Imatinib se una a la quinasa y también acelerar la liberación del fármaco de la quinasa. En la Angewandte Chemie artículo, el equipo de investigación tomó la metodología computacional, desarrollada por el coautor Pratyush Tiwary de la Universidad de Maryland, que les permitió estudiar la liberación muy lenta de imatinib de la quinasa.
"Este método en sí mismo es un logro técnico importante que amplía las capacidades computacionales para la investigación de la resistencia a los medicamentos y, lo que es más importante, nos permitió predecir la rapidez con la que las proteínas sanas y mutantes liberarían este fármaco", dice Seeliger, profesor asociado en el Departamento de Farmacología. Ciencias. "Por primera vez, pudimos ver la liberación de un fármaco de una proteína con tanto detalle y precisión. Además, pudimos demostrar que la mutación cambia fundamentalmente dentro de la ruta de salida del fármaco de la proteína.
"Esto es importante ya que la velocidad de liberación del fármaco puede ser tan importante para el efecto terapéutico de un fármaco como la fuerza con que se une un fármaco a la proteína".
Seeliger explica además que el método podría proporcionar una base para comprender los mecanismos moleculares detrás de la resistencia a la quimioterapia.
En términos más generales, las implicaciones de lo que descubrieron son que si los científicos pueden entender cómo se liberan los medicamentos de sus proteínas, pueden diseñar medicamentos con una liberación más lenta y un mayor impacto terapéutico. Además, si la liberación rápida del fármaco puede causar resistencia al fármaco, y los médicos pueden demostrar que esto está sucediendo, es posible que puedan reactivar la eficacia del fármaco pidiéndole al paciente que tome el fármaco con más frecuencia.
El trabajo preliminar para la prueba de mutación a través del método computacional se describió en el PNAS papel. Seeliger y sus colegas probaron cómo imatinib se une a las mutaciones en pacientes con LMC resistente a imatinib. Descubrieron que la mayoría de las mutaciones se unen fácilmente a imatinib, por lo que planteó la pregunta de cómo estas mutaciones causan resistencia en los pacientes. Luego, los investigadores identificaron varios mutantes que se unían fácilmente a imatinib pero liberaban el fármaco mucho más rápido.
Después de identificar estos mutantes con una liberación más rápida del fármaco, el equipo utilizó resonancia magnética nuclear (RMN) y dinámica molecular para vincular la proteína con la disociación del fármaco, lo que subraya la importancia de la cinética de disociación del fármaco para la eficacia del fármaco. Esto les permitió identificar un nuevo mecanismo de resistencia a imatinib.
El trabajo que dio como resultado el artículo publicado en PNAS Involucró los esfuerzos de colaboración de Seeliger y sus colegas en Stony Brook, e investigadores en el Centro de Cáncer Memorial Sloan Kettering y en la Universidad Goethe de Frankfurt, Alemania.
La investigación que dio como resultado el artículo más reciente fue dirigida por Tiwary y sus colegas de la Universidad de Maryland, en colaboración con Seeliger y científicos del Instituto Broad del MIT y la Universidad de Harvard. Nueva simulación por computadora descifra el misterio de la resistencia a los medicamentos contra el cáncer