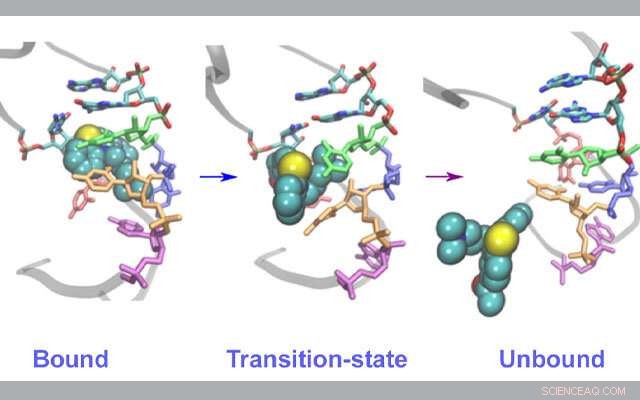
Cambios estructurales en el ARN durante la unión / desvinculación del fármaco. Crédito:Lev Levintov, Universidad de New Hampshire
Investigadores de la Universidad de New Hampshire (UNH) utilizaron recientemente Comet en el Centro de Supercomputación de San Diego en UC San Diego y Stampede2 en el Centro de Computación Avanzada de Texas para identificar nuevas vías de unión / desvinculación de inhibidores en un virus basado en ARN. Los hallazgos podrían ser beneficiosos para comprender cómo reaccionan estos inhibidores y potencialmente ayudar a desarrollar una nueva generación de medicamentos para atacar virus con altas tasas de mortalidad. como el VIH-1, Zika, Ébola, y SARS-CoV2, el virus que causa COVID-19.
"Cuando comenzamos esta investigación, nunca anticipamos que estaríamos en medio de una pandemia causada por un virus de ARN, "dijo Harish Vashisth, profesor asociado de ingeniería química en la UNH. "A medida que surgen estos tipos de virus, Con suerte, nuestros hallazgos ofrecerán una mejor comprensión de cómo los ARN virales interactúan con los inhibidores y se utilizarán para diseñar mejores tratamientos ".
Similar a cómo los humanos codifican su genoma usando ADN, muchos virus tienen una composición genética de moléculas de ARN. Estos genomas basados en ARN contienen sitios potenciales donde los inhibidores pueden adherir y desactivar el virus. Parte del desafío en el desarrollo de fármacos son las variaciones o mutaciones en el genoma viral que pueden evitar que los inhibidores se adhieran.
En su estudio, publicado recientemente en el Revista de letras de química física , Vashisth y su equipo crearon simulaciones de dinámica molecular utilizando las supercomputadoras Comet y Stampede2 para observar específicamente un fragmento de ARN del virus VIH-1 y su interacción con acetilpromazina. una pequeña molécula que se sabe que interfiere con el proceso de replicación del virus.
Los científicos se centraron en los elementos estructurales del genoma del ARN del VIH-1 porque se consideran un buen modelo para estudiar los mismos procesos en una amplia gama de virus de ARN. Estas simulaciones les permitieron descubrir las rutas del inhibidor que se separa del ARN viral en varios eventos raros, que a menudo son difíciles de observar experimentalmente, que inesperadamente mostraron un movimiento coordinado en muchas partes del bolsillo de unión que son los componentes básicos del ARN.
Gracias a las asignaciones de Extreme Science and Engineering Environment (XSEDE) de la National Science Foundation (NSF) en Comet y Stampede2, los investigadores pudieron ejecutar cientos de simulaciones al mismo tiempo para observar lo que se denominan eventos raros de cambio de base involucrados en el proceso de unión / desvinculación del inhibidor que proporcionaron los nuevos detalles del mecanismo subyacente de este proceso.
"Nuestra esperanza es que esto agregue nuevas posibilidades a un campo tradicionalmente enfocado en estructuras biomoleculares estáticas y conduzca a nuevos medicamentos, "Dijo Vashisth.