
Una ilustración muestra una proteína de histocompatibilidad principal (gris) que abarca un péptido extraído de un virus SARS-CoV (rosa). El complejo ayuda a desencadenar la activación de las células T que forman parte del sistema inmunológico. Los investigadores de la Universidad de Rice descubrieron un residuo de unión sin anclaje en el péptido que podría contribuir tanto a la unión como a la activación de las células T necesarias para derrotar al virus. Crédito:Kavraki Lab / Rice University
La "posición 4" no parecía importante hasta que los investigadores examinaron detenidamente un péptido en particular.
Esa parte del péptido extraída de un virus SARS-CoV resultó tener una influencia inesperada pero significativa sobre cómo se une de manera estable con un receptor central para la capacidad del sistema inmunológico de atacar las células enfermas.
En un estudio publicado por el procedimientos de la Academia Nacional de Ciencias , Los investigadores de la Escuela de Ingeniería Brown de la Universidad de Rice y el Centro de Cáncer MD Anderson de la Universidad de Texas revelaron modelos a resolución atómica que detallan no solo la unión sino también, por primera vez, los mecanismos de desvinculación que subyacen a un componente clave del sistema inmunológico.
Dicen que una mejor comprensión de todo el mecanismo podría conducir a avances en la inmunoterapia que aumentan la capacidad del cuerpo para combatir las enfermedades.
La científica informática de Rice Lydia Kavraki, ex alumno Jayvee Abella y el investigador postdoctoral Dinler Antunes, dirigió el estudio.
"Encontrar buenos objetivos para desencadenar una respuesta inmunitaria protectora es muy difícil, especialmente en la investigación del cáncer, "Dijo Antunes." El hecho de que se predijo que este péptido en particular no se uniría a los HLA (antígenos leucocitarios humanos) mediante métodos basados en secuencias destaca un punto ciego en nuestra capacidad de predicción actual.
"Al incorporar el análisis estructural, podemos detectar la contribución de estas interacciones secundarias a la unión y estabilidad de los péptidos, con suerte, nos permitirá encontrar mejores objetivos para el desarrollo de vacunas antivirales y la inmunoterapia contra el cáncer basada en células T, " él dijo.
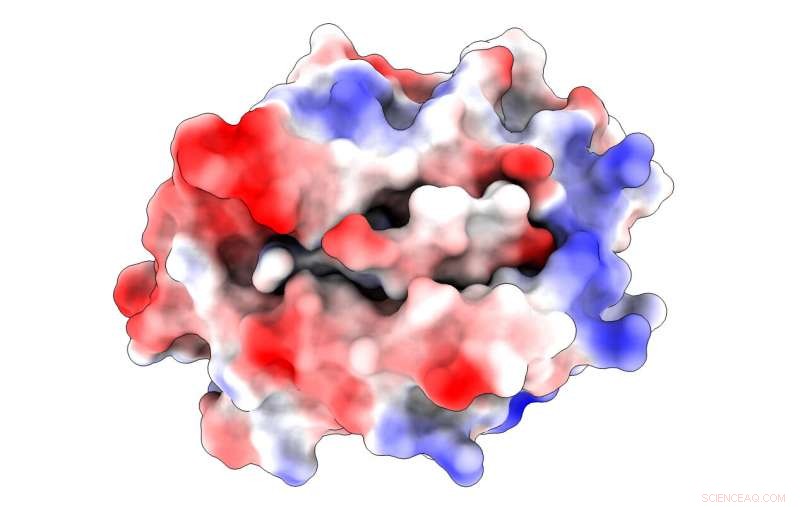
Una ilustración muestra las cargas electrostáticas (el azul es positivo, rojo negativo) en una proteína de histocompatibilidad principal unida a un péptido extraído de un virus del SARS-CoV. El complejo ayuda a desencadenar la infección de las células T que forman parte del sistema inmunológico. Los investigadores de la Universidad de Rice descubrieron un residuo de unión sin anclaje en el péptido que podría contribuir tanto a la unión como a la activación de las células T necesarias para derrotar al virus. Crédito:Kavraki Lab / Rice University
Los investigadores utilizaron sus simulaciones para iluminar detalles de cómo el péptido intracelular del SARS, QFKDNVILL, se une a una proteína receptora del MHC conocida como HLA-A * 24:02, principalmente en los anclajes dominantes en ambos extremos del péptido (en las posiciones 2 y 9) y los presenta para inspección a las células T del sistema inmunológico.
La unión estable de un péptido y MHC es un requisito previo para la activación de las células T, que buscan péptidos que normalmente no se encuentran en células sanas. Si el péptido y la proteína no se unen, no se le pide a la célula T que ataque.
"Eso se sabía de estudios previos sobre los estados ligados y libres de muchos de estos complejos, ", Dijo Kavraki." Lo que no capturaron fueron los estados intermedios y las transiciones que conducen de un estado a otro, especialmente la desvinculación.
"Creo que este es el único análisis que muestra la desvinculación de péptidos del MHC con resolución atómica, ", Dijo Kavraki." Otros péptidos tienen características similares y creemos que tendrían comportamientos similares ".
Todas estas interacciones se revelaron con gran detalle a través de modelos de estado de Markov que analizan cómo cambian los sistemas a lo largo del tiempo. En este caso, los modelos revelaron la importancia de los sitios secundarios que soportan los anclajes primarios del péptido. Ahí es donde se destacó la posición 4.
"Están los principales, anclas canónicas que la gente conoce, pero existen estas interacciones secundarias que contribuyen a la unión y la estabilidad, ", Dijo Antunes." Estos son más difíciles de capturar, pero en este estudio, parece que la posición 4 juega un papel muy importante. Cuando lo mutes, afecta el comportamiento del péptido cuando se desprende de la molécula ".
Los investigadores modelaron mutaciones del MHC para ver cómo influirían en la unión y encontraron que apoyaban la importancia de la posición 4 para la estabilidad del complejo.
Los mapas de potencial electrostático creados a partir de modelos por la Universidad de Rice muestran una proteína de histocompatibilidad importante unida a un péptido extraído de los virus del SARS-CoV. Estos complejos dinámicos pueden desencadenar la activación de las células T que forman parte del sistema inmunológico. El equipo de Rice descubrió la influencia estabilizadora de un residuo de unión alternativo (en la posición 4) común al péptido que estudiaron, hasta arriba, y uno asociado con el SARS-CoV-2, en el fondo, responsable de la enfermedad COVID-19. Crédito:Kavraki Lab / Rice University
"Nuestro enfoque computacional fue capaz de hacer predicciones sobre el efecto de mutaciones que luego se verifican experimentalmente, "dijo la coautora Cecilia Clementi, un ex profesor de Rice que recientemente se convirtió en profesor de Física Einstein en la Universidad Libre de Berlín.
Los investigadores desarrollaron un proceso de dos etapas para simplificar la complejidad computacional del análisis a escala de átomo de moléculas grandes. La primera etapa utilizó una técnica llamada muestreo paraguas para acelerar la exploración inicial de las moléculas. El segundo, etapa exploratoria utilizada muestreo adaptativo, en el que se impulsan simulaciones para acelerar la construcción del modelo de Markov.
"El desafío es que estos MHC son sistemas bastante grandes para que los simulen los químicos computacionales, "dijo Abella, cuya investigación sobre el tema formó gran parte de su tesis doctoral. "Tuvimos que hacer algunas aproximaciones y aprovechar los avances en estas clases de métodos para seguir adelante.
"No somos los primeros en estudiar la desvinculación, pero lo que caracteriza nuestro trabajo sobre otros es que mantenemos la resolución atómica completa en nuestras simulaciones, ", dijo." Otros trabajos utilizan una técnica conocida como cadena de Markov Monte Carlo, mientras que usamos la dinámica molecular, lo que nos permite incorporar tiempo en nuestro cálculo para capturar la cinética ".
Sus métodos se pueden aplicar a otros complejos péptido-MHC con modelos 3D existentes. "Esto era, en algún sentido, un estudio de viabilidad para demostrar que podemos usar la dinámica molecular y construir un modelo de estado de Markov de un sistema de este tamaño, "Dijo Abella.
Los investigadores también señalaron la relevancia del estudio para la lucha actual contra COVID-19, como el péptido del SARS que vieron, QFKDNVILL, es muy similar al péptido NFKDQVILL en el SARS-CoV-2, con los mismos bolsillos de encuadernación en las posiciones 2, 4 y 9.
"Estos resultados sugieren que ambos péptidos pueden unirse a HLA-A * 24:02 y proporcionar objetivos para las respuestas antivirales de las células T, que son de gran interés a la luz de la pandemia actual, "dijo el coautor Gregory Lizée, profesor del Departamento de Oncología Médica del Melanoma del MD Anderson. "Pero estos resultados también arrojan luz sobre muchos otros posibles objetivos inmunitarios, incluidos los de otros virus e incluso cánceres humanos ".
Kavraki señaló que el trabajo experimental de Lizée y Kyle Jackson, colaboradoras a largo plazo, un asistente de investigación graduado en el laboratorio de Lizée que produjo las proteínas mutantes, fueron fundamentales para validar sus simulaciones. El propio laboratorio de Kavraki ganó una subvención de Investigación de Respuesta Rápida de la National Science Foundation (NSF) para ayudar a identificar fragmentos de proteínas virales del SARS-CoV-2 como posibles objetivos para el desarrollo de vacunas.