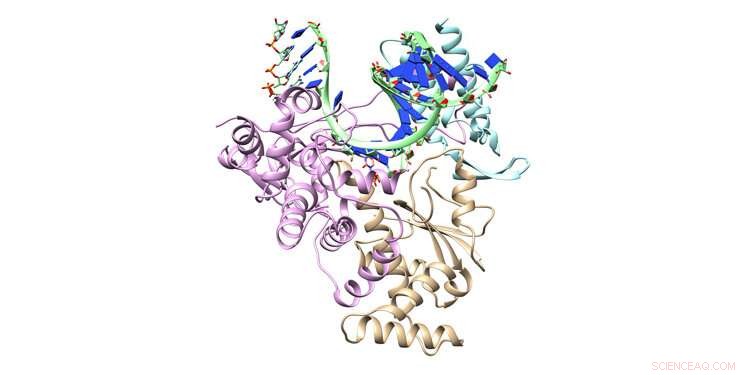
El medicamento antiviral remdesivir forma la línea principal de defensa terapéutica aprobada por la FDA contra el virus COVID-19. Investigadores de la Universidad del Norte de Texas están utilizando la supercomputadora Frontera para modelar cómo remdesivir bloquea la reproducción del coronavirus, con la esperanza de desarrollar mejoras en la droga. Aquí se muestran las estructuras cristalinas del modelo de complejo ternario de ARN polimerasa dependiente de ARN con ARN bicatenario y trifosfato de remdesivir entrante. Crédito:Grupo de Investigación Cisneros, UNT
Las proteínas clave utilizadas por el coronavirus para su reproducción están modeladas en la supercomputadora Frontera, financiada por la NSF, por el grupo de investigación Andrés Cisneros de la Universidad del Norte de Texas. Los objetivos de la investigación incluyen encontrar formas de mejorar el remdesivir terapéutico COVID-19. Asignación de Frontera financiada por NSF otorgada a Cisneros a través del Consorcio de Computación de Alto Rendimiento COVID-19.
En mayo de 2020, la Administración de Drogas y Alimentos de los EE. UU. autorizó el medicamento antiviral remdesivir para el tratamiento de emergencia de COVID-19, una de las cuatro terapias que se encuentran actualmente en este estado. Remdesivir detiene la maquinaria química que usa el coronavirus para copiarse a sí mismo, unión a una enzima que realiza el ensamblaje. Si bien el remdesivir se ha mostrado prometedor para ayudar a los pacientes a recuperarse del COVID-19, los científicos están investigando formas de mejorar su eficacia.
Un equipo de científicos dirigido por G. Andres Cisneros de la Universidad del Norte de Texas está modelando las partes clave del coronavirus que usa para copiarse a sí mismo. Las simulaciones se están realizando en las supercomputadoras Stampede2 y Frontera en el Centro de Computación Avanzada de Texas (TACC).
"Fuimos muy afortunados de recibir una asignación en Frontera para poder trabajar en la investigación del mecanismo de los medicamentos que se dirigen a dos proteínas específicas en COVID-19, "Dijo Cisneros. Su trabajo investiga cómo el remdesivir y otros fármacos disponibles inhiben las proteínas NSP-12 y la proteasa principal, ambas enzimas que el coronavirus necesita para su replicación. "Al observar cómo funcionan estos medicamentos, tal vez esta información se pueda utilizar para mejorarlos ".
La proteína NSP-12 junta los nucleótidos que forman el ARN viral, abreviado como A, U, GRAMO, y C, construir juegos completos de material genético para nuevas copias de coronavirus. NSP-12 es en realidad parte de una estructura más grande llamada ARN polimerasa dependiente de ARN (RDRP) que copia el ARN completo. Remdesivir se une con RDRP, taponando la maquinaria.
"Estamos investigando cómo ocurre este proceso, "dijo Cisneros." Al hacer esto, tal vez haya una manera para que nosotros y otros científicos se nos ocurran ideas sobre si se puede mejorar el remdesivir y cómo hacerlo ".
La otra proteína que Cisneros está estudiando se llama proteasa principal. Separa una poliproteína producida por el SARS-CoV-2 traducida del ARN viral en proteínas funcionales que ponen "carne" en sus huesos virales. Detén la proteasa y detiene la formación del virus. Esto lo convierte en un gran objetivo farmacológico.
Cisneros explicó que usa las matemáticas y la física básicas de las ecuaciones de Newton y la mecánica cuántica para calcular las propiedades de las proteínas. incluyendo todo lo relevante para su funcionamiento, como el ARN y el agua. Un enfoque llamado dinámica molecular clásica utiliza las ecuaciones de Newton para simular cómo las proteínas se mueven e interactúan dinámicamente en el tiempo. "Estamos hablando de sistemas que simulamos que están en los cientos de miles de átomos, "Dijo Cisneros.
También simula las reacciones químicas dentro de las proteínas para investigar cómo los medicamentos detienen la RDRP o la proteasa. Un método híbrido llamado QM / MM (mecánica cuántica / mecánica molecular) ahorra tiempo y dinero de cálculo al centrarse más intensamente en las interacciones en el sitio activo, utilizando la dinámica molecular recta más aproximada para todo lo demás.
El grupo Cisneros desarrolló y mantiene un programa llamado LICHEM que les permite usar el enfoque QM / MM. “Una de las características de LICHEM es que nos permite utilizar enfoques para la parte de mecánica clásica que incluyen una mejor descripción de la física que está sucediendo entre las moléculas en el entorno clásico, específicamente, el potencial de AMOEBA ", dijo Cisneros. AMOEBA es desarrollado por Pengyu Ren de UT Austin; Jay Ponder de la Universidad de Washington; y Jean-Philip Piquemal en la Universidad de la Sorbona en París con contribuciones del grupo Cisneros para líquidos iónicos.
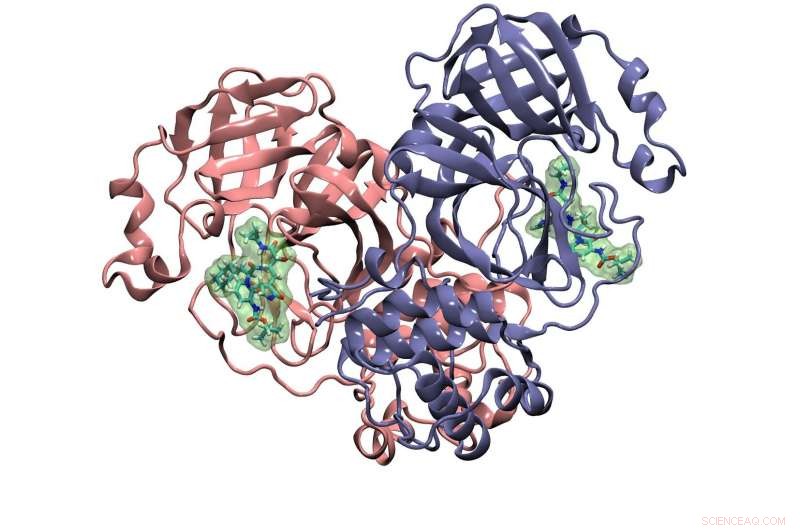
Otro objetivo de investigación del grupo Cisneros que se basa en Frontera es una proteína llamada proteasa principal. Escinde una poliproteína producida por el virus que acumula las proteínas funcionales de las copias de sí mismo que genera. Aquí se muestra la estructura cristalina de la proteasa principal del coronavirus con el inhibidor unido. Crédito:Grupo de Investigación Cisneros, UNT
"Frontera, con no solo la potencia de cálculo sino la intercomunicación entre los nodos, nos permite ejecutar estos cálculos QM / MM con mucho más alto, no solo velocidad, pero también rendimiento, "Dijo Cisneros. Frontera los liberó para ejecutar varios sistemas a la vez". En mi grupo, Tengo cinco científicos diferentes, estudiantes graduados y postdoctorados, que funcionan en ambos sistemas, pero en diferentes piezas del rompecabezas. Todos ellos pueden acceder a estos recursos. Definitivamente es muy útil y agradecemos mucho la asignación ".
Lo que hizo que Cisneros se pusiera en marcha fueron las noticias en abril de 2020 sobre la estructura cristalina del SARS-CoV-2 RDRP que se informó. "Me comuniqué con mi grupo y les dije que con esta información, hay algo que podemos hacer para ayudar con la pandemia, " él dijo.
Dos días después de la noticia, Cisneros propuso con éxito su investigación sobre objetivos de medicamentos contra el coronavirus al Consorcio de Computación de Alto Rendimiento COVID-19. Decenas de instalaciones de supercomputación nacionales e internacionales, industria, y organizaciones, incluida TACC, han ofrecido sus recursos al consorcio en apoyo del esfuerzo de los científicos para combatir el coronavirus.
La asignación se otorgó inicialmente solo en Stampede2 de TACC, el buque insignia de supercomputación de la National Science Foundation (NSF) que ocupa el puesto 21 más rápido del mundo y el número 2 en sistemas académicos según el Top500. "Luego, TACC se puso en contacto con nosotros y agradecimos que se nos concediera acceso a Frontera. Ahora tenemos acceso a ambos sistemas, que es realmente genial, "Dijo Cisneros.
La supercomputadora Frontera es la supercomputadora académica # 1 más rápida y la # 8 más rápida del mundo. Tanto Frontera como Stampede2 son financiados por la NSF.
"Estamos muy contentos con este sistema. Pudimos transferir parte del conocimiento que teníamos de Stampede2 a Frontera, "Dijo Cisneros. Uno de sus estudiantes recién graduados, Erik Vázquez Montelongo, configuró todos los cálculos para LICHEM en Frontera basándose en lo que aprendió en Stampede2. "Eso realmente ha sido una bendición. Frontera para nuestros cálculos ha funcionado muy bien. Estamos muy contentos con él".
Uno de los postdoctorados de The Cisneros Group, Sehr Nazeem-Kahn, generó el modelo para RDRP, el remdesivir y otros fármacos candidatos, todo en el sitio activo. Con eso en la mano comenzaron a ejecutar simulaciones.
"Nos alegró mucho ver que su modelo estaba realmente muy cerca de la estructura experimental. Eso es realmente útil para nosotros, porque valida el modelo que ha sido construido por el grupo y muestra que estamos en el camino correcto, "añadió.
En la actualidad, El Dr. Naseem-Khan está ejecutando simulaciones de dinámica molecular de este modelo con remdesivir en Frontera. "También comenzamos con nuestros cálculos QM / MM para RDRP. En el caso de la proteasa principal, había estructuras que también necesitaban ser modeladas y posteriormente confirmadas. Eso también fue muy satisfactorio, "Dijo Cisneros.
Con esa estructura de datos, están mirando seis moléculas inhibidoras diferentes. "Uno de esos, ya estamos comenzando los cálculos de QM / MM en Frontera, y otro en Stampede2, "Dijo Cisneros. Si todo va bien, espera obtener resultados en los próximos cinco a seis meses. "Estos son cálculos muy costosos, ", agregó." Además, ejecutar el análisis lleva tiempo. Si usáramos solo los recursos en casa, se necesitarían varios años ".