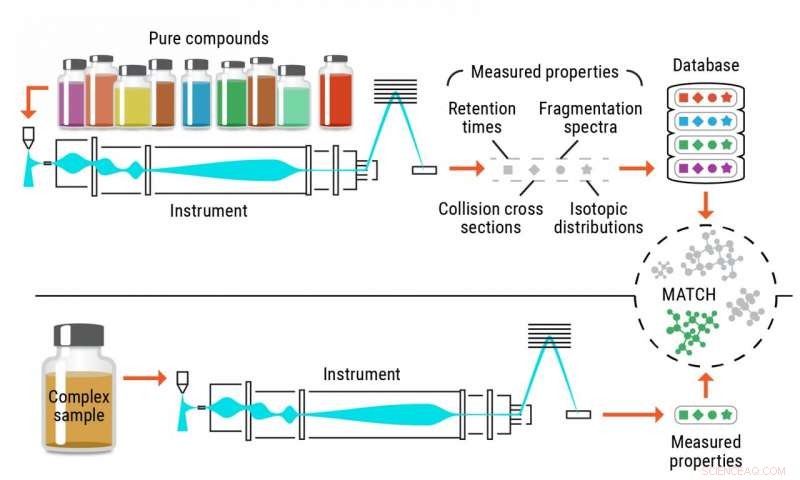
Ilustración del proceso de identificación convencional de metabolitos. Crédito:Laboratorio Nacional del Noroeste del Pacífico
Identificación precisa de metabolitos, y otros productos químicos pequeños, en muestras biológicas y ambientales históricamente se ha quedado corto cuando se utilizan métodos tradicionales. Las tácticas convencionales se basan en compuestos de referencia puros, llamados estándares, reconocer las mismas moléculas en muestras complejas. Estos enfoques están limitados por la disponibilidad de los productos químicos puros que se utilizan como estándares.
"Realmente queríamos pasar por alto el paradigma actual de cómo se realiza un experimento de metabolómica y cómo se identifican con confianza las moléculas, "dijo Tom Metz, científico biomédico del Laboratorio Nacional del Noroeste del Pacífico (PNNL) y director del Núcleo de Identificación de Compuestos Avanzados del Noroeste del Pacífico.
Un problema con el método actual es que hay una cantidad limitada de compuestos puros que los investigadores pueden comprar a los proveedores; la mayoría de los proveedores tienen acceso a alrededor de 3, 000–4, 000 compuestos.
"Si considera lo que se prevé que ocurra en la naturaleza, estás mirando> 1030 compuestos o más que podrían ser posibles, "dijo Metz." Entonces, cuando compara los pocos miles de productos químicos estándar a los que tiene acceso con la gran cantidad de compuestos potenciales, ni siquiera estás cerca ".
Enfoque de identificación sin estándares
Para resolver este problema, Metz y su equipo en PNNL conceptualizaron un enfoque (metabolómica libre de estándares) con el que calculan o predicen información sobre múltiples propiedades para moléculas de interés con el fin de generar bibliotecas de referencia completas y luego relacionar los datos experimentales que contienen las mismas propiedades con estas bibliotecas, permitiendo la identificación compuesta.
Usando este nuevo enfoque, Los investigadores envían estructuras químicas a través de programas de aprendizaje automático o química cuántica para predecir con precisión las propiedades experimentales de los metabolitos.
"Si somos lo suficientemente precisos en estas predicciones, en teoría nunca más necesitaríamos analizar un compuesto puro, ", dijo Metz." Esta colección de herramientas cambiará el paradigma actual en metabolómica, y en un futuro cercano habrá algunas aplicaciones realmente buenas para mostrar a la comunidad de investigadores los beneficios de este nuevo enfoque ".
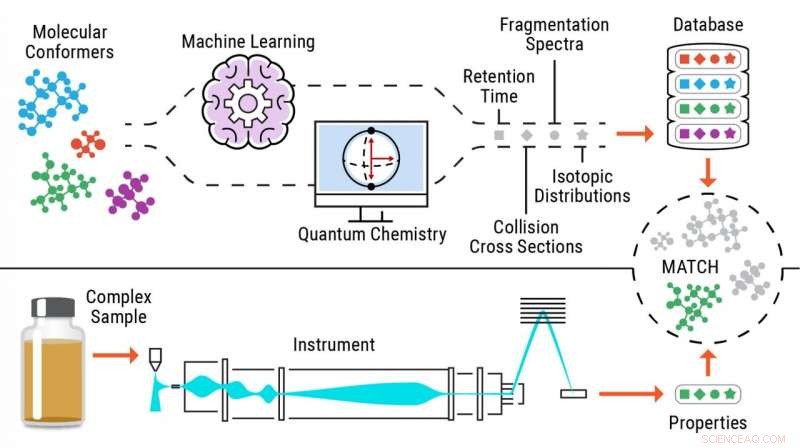
Ilustración del proceso de identificación de metabolitos sin estándares. Crédito:Laboratorio Nacional del Noroeste del Pacífico
Al no tener que depender de datos de análisis de estándares puros para identificar moléculas pequeñas, El enfoque sin estándares permite la identificación de hasta un 90 por ciento más de sustancias químicas en las muestras y hace que estas herramientas computacionales sean muy útiles en varias áreas de aplicación. incluido el descubrimiento de nuevos fármacos, forense químico, e investigación ambiental y biomédica.
"Por ejemplo, en el diseño de un nuevo fármaco, un usuario podría decir:'Tengo un cierto número de propiedades con estos ciertos medicamentos, pero resultan ser tóxicos. ¿Podemos predecir un compuesto que tendría propiedades similares pero que podría no ser tóxico? '", Dijo Metz." Si se pudieran proporcionar los datos de entrenamiento correctos al programa DarkChem, DarkChem podría entonces realizar esa predicción ".
Conjunto de programas personalizables
El nuevo enfoque para la identificación metabolómica libre de estándares utiliza cuatro herramientas clave para generar en bibliotecas de referencia de metabolitos derivados de sílice, y extraer y comparar datos experimentales para obtener identificaciones de compuestos:
Las herramientas han sido diseñadas para trabajar juntas, pero también se pueden utilizar por separado. Los investigadores pueden personalizar las diferentes aplicaciones en función de las necesidades o áreas de investigación de un cliente, creando un enfoque completamente modular.
Avanzando en un campo de investigación
Ahora, en la comunidad metabolómica, todos los investigadores identifican el mismo conjunto de moléculas en cada muestra. La razón de esto es que todos tienen los mismos compuestos puros que compraron para construir sus bibliotecas de referencia.
"Nuestra visión es que al utilizar el enfoque sin estándares, nunca estará limitado por la extensión de moléculas pequeñas que se pueden identificar en una muestra, ", dijo Metz." Eso es realmente un cambio de juego para la metabolómica. Y es muy emocionante ver lo que el próximo año tiene reservado para esto ".