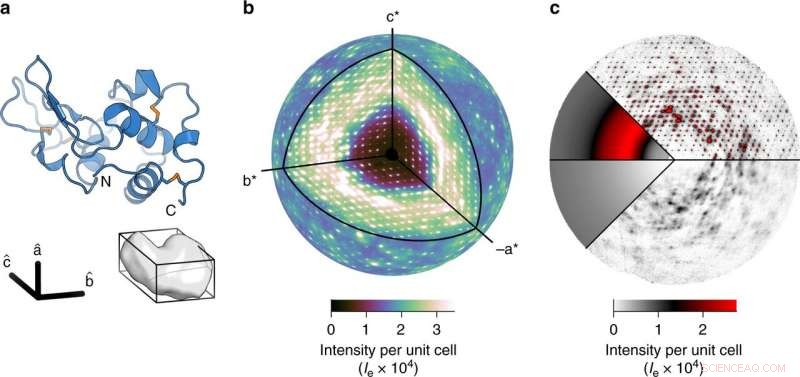
un diagrama de cinta de lisozima (arriba) y la celda unitaria triclínica que contiene una proteína (abajo). b Se obtuvo un mapa tridimensional de dispersión difusa muy detallado. La esfera exterior se dibuja con una resolución de 2 Å. c La dispersión total se compone de tres componentes:dispersión de Compton inelástica (abajo a la izquierda), un anillo isotrópico amplio que domina la señal difusa (arriba a la izquierda), y características variacionales en la dispersión difusa (derecha). Los halos intensos son visibles en las capas que contienen picos de Bragg (plano l =0, superior derecha). La dispersión nublada se visualiza mejor en los planos a medio camino entre los picos de Bragg (plano l =1∕2, inferior derecha). Crédito: Comunicaciones de la naturaleza (2020). DOI:10.1038 / s41467-020-14933-6
Los biólogos estructurales de Cornell adoptaron un nuevo enfoque para utilizar un método clásico de análisis de rayos X para capturar algo que el método convencional nunca había tenido en cuenta:el movimiento colectivo de las proteínas. Y lo hicieron mediante la creación de software para unir minuciosamente los fragmentos de datos que generalmente se ignoran en el proceso.
Su papel "Dispersión difusa de rayos X a partir de movimientos correlacionados en un cristal de proteína, "publicado el 9 de marzo en Comunicaciones de la naturaleza .
Como biólogo estructural, Nozomi Ando, SRA. '04, Doctor. '08, profesor asistente de química y biología química, está interesado en trazar el movimiento de las proteínas, y sus partes internas, para comprender mejor la función de las proteínas. Este tipo de movimiento es bien conocido, pero ha sido difícil de documentar porque la técnica estándar para obtener imágenes de proteínas es la cristalografía de rayos X, que produce instantáneas esencialmente estáticas.
"Debido a que estamos estudiando sistemas biológicos realmente desafiantes, el grupo a menudo también tiene que ser pionero en nuevos métodos estructurales, "dijo el investigador postdoctoral Steve Meisburger, Doctor. '14, el autor principal del artículo. "Una de las preguntas que nos han interesado desde el principio es cómo los movimientos respiratorios sutiles de una proteína dirigen la función bioquímica".
Los investigadores llevaron su proyecto a Cornell High Energy Synchrotron Source (CHESS), donde aprovecharon el detector de matriz de píxeles Pilatus 6M de la instalación, lo que les permitió hacer imágenes de muy alta resolución.
Por este trabajo, como en la cristalografía regular, Se emitieron rayos X a un cristal de muestra. El detector de matriz de píxeles registró la intensidad de los rayos X que fueron difractados por las proteínas del cristal. codificando así la estructura atómica. Cualquier trastorno, es decir, movimiento:el interior del cristal hizo que rebotaran fotones adicionales, creando una señal de fondo muy débil llamada dispersión difusa. Esta información se ha descartado tradicionalmente durante el procesamiento de datos.
"Los fotones van a todas partes, y la señal parece extremadamente débil porque está dispersa, "dijo Ando, el autor principal del artículo. "Por décadas, la gente no podía medirlo con precisión, y no supieron interpretarlo ".
Meisburger creó un software para procesar los aproximadamente 50 millones de puntos de datos únicos, resultando en un mapa tridimensional de alta calidad. Para sorpresa de los investigadores, el mapa reveló que un componente significativo de este patrón de dispersión difusa era en realidad el resultado de la vibración de la red de proteínas. Este movimiento de sacudidas era tan dominante, parecía oscurecer cualquier movimiento dentro de las proteínas, lo que inicialmente fue una decepción para los investigadores.
Pero después de tener en cuenta estas vibraciones de celosía en simulaciones, los investigadores también identificaron los movimientos internos de las proteínas. Estos movimientos incluyeron la apertura y el cierre del sitio activo de la proteína.
"Imagina que el cristal es como una fila de personas que intentan caminar juntas mientras se toman de la mano, pero al mismo tiempo, cada individuo puede estar haciendo algo ligeramente diferente, "Dijo Ando." La señal de todos moviéndose juntos es dominante, así que no pudimos discernir la señal sutil que venía de los individuos. Eso era algo que nunca se había tenido en cuenta ".
Este nuevo enfoque de la dispersión difusa podría ayudar a los investigadores a obtener una imagen más clara de la estructura y dinámica de las proteínas y, por último, una mejor comprensión de las reacciones bioquímicas.
"Realmente queremos impulsar esto en una dirección en la que muchas personas puedan usar la técnica y aprender algo nuevo sobre su proteína, "Meisburger dijo." Una gran cosa acerca de esto es que obtienes dispersión difusa de forma gratuita cada vez que haces un experimento de cristalografía regular. Esta técnica realmente agrega información a lo que normalmente obtendría ".