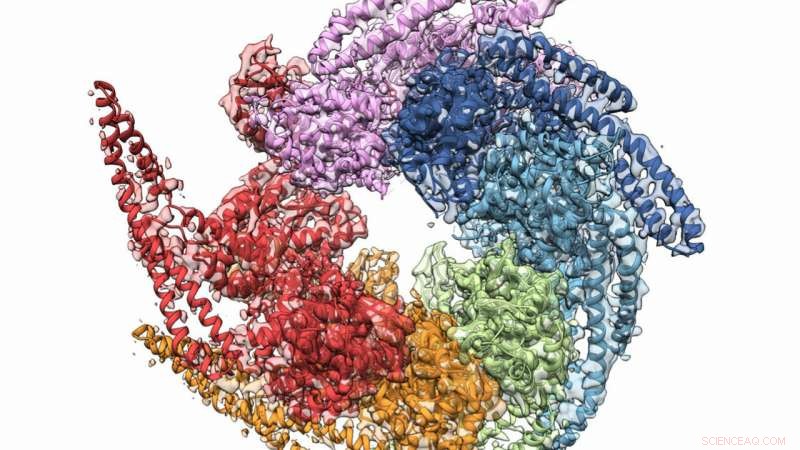
Las estructuras combinadas de microscopía de cristal y crioelectrónica de Hsp104 se disgregan del hongo Calcarisporiella thermophila . Crédito:Grigore Pintilie / Universidad de Stanford
Las técnicas de microscopía electrónica y de rayos X ayudan a desarrollar la historia de las proteínas acompañantes.
Una combinación de cristalografía de rayos X y microscopía crioelectrónica (crio-EM) ayudó en un esfuerzo colaborativo para obtener la estructura de mayor resolución de la proteína fúngica Hsp104, que puede servir para dificultar la formación de determinadas enfermedades degenerativas. El equipo, cuyos miembros incluían investigadores del Laboratorio Nacional Argonne del Departamento de Energía de EE. UU. (DOE), También verificó una estructura helicoidal para los hexámeros formados por proteínas, una vez creído plano. Los resultados se publicaron en línea el 27 de diciembre de 2018 número de Estructura .
Hsp104 es una proteína AAA + hexámera conocida como chaperona, que ayuda en los procesos naturales de plegamiento de las proteínas para el correcto funcionamiento celular. Más importante, quizás, esta clase de hexámeros puede reparar proteínas mal plegadas o agregadas que pueden conducir a anomalías causadas por proteínas como la neurodegeneración.
Las variantes de estos acompañantes son comunes entre los organismos multicelulares, pero Hsp104 no tiene homólogo entre humanos y otros animales. Si bien es más robusto que los acompañantes de origen humano que pueden fallar bajo estrés celular, Se ha observado que Hsp104 plega proteínas humanas.
"Hicimos algunos experimentos en los que expresamos proteínas humanas en levadura, lo que enferma la levadura porque se agregan. Pero si expresa Hsp104, el del hongo Calcarisporiella thermophila , protege a la levadura de esta enfermedad de agregación, "dijo Argonne Distinguished FellowAndrzej Joachimiak.
Joachimiak trabajó con un equipo formado por investigadores de Argonne, la Universidad de Chicago, Stanford University y University of Pennsylvania para comprender mejor la estructura y función de Hsp104.
El equipo recurrió a la fuente de fotones avanzada de Argonne, una instalación para usuarios de la Oficina de Ciencias del DOE, para sondear la estructura de la proteína mediante cristalografía de rayos X. Examinaron 30 muestras diferentes, incluyendo 14 acompañantes Hsp104 de diferentes fuentes. En este proceso, se sumergió un monocristal en nitrógeno líquido y se sometió a rayos X duros de los que se recogieron patrones de difracción y se reconstruyó la estructura. Solo la proteína del Calcarisporiella hongo difractado a alta resolución, señaló Joachimiak.
Para comprender mejor la relación estructural de la proteína dentro de una célula viva, luego pasaron a una investigación de la proteína en solución. Las muestras se examinaron mediante microscopía electrónica en la Universidad de Chicago y luego se enviaron a la Universidad de Stanford, donde microscopios crioelectrónicos recopilaron los datos moleculares de alta resolución necesarios para determinar la estructura a través de una computación compleja. Al igual que con la investigación de los cristales, los Calcarisporiella muestra proporcionó la mejor resolución.
Ahora armado con la resolución más alta de Hsp104 tanto en forma de cristal como en solución, el equipo pudo realizar determinaciones desconocidas o inciertas acerca de las características y comportamientos de la proteína.
Antes del descubrimiento del equipo, biólogos creían que Hsp104 soportaba cinco dominios, secciones independientes de la proteína que cumplen funciones específicas. Pero las nuevas estructuras de alta fidelidad no solo revelaron solo cuatro dominios, pero una imagen más clara de cómo se relacionan entre sí.
El dominio de N terminal, por ejemplo, se cree que se une a una proteína mal plegada y la ayuda a desplegarse en una forma lineal. Esto le permite pasar a través de un canal central estrecho en el hexámero creado por dos dominios AAA (ATPasas asociadas con diversas actividades celulares). La proteína es guiada, o trasladado, a través de este canal al otro lado del hexámero, donde ahora tiene una nueva oportunidad de plegarse correctamente.
"La gente afirmó anteriormente que otro, el dominio del terminal C, fue esencial para la formación del hexámero, pero hemos demostrado que esto no es cierto, "dijo Joachimiak.?" No interactúa con otras subunidades, pero puede funcionar como un área de acoplamiento para otros acompañantes para ayudar a replegar las proteínas ".
Otro hallazgo clave ayudó a cambiar la creencia de que los hexámeros de Hsp104 eran planos. Ambos métodos de investigación demostraron inequívocamente que son helicoidales, ayudando a los investigadores a comprender cómo la chaperona forma el canal de "reparación".
El equipo planea continuar la exploración de los muchos y variados acompañantes que existen fuera del genoma humano. Creen que estos acompañantes pueden servir como una estrategia terapéutica con el potencial de desenredar literalmente proteínas que pueden conducir a enfermedades graves.
"Es posible que haya otros acompañantes que sean mejores que los nuestros, y que podrán tratar proteínas que se agregan en el cerebro y causan enfermedades, "dijo Joachimiak.?" Eso sería realmente fantástico ".
El papel, "Estructura de Calcarisporiella thermophila Hsp104 disgregasa que antagoniza diversos eventos de plegamiento incorrecto proteotóxico, "aparece en la edición en línea del 27 de diciembre de la revista Estructura . Además de Joachimiak, La investigadora de Argonne, Karolina Michalska, contribuyó a este estudio.